UNIVERSITE DJILLALI LIABES FACULTE DES SCIENCES EXACTES
SIDI BEL ABBES
THESE
DE DOCTORAT
Présentée par
:Mr BOUDHEB Tarik
Spécialité : Informatique
Option : Réseaux et sécurité de l’information
Intitulée
« ……… »
Soutenue le 17/07/2019
Devant le jury composé de :
Président :
Dr BOUKLI HACENE Sofiane
UDL-SBA
Examinateurs :
Pr BELALEM Ghalem
Université d’Oran 1
Dr BERRABAH Djamel
UDL-SBA
Directeur de thèse :
Pr. ELBERRICHI Zakaria
UDL-SBA
N° d’ordre
Privacy Preserving Classification
of Biomedical Data
UNIVERSITE DJILLALI LIABES FACULTE DES SCIENCES EXACTES
SIDI BEL ABBES
THESE
DE DOCTORAT
Présentée par
:Mr BOUDHEB Tarik
Spécialité : Informatique
Option : Réseaux et sécurité de l’information
Intitulée
« ……… »
Soutenue le 17/07/2019
Devant le jury composé de :
Président :
Dr BOUKLI HACENE Sofiane
UDL-SBA
Examinateurs :
Pr BELALEM Ghalem
Université d’Oran 1
Dr BERRABAH Djamel
UDL-SBA
Directeur de thèse :
Pr. ELBERRICHI Zakaria
UDL-SBA
Année universitaire : 2018 / 2019
CLASSIFICATION PRESERVANT LA
Résumé : Dans le domaine médical, des organismes de santé, tels que les hôpitaux, les cliniques et
les laboratoires, souhaitent unifier leurs bases de données et collaborer ensemble pour concevoir des modèles Data Mining plus performants. Cependant, cette approche fait face à un sérieux problème qui est la protection de la vie privée des patients. A cause des lois sévères qui condamnent la violation de la vie privée, tel que le RGPD, les parties intéressées par un Data Mining collaboratif sont perplexes. Elles craignent que des informations médicales sensibles, relatives aux patients, tombent entre les mains de personnes mal intentionnées. Ces derniers peuvent porter de graves préjudices aux patients et aux organismes.
A travers cette thèse, nous avons proposé des solutions originales et sécurisées, incluant le respect de la vie privée des patients. Nous avons réalisé la déduplication des enregistrements redondants, la sélection d‘attributs pertinents et la classification supervisée et ce, pour des données biomédicales privées, distribuées géographiquement et appartenant à des tiers organismes de santé. Dans nos solutions, les données biomédicales, originales, ne sont pas perturbées pour garantir la confidentialité. Ainsi, l‘utilité des données originales et les performances initiales des modèles Data Mining sont préservés intactes. Ceci est très important dans le domaine médical, car plus de vies humaines peuvent être sauvées.
Mots clés : Data Mining Collaboratif, Naïve Bayes Distribué, Les Algorithmes Génétiques,
Technique de Hachage, Matrice de Probabilité Perturbée, Sélection d‘attributs, Préservation de la vie privée, Valeurs Aléatoires, Technique Wrapper.
Abstract: In the medical field, health organizations, such as hospitals, clinics and laboratories,
want to unify their databases and work together to design better data mining models. However, this approach faces a serious problem that is the privacy of patients. Because of the harsh laws that condemn the violation of privacy, such as the RGPD, parties interested in collaborative data mining are perplexed. They fear that sensitive medical information about patients will fall into the hands of malicious people. These can cause serious harm to patients and organizations.
Through this thesis, we have proposed original and secure solutions, including the respect of the patients‘ privacy. We performed deduplication of redundant records, selection of relevant attributes, and supervised classification for private biomedical data, geographically distributed and owned by third-party health organizations. In our solutions, the original biomedical data are not disturbed to guarantee confidentiality. Thus, the usefulness of the original data and the initial performance of the Data Mining models are preserved intact. This is very important in the medical field because more lives can be saved.
Key words: Collaborative Data Mining, Distributed Naïve Bayes, Genetic Algorithms, Hashing
Technique, Perturbed Probability Matrix, Attribute Selection, Privacy Preservation, Random Values, Wrapper Technique.
صخلم : ف ً لادًنا ،ًبطنا بغشت ثاًظًُنا ضعب ،تٍحصنا ثاداٍعنأ ثاٍفشتسًنا مثي ،ثاشبتخًنأ آتاَاٍب ذعإق ذٍحٕت ًف ٌأ لاإ .مضفأ جراًَ ىًٍصتن اًعي مًعنأ ٍٍَإقهن ا ًشظَ .ىضشًنا تٍصٕصخ ىهع ظافحنا ًف مثًتت ةشٍطخ تهكشي ّخإٌ حُٓنا ازْ مثي ،تٍصٕصخنا كآتَا ٌٍذت ًتنا تيساصنا RGPD فاشطلأا ٌإف ، بٍقُتب تًتًٓنا عقت ٌأ ٌٕشخٌ ىَٓإ .ةشٍح ًف ثاَاٍبنا تٍبطنا ثايٕهعًنا ىضشًهن تساسحنا .ثاًظًُنأ ىضشًهن اسشض إببسٌ ٌأ ىُٓكًٌ ساششأ صاخشأ يذٌأ ًف .ىضشًنا تٍصٕصخ وشتحت ،تُيآٔ تٍهصأ ًلإهح اُحشتقا ،تحٔشطلأا ِزْ للاخ ٍي ذقهف ،ةسشكًنا ثاَاٍبنا ءاغنإب اًُق ساٍتخا ثايٕهعًنا ًُنا ،تبسا تٌٍٕحنا تٍبطنا ثاَاٍبهن ّخًٕنا فٍُصتنأ ،تصاخنا تٍحصنا ثاًظُي مبق ٍي تكٕهًئ اٍفاشغخ تعصًٕنا ًف .تفهتخي ،اُنٕهح .تٌشسنا ٌاًضن شٍغت لا تٍهصلأا تٌٍٕحنا تٍبطنا ثاَاٍبنا ،ًناتنابٔ ءادلأأ تٍهصلأا ثايٕهعًنا ىهع اُظفاح ي ازْ .ثاَاٍبنا جاشختسا جراًُن ًنٔلأا .ذأسلأا ٍي ذٌضًنا راقَإ ٍكًٌ َّلأ ًبطنا لادًنا ًف تٌاغهن ىٓ :ثحبلا تاملك ،تٍصٕصخنا ىهع ظافحنا ،تٍَٔاعتنا ثاَاٍبنا جاشختسا تٍئإشعنا داذعلأا تلاامتحلاا ةفوفصم ، ةريغملا ، رايتخا تانايبلا ، ةينيجلا تايمزراوخلا ، بٍقُت ثاَاٍبنا .
Je remercie, en premier lieu, Allah, le tout puissant, de m’avoir permis et accorder la volonté, la
patience et le courage pour réaliser ce travail.
Je présente mon immense gratitude et mes remerciements, les plus sincères, à monsieur ELBERRICHI Zakaria, Directeur de la présente thèse, Professeur à l’université Djillali Liabbes, Sidi bel Abbes, de m’avoir fait confiance et accepter dans son équipe. Sa disponibilité, ses orientations pertinentes et avisées, sa patience, et surtout ces qualités humaines ont constitué un apport considérable, sans lequel, ce travail n’aurait pas vu le jour.
Mon profond respect à Mr BOUKLI HACEN Sofiane, Maître de conférences à l’université Djillali Liabes , Sidi Bel Abbes, de nous avoir fait l’honneur d’accepter d’être le président de jury. Mes sincères remerciements et respects à Mr BELALEM Ghalem, Professeur à l’université Ahmed Ben Bella Oran
1
, et Mr BERRABAH Djamel, Maître de conférence à l’université Djillali Liabes de Sidi Bel Abbes, de m’avoir fait honneur en acceptant d’être membre de jury. Leurs pertinentes remarques et suggestions permettrons surement d’enrichir et d’améliorer notre travail.Je tiens à exprimer mes sincères remerciements à tous les professeurs qui m’ont enseigné et qui par leurs compétences et sérieux m’ont permis de poursuivre mes études.
Mes remerciements s’adressent aussi à toutes les personnes qui m’ont soutenu, de prêt ou de loin, durant la réalisation de cette thèse.
Dédicace
A mes chers parents, A ma chère femme, A mes enfants Chakib et Chanez, A ma sœur et à mes frères, A mes beaux-parents.
Introduction Générale
1 Introduction ... 16
2 Problématique ... 18
3 Contributions ... 20
4 Organisation de la thèse ... 22
Chapitre 1: Classification des données biomédicales
1.1. Digitalisation du domaine médical ... 251.1.1. Introduction ... 25
1.1.2. Dossier patient (dossier médical) ... 26
1.1.2.1. Définition ... 26
1.1.2.2. Objectif du dossier patient ... 27
1.1.2.3. Rôle des acteurs intervenant dans le dossier médical de patient ... 28
1.2. Data Mining ... 29
1.2.1. Définition du Data Mining (fouille des données) ... 29
1.2.2. Processus Data Mining ... 29
1.2.2.1. Prétraitement des données ... 30
1.2.2.2. Types d‘apprentissage ... 31
1.2.2.2.1. Apprentissage supervisé ... 31
1.2.2.2.2. Apprentissage non supervisé ... 31
1.2.2.2.3. Apprentissage semi-supervisé ... 31
1.2.2.3. Les méthodes du processus Data Mining ... 32
1.2.3. Classification supervisée ... 32
1.2.4. La matrice de confusion ... 33
1.2.5. Algorithmes de la classification supervisée ... 34
1.2.5.2. Support Vector Machine (SVM) ... 35
1.2.5.3. Arbres de décision ... 36
1.2.5.4. Réseaux de neurones ... 37
1.2.5.5. K plus proches voisins (KNN) ... 37
1.3. Data Mining biomédical ... 38
1.3.1. Définitions ... 38
1.3.1.1. Définition du biomédical ... 38
1.3.1.2. Définition du Data Mining biomédical ... 39
1.3.2. Travaux de recherches ... 39
1.3.3. Projets connus incluant le Data Mining dans le domaine médical ... 42
1.4. Classification supervisée dans le domaine biomédical ... 44
1.5. Conclusion ... 49
Chapitre 2: Classification sécurisée des données biomédicales 2.1. La préservation de la vie privée ... 52
2.1.1. Introduction ... 52
2.1.2. Vie privée dans le domaine médical ... 53
2.1.3. Définition d‘une donnée personnelle ... 54
2.1.4. La protection de la vie privée en Algérie ... 54
2.1.5. Le Data Mining et la préservation de la vie privée des patients ... 55
2.2. La distribution des données ... 56
2.2.1. Données centralisées... 56
2.2.2. Données distribuées ... 56
2.2.2.1. Distribution horizontale ... 57
2.2.2.2. Distribution verticale ... 57
2.3. Les différentes techniques pour la protection de la vie privée ... 58
2.3.1.3. Les méthodes t-proximity ... 62
2.3.2. Les méthodes cryptographiques ... 62
2.3.2.1. Calcul multipartite sécurisé ... 62
2.3.2.2. Techniques de Hachage ... 63
2.3.2.3. Les cryptosystèmes homomorphes ... 64
2.3.2.3.1. Le cryptosystème homomorphe multiplicatif RSA ... 65
2.3.2.3.2. Le cryptosystème homomorphe multiplicatif El Gamal... 66
2.3.2.3.3. Le crypto système homomorphe additif de Paillier ... 67
2.4. Travaux sur la Privacy Preserving Data Mining (PPDM) ... 68
2.5. Conclusion ... 77
Chapitre 3: Déduplication sécurisée de données biomédicales horizontalement distribuées 3.1. Introduction ... 79
3.2. Etat de l‘art ... 80
3.3. La solution proposée ... 84
3.3.1. Partie 1 : Déduplication sécurisée des données biomédicales horizontalement distribuées ... 84
3.3.2. Partie 2 : Classification sécurisée des données biomédicales horizontalement distribuées en utilisant Naïve Bayes dans un environnement semi honnête ... 96
3.4. Analyse de la sécurité ... 103
3.5. Expérimentations ... 104
3.6. Conclusion ... 109
Chapitre 4 : Sélection sécurisée d’attributs pertinents sur des bases de données distribuées 4.1. Introduction ... 112
4.2. Etat de l‘art des méthodes pour la sélection d‘attributs incluant la préservation de la vie privée ... 116
4.3. Approche de sélection d‘attributs sécurisée pour les données biomédicales
réparties horizontalement ... 118
4.3.1. Etape 1 : Calcul de la matrice de probabilités locales ... 120
4.3.1.1. Naïve Bayes sécurisé pour les données horizontalement distribuées ... 120
4.3.1.2. Matrice de probabilité locale (LPM) ... 122
4.3.1.3. Matrice Local de Probabilité Perturbée (PLPM) ... 123
4.3.1.4. Encodage de l‘attribut classe (ECA) ... 124
4.3.2. Etape 2 : Les algorithmes génétiques pour la sélection d‘attributs au niveau du Cloud ... 125
4.3.2.1. Population initiale ... 125
4.3.2.2. La fonction fitness ... 127
4.3.2.2.1. Matrice de Probabilité Globale (GPM) ... 128
4.3.2.2.2. Matrice Finale de Probabilité (FPM) ... 129
4.3.2.2.3. Matrice de classification finale (FCM) ... 130
4.3.2.2.4. Calcul du F-score (FS) ... 130
4.3.2.3. Sélection de chromosomes (CS) ... 131
4.3.2.4. Croisement ... 132
4.3.2.5. Mutation ... 132
4.4. Approche de sélection d‘attributs sécurisée pour les données biomédicales réparties verticalement ... 132
4.4.1. Synchronisation des données entre les sites collaboratifs pour les données verticalement distribuées ... 134
4.4.1.1. Distribution des attributs ... 134
4.4.1.2. Sélection des patients pour les données distribuées verticalement ... 134
4.4.2. Etape 1 : Calcul de la matrice de probabilité locale ... 135
4.4.2.1. Naïve Bayes sécurisé pour les données verticalement distribuées ... 136 4.4.3. Etape 2 : Les algorithmes génétiques pour la sélection d‘attributs pertinents au
4.4.3.2. Encodage de l‘attribut classe (ECA) ... 138
4.5. Expérimentations ... 138
4.6. Conclusion ... 147
Chapitre 5 : Classification sécurisée des données biomédicales en utilisant des sous-modèles privés 5.1. Introduction ... 150
5.2. Définition d‘un modèle Data Mining Distribué ... 151
5.3. Approche proposée ... 152
5.3.1. Construction du modèle Data Mining ... 152
5.3.2. Distribution des attributs ... 153
5.3.3. Classification sécurisée des instances ... 153
5.3.3.1. Sélection sécurisée des attributs selon l‘instance à classer... 153
5.3.3.2. Calcul distribué des probabilités... 156
5.3.3.3. Calcul de la classe finale ... 157
5.4. Expérimentation ... 162
5.5. Conclusion ... 165
6. Conclusion générale et persfectives... 167
Liste de figures
Figure 1.1. Le processus KDD (Tomar & Agarwal, 2013) ... 30
Figure 1.2. SVM classification binaire ... 36
Figure 2.1. Données centralisées ... 56
Figure 2.2. Distribution horizontale des données ... 57
Figure 2.3. Distribution verticale des données ... 57
Figure 2.4. Ré-identification des données anonymes par combinaison de deux tables (Samarati & Sweeney, 1998) ... 58
Figure 2.5. Exemple de perturbation 3-anonymat (Nguyen, 2014) ... 59
Figure 2.6. Perturbation des données 3-diversity (Nguyen, 2014) ... 61
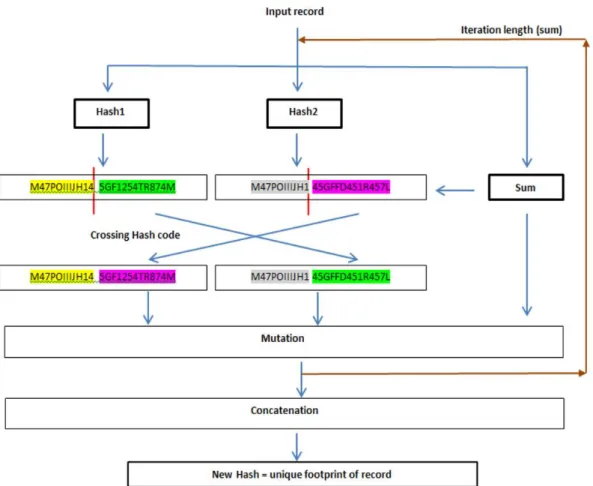
Figure 3.1. Principe du protocole de déduplication ... 85
Figure 3.2. Déduplication sécurisée des enregistrements distribués horizontalement ... 85
Figure 3.3. La solution proposée pour renforcer les codes haches ... 90
Figure 3.4. Déduplication sécurisée en utilisant une tierce partie ... 94
Figure 3.5. Le calcul collaboratif circulaire ... 96
Figure 3.6. Les attaques bloquées par la solution proposée ... 97
Figure 3.7. Classification sécurisée des données biomédicales horizontalement distribuées dans un environnement semi honnête ... 98
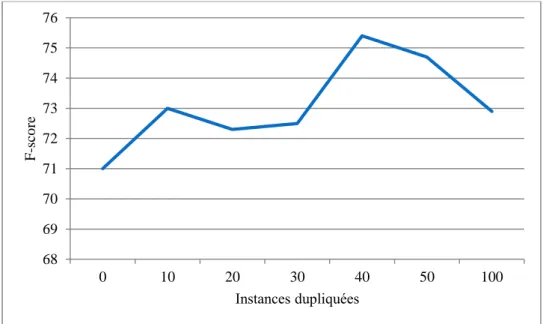
Figure 3.8. Impact des instances dupliquées -Puma Indian Dataset- ... 106
Figure 3.9. Impact des instances dupliquées –Parkinson dataset- ... 106
Figure 3.10. Impact des instances dupliquées -Vertebral 2C Dataset- ... 107
Figure 3.11. Impact des instances dupliquées - Hepatitis Dataset- ... 107
Figure 3.12. Temps d‘exécution pour effectuer une déduplication sécurisée ... 109
Figure 4.1. La technique wrapper ... 112
Figure 4.2. Solution proposée pour la sélection d‘attributs pour des données horizontalement distribuées ... 119
Figure 4.3. Naïve Bayes distribué pour un partitionnement horizontal... 122
Figure 4.4. Structure de la matrice de probabilité locale ... 123
Figure 4.5. PLPM, exemple de perturbation de la LPM du sitei ... 124
Figure 4.6. Processus des Algorithmes Génétiques ... 126
Figure 4.7. Chromosome, la représentation binaire des attributs ... 126
Figure 4.11. GPM et GEC créés pour les données horizontalement distribuées ... 129
Figure 4.12. FPM, La Matrice des Probabilités Finales pour classi ... 130
Figure 4.13. La Matrice de classification Finale FCM ... 131
Figure 4.14. La liste F-score ... 131
Figure 4.15. Solution proposée pour la sélection d‘attributs pertinents ... 133
Figure 4.16. Flux de données pour calculer la GPM pour des données verticalement distribuées ... 137
Figure 4.17. GPM pour la classe ‗i‘ ... 138
Figure 4.18. Codification de l'attribut classe ... 139
Figure 4.19. Holdout répété pour les horizontalement distribuées ... 139
Figure 4.20. Holdout répété pour les données verticalement distribuées ... 140
Figure 4.21. Les attributs pertinents sélectionnés par la solution proposée ... 143
Figure 4.22. Amélioration du F-score par l'approche proposée ... 143
Figure 4.23. Confidentialité vs performances dans la solution proposée ... 145
Figure 4.24. Validation des attributs sélectionnés dans Weka ... 147
Figure 5.1. Modèle distribué... 152
Figure 5.2. Conception de N modèles collaboratifs avec des données privées distribuées verticalement ... 153
Figure 5.3. Schéma global de l‘approche proposée ... 154
Figure 5.4. Vue détaillée du protocole de classification ‗PPCNBVPD‘ ... 154
Figure 5.5. Distribution verticale des valeurs des instances à classer aux sites collaboratifs distants ... 155
Figure 5.6. Temps d'exécution pour créer le modèle - Breast cancer Dataset- ... 164
Figure 5.7. Temps d'exécution pour créer le modèle - Vertebral column Dataset- ... 164
Figure 5.8. Temps classification en utilisant 02 sites - Breast Cancer dataset - ... 165
Liste des tableaux
Table 1.1. Matrice de confusion pour une classification supervisée binaire ... 33
Table 2.1. Exemple de généralisation des données ... 60
Table 2.2. Agrégation des données ... 60
Table 2. 3. Exemple de masquage de valeurs ... 60
Table 2.4. Résumé des approches étudiées ... 74
Table 3.1. Récap des approches de l‘état de l‘art pour la déduplication sécurisée ... 83
Table 3.2. Exemples de Patient (Dataset Breast Cancer) ... 88
Table 3.3. Somme linéaire ... 88
Table 3.4. Concaténation des instances ... 88
Table 3.5. Hachage des instances ... 89
Table 3.6. Taches parallélisées ... 104
Table 3.7. Impact des instances dupliquées sur le F-score ... 105
Table 3.8. Déduplication des enregistrements (deux techniques de hachage sont utilisées: SHA256 et SHA512) ... 108
Table 3.9. Classification sécurisée en utilisant Naïve Bayes ... 108
Table 3.10. Comparaison entre la solution propose avec Weka ... 109
Table 4.1. Datasets utilisés ... 141
Table 4.2. Sélection d‘attributs basée sur l‘utilisation des matrices PLPM ... 142
Table 4.3. Comparaison de notre solution avec la solution de (Jena and al., 2014)... 144
Table 4.4. Confidentialité vs performances dans la solution proposée ... 145
Table 4.5. Validation des attributs sélectionnés dans Weka... 146
Table 5.1. Résultats de l‘expérimentation – Dataset Breast Cancer Winsconsin- ... 163
AD Attribute Distribution
CS Chromosomes Selection
DM Data Mining
DNB Distributed Naïve Bayes
ECA Encoded Class Attribute
ECD Extraction des Connaissances à partir de Données
EPPCNBHD Enhanced Privacy Preserving Classification based on Naive Bayes for Horizontally distributed data
FCM Final Classification Matrix
FPM Final Probability Matrix
FS F-Score list
GA Genetic Algorithm
GPM Global Probabily Matrix
HIPAA Health Insurance Portability and Accountability Act
KDD Knowledge Data Discovery
LPM Local Probability Matrix
NB Naïve Bayes
PLPM Perturbed Local Probability Matrix
PPCNBVPD Privacy Preserving Classification based on Naïve Bayes for Vertically
Partitioned Data
RGPD Règlement Général de la Protection des Données
Sitei Site number i
UCI University of California, Irvine
I
NTRODUCTION
G
ENERALE
Sommaire 1 Introduction ... 16 2 Problématique ... 18 3 Contributions ... 20 4 Organisation de la thèse ... 221 Introduction
Le Data Mining est un outil puissant, car il permet d‘analyser de grande masse de données pour extraire de précieuses connaissances, non évidentes, qui servirons à développer des outils d‘aides à la décision, à comprendre et à prévoir des comportements.
A l‘instar des autres domaines, le Data Mining est devenu un outil indispensable dans le domaine de la santé, voir essentiel (Koh & Tan, 2011). Il permet de mieux comprendre les données médicales, en distinguant les données pathologiques des données normales, et d‘identifier les relations complexes entre elles (Acharya & Yu, 2010). Grace au Data Mining, les soins médicaux se sont nettement améliorés, les maladies sont mieux diagnostiquées et la communication entre le docteur et le patient est personnalisée, donc plus efficace. D‘après
(Eliason & Crockett, 2017), les coûts des soins médicaux peuvent être réduits jusqu‘à 30%. Afin de concevoir des modèles Data Mining plus performants, certaines institutions et organismes dans le domaine de la santé ou autres (tels que les ministères, les hôpitaux, les cliniques, les assurances, les laboratoires et les entreprises) souhaitent unifier leurs efforts, à travers le partage et la consolidation de leurs bases de données. Le croisement transversal de ces données hétérogènes offre de nombreuses opportunités, car il permet d‘élargir le domaine d‘exploration (Vaidya, Shafiq, Fan, Mehmood & Lorenzi, 2014). Ainsi, de nouveaux services efficaces, de qualités, mieux adaptées, seront mis au point pour discerner les causes et les remèdes des maladies. A titre d‘exemple, pour étudier les causes des maladies cancéreuses, en plus des données médicales des patients, d‘autres données externes au domaine de la santé peuvent être utilisées, telles que les données sur le régime alimentaire des patients (consommation et habitudes alimentaires), les données environnementaux (proximité des lignes à haute tension, la circulation, la pollution, le changement climatique, la densité pollinique, les données de température et humidité, l‘utilisation de quelles types des pesticides), etc.
Cependant, cette approche fait face à un sérieux problème qui est la violation de la vie privée des patients. A cause des lois juridiques nationales et internationales sévères interdisant la violation de la vie privée, l‘exploitation des données médicales sensibles est devenue plus que jamais critiques. Sans des garanties sérieuses de sécurité, les parties intéressées par un processus Data Mining collaboratif sont hésitantes. Elles ont peur que les données sensibles de leurs patients tombent entre les mains de personnes ou des organismes mal intentionnées
Introduction Générale
qui peuvent les utiliser à d‘autres fins. En conséquence, les patients peuvent subir de graves préjudices. Face à cette contrainte juridique bloquante et incontournable pour le Data Mining collaboratif, il fallait mettre au point de nouvelles solutions pour être conforme vis-à-vis de la loi en vigueur, sinon de lourdes amendes, allant jusqu‘à 4% du chiffre d‘affaire annuel, seront infligées au contrevenant selon la loi RGPD1(Sylvain, 2018).
L‘axe de recherche ‗Data Mining incluant le respect de la vie privée‘, en anglais Privacy Preserving Data Mining (PPDM), apporte des solutions concrètes à ce problème. Il permet de conduire un processus Data Mining sans avoir accès aux données sensibles, tout en préservant une balance équilibrée entre l‘utilité des données et les performances initiales2 des modèles Data Mining. De nos jours, PPDM est devenu une étape incontournable et essentielle. En effet, les méthodes Data Mining classiques ont été réadaptées et enrichies par de nouveaux mécanismes pour assurer la sécurité des données. Plusieurs solutions ont été proposées. Généralement, elles sont classées selon les techniques utilisées. Principalement, ils existent deux techniques d‘approches différentes, chacune à des avantages et des inconvénients. La première est la technique de perturbation des données. Elle consiste à modifier les données en amont pour dissimuler toutes possibilités d‘identification directes ou indirecte des patients. Ils existent différentes approches de perturbation, telles que : la généralisation, la discrétisation, la K-anonymat, l-diversité, etc. L‘avantage de ces méthodes est qu‘elles ne nécessitent pas beaucoup de calcul mais en contrepartie, il est difficile de préserver les performances initiales des modèles Data Mining parce que l‘utilité des données est sacrifiée au profit de la sécurité. En effet, le challenge avec ces méthodes est de trouver un compromis satisfaisant entre l‘utilité et la sécurité des données. La deuxième technique est basée sur le calcul multiparti sécurisé. Des parties participent à un calcul commun sans se partager les données sensibles. Cette technique peut inclure des techniques cryptographiques, de tierce partie, etc. Contrairement à la première approche, cette approche préserve les performances des modèles Data Mining intactes mais nécessite beaucoup de calcul et de communication réseaux.
Par la présente thèse, nous avons traité le problème de la sécurité des données biomédicales lors de l‘élaboration d‘un processus Data Mining collaboratif dans un environnement distribué. Nous avons proposé des solutions originales et sécurisées pour la phase de
prétraitement des données, la phase conception du modèle et finalement, la phase exploitation du modèle. Les résultats des expérimentations ont validé nos approches.
2 Problématique
1. Les données de santé, collectés par les organismes médicaux sur les patients, sont classées données spéciales et hautement confidentielles, selon le RGPD. L‘exploitation de ces données sensibles est réglementée par des lois juridiques très sévères. Sans le consentement et le respect de la vie privée de leurs propriétaires, le contrevenant risque de lourdes peines (amendes et prison). En conséquence, le processus Data Mining biomédical est directement concerné par ses mesures juridiques. L‘accès aux données médicales pour effectuer un apprentissage automatique et générer des modèles ne peut pas être effectué sans le respect de la loi (Tilman, 2015). Face à cette situation, la mise en place de nouvelles solutions, incluant le respect de la vie privée des patients, s‘avère obligatoire.
2. Le Data Mining collaboratif est bénéfique pour le domaine médical. Le croisement des bases de données, de même ou différentes spécialités, permet d‘élargir et d‘enrichir la base d‘apprentissage. Par exemple, les données sur l‘exposition des personnes aux produits chimiques couplées avec le fichier de décès pourraient aider à identifier les substances cancérogènes (Chukka, Swathi & Nagendranath, 2012). Toutefois, cette démarche est confrontée à des obstacles, parmi eux :
2.1. La pertinence des données utilisées :
L‘utilisation des données externes pour générer des modèles Data Mining plus performants peut conduire à un résultat inverse. Les performances initiales peuvent être dégradées à cause des attributs non pertinents ou une combinaison inadéquate d‘attributs.
La sélection des attributs pertinents à partir des bases de données distribuées appartenant à de tierces parties est une tâche difficile. Ajouter à cela la contrainte du respect de la vie privée des patients, la tâche s‘annonce un challenge encore plus difficile. La plupart des solutions proposées sont de type ‗Filter‘ et elles utilisent des techniques de perturbation de données. Ces dernières ont l‘inconvénient de réduire les performances en contrepartie de la sécurité des données personnelles. Or, dans le domaine médical, les performances sont essentielles, car 1% de plus en performances pour diagnostiquer les maladies peut sauver plus de vies humaines. A cet effet, il faut
Introduction Générale
concevoir des solutions, sécurisées et plus performantes, pour effectuer une sélection d‘attributs pertinents dans un environnement distribué.
2.2. La redondance des données :
Les mêmes patients peuvent visiter différents organismes de santé durant leurs vies, selon leurs maladies ou leurs lieux de résidences. De ce fait, lors de l‘élaboration d‘un processus Data Mining collaboratif sur des données distribuées, des instances (enregistrement) peuvent être dupliqués (redondant). D‘après (Kołcz, Chowdhury &
Alspector, 2003), cela influe négativement sur les performances des processus Data Mining.
Les solutions existantes, basées sur les techniques de hachage telles que MD5 et SHA1, présentent des défaillances de sécurité. Grace aux avancées technologiques dans plusieurs domaines (puissance de calcul, réseaux, Cloud, etc.), la sécurité de ces méthodes a pu être cassez par la cryptanalyse. En conséquence, les données biomédicales sensibles cryptées par ces méthodes ne sont plus en sécurité. Les hackers peuvent faire un reverse engineering et extraire des informations sensibles. A cet effet, des solutions robustes et sécurisées pour la déduplication des enregistrements redondants dans un environnement distribué, sans porter atteinte à la vie privée, sont indispensables.
2.3. Dans un processus Data Mining collaboratif utilisant des données biomédicales verticalement distribuées :
- La conception du modèle est effectuée selon les patients du site principal qui détient l‘attribut classe. Cependant, les autres sites n‘ont pas forcément le même nombre de patients. Alors, comment sélectionner, d‘une manière sécurisée, sans avoir accès aux données sensibles, seuls les patients concernés et ce, à travers tous les sites collaboratifs.
- Les solutions existantes prévoient le partage du modèle Data Mining final. Cela peut constituer une faille de sécurité parce que le modèle peut être révélateur d‘informations sensibles. C‘est le cas du classificateur Naïve Bayes qui affiche des données statistiques telles que la moyenne et la variance des attributs numériques ainsi que les probabilités des valeurs catégoriques.
- Si le modèle calculé n‘est pas partagé entre les sites collaboratifs, le cas des données verticalement distribuées, alors comment classer les nouvelles instances soumises intégralement à un seul site. Ce dernier n‘a pas tous les attributs. Il doit
3 Contributions
Le processus d‘extraction des connaissances à partir de données (ECD), dénommé en anglais ‗Knowledge Discovery in Databases‘ (KDD), est définie, selon (Han, Pei & Kamber, 2011) (Preux, 2011), en plusieurs étapes comme suit :
1- Collecte des informations et organisation de ces infos dans une base de données. 2- Nettoyage des données.
3- Intégration des données, dans le cas de plusieurs sources. 4- Sélection des données pertinentes pour analyse.
5- Transformation et consolidation des données dans des formats appropriés par agrégation, etc.
6- Fouille des données (processus Data Mining) partie importante dans le processus ECD. Des algorithmes intelligents (machine learning) sont utilisés pour extraire de la connaissance.
7- Evaluation des résultats. Identification des connaissances intéressantes.
8- Présentation, visualisation des connaissances découvertes d‘une manière adéquates à l‘utilisateur final.
9- Exploitation du modèle Data Mining.
Pour être conforme aux lois juridiques relatives à la protection de la vie privée des patients, la sécurité des données doit être considérer à toutes les étapes du processus ECD (Malik, Ghazi & Ali, 2012). Aucune information sensible ne doit être divulguée, si elle va être céder ou manipuler par une tierce partie. Dans cette thèse, nous avons apporté nos contributions dans certaines étapes du processus ECD comme suit :
Déduplication des données pour l’étape prétraitement et intégration virtuelle des données : Pour la déduplication des enregistrements biomédicaux redondants dans un environnement distribué, dans le cadre du respect de la vie privée des patients, nous avons proposé une solution originale basée sur des techniques de hachages standards mais renforcés par des techniques inspirées des algorithmes génétiques. En effet, de nouveaux codes haches sont créés en utilisant des codes haches standards (MD5, SHA1, SHA256, SHA512, etc.) et les techniques de croisement et de mutation.
Introduction Générale
Sélection d’attributs pour l’étape sélection des données :
Pour la sélection d‘attributs, dans un environnement distribué, nous avons proposé des solutions originales de type ‗Wrapper‘ adaptées à des données horizontalement ou verticalement réparties. Dans notre solution :
La prise en considération de la contrainte ‗protection de vie privée des patients‘ n‘influe pas sur les performances initiales des modèles Data Mining générés, ce qui est essentiel et important dans le domaine médical.
Les données originales d‘apprentissage de chaque site collaboratif ne sont ni perturbées ni divulguées.
Les résultats intermédiaires des traitements ne sont pas partagés entre les sites collaboratifs.
Conception du modèle Data Mining pour l’étape apprentissage automatique : Nous avons proposé plusieurs solutions, selon les cas suivants :
Des organismes médicaux traitant le même sujet (distribution horizontale) désirent collaborer, sans se partager les données sensibles, pour concevoir un modèle Data Mining plus performants. Deux solutions ont été proposées :
- La première solution est basée sur l‘utilisation de deux crypto systèmes (Pallier et RSA) pour effectuer un calcul collaboratif sécurisé et circulaire sans utiliser une tierce partie. Le cryptosystème de Pallier est utilisé pour calculer les paramètres du modèle Naïve Bayes et sécuriser les résultats intermédiaires entre les sites des attaques externes. Le cryptosystème RSA est utilisé pour sécuriser les résultats intermédiaires des attaques internes provenant du site maitre. Ce dernier détient les clés de cryptage. Donc, il peut à tout moment intercepter le trafic échangé entre les sites collaboratifs et déduire des informations sensibles.
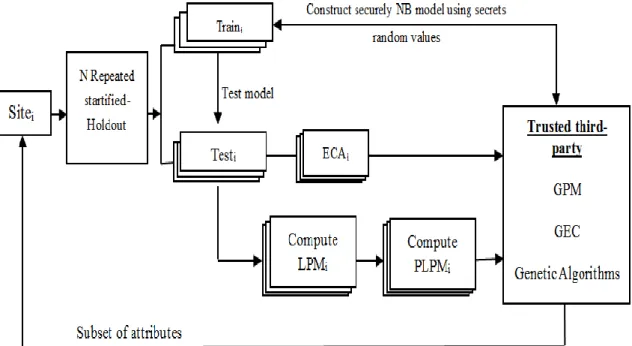
- La deuxième solution fait appelle à une partie tierce de confiance (exemple : Cloud). Les données sensibles des patients ne sont pas partagées et des variables aléatoires secrètes sont utilisées pour dissimuler les résultats intermédiaires lors de l‘envoi à la tierce partie pour effectuer des calculs.
Des organismes médicaux traitant des sujets différents (distribution verticale) désirent collaborer pour concevoir un modèle Data Mining. Pour cela, nous avons proposé une solution qui permet de concevoir un modèle distribué sans divulguer les données
biomédicales sensibles locales, les données intermédiaires de calcul et les sous modèles générés dans chaque site.
Aussi, nous avons traité le cas où les sites collaboratifs possèdent un nombre différent de patients. Pour sélectionner seulement les patients concernés par le Data Mining collaboratif selon les patients du site maitre, nous avons proposé une solution sécurisée basée sur la technique de hachage et une technique de trie privée.
Classification de nouvelles instances pour l’étape exploitation du modèle : Nous avons proposé une solution dans le cas où :
- Les données médicales sensibles sont verticalement distribuées entre les sites. - Le modèle final n‘est pas partagé entre les sites. Chaque site détient sa partie
du modèle, calculé localement.
- Chaque site peut générer plusieurs sous modèles privés.
- Les nouvelles instances à classer sont soumises intégralement au site maitre, l‘initiateur du processus. Ce dernier n‘a pas tous les attributs. Donc, il doit solliciter d‘une manière sécurisée les autres sites collaboratifs. La solution proposée inclue une tierce partie, des crytosystèmes homomorphes.
Ces contributions seront détaillées dans les prochains chapitres.
4 Organisation de la thèse
La thèse est organisée en six (06) chapitres. Le premier chapitre présente le Data Mining et la classification supervisée des données biomédicales. Le deuxième chapitre aborde le respect de la vie privée. Le troisième chapitre présente l‘approche proposée pour la déduplication et la classification sécurisée des données biomédicales distribuées horizontalement. Le quatrième chapitre décrit une approche originale et sécurisée pour la sélection des attributs pertinents à partir des bases de données biomédicales distribuées horizontalement ou verticalement. Le cinquième chapitre détaille une solution originale pour la classification de nouvelles instances dans un environnement distribué verticalement en utilisant des sous modèles Data Mining, privés et non partagés. Le dernier chapitre dresse la conclusion générale ainsi que les futurs travaux.
Chapitre 1 Classification des données biomédicales
Chapitre 1.
Classification des données
biomédicales
Sommaire
1.1. Digitalisation du domaine médical ... 25 1.1.1. Introduction ... 25 1.1.2. Dossier patient (dossier médical) ... 26 1.1.2.1. Définition ... 26 1.1.2.2. Objectif du dossier patient ... 27 1.1.2.3. Rôle des acteurs intervenant dans le dossier médical de patient ... 28 1.2. Data Mining ... 29 1.2.1. Définition du Data Mining (fouille des données) ... 29 1.2.2. Processus Data Mining ... 29 1.2.2.1. Prétraitement des données ... 30 1.2.2.2. Types d‘apprentissage ... 31 1.2.2.2.1. Apprentissage supervisé ... 31 1.2.2.2.2. Apprentissage non supervisé ... 31 1.2.2.2.3. Apprentissage semi-supervisé ... 31 1.2.2.3. Les méthodes du processus Data Mining ... 32 1.2.3. Classification supervisée ... 32 1.2.4. La matrice de confusion ... 33 1.2.5. Algorithmes de la classification supervisée ... 34 1.2.5.1. Naïve Bayes ... 34
1.2.5.3. Arbres de décision ... 36 1.2.5.4. Réseaux de neurones ... 37 1.2.5.5. K plus proches voisins (KNN) ... 37 1.3. Data Mining biomédical ... 38 1.3.1. Définitions ... 38 1.3.1.1. Définition du biomédical ... 38 1.3.1.2. Définition du Data Mining biomédical ... 39 1.3.2. Travaux de recherches ... 39 1.3.3. Projets connus incluant le Data Mining dans le domaine médical ... 42 1.4. Classification supervisée dans le domaine biomédical ... 44 1.5. Conclusion ... 49
Chapitre 1 Classification des données biomédicales
1.1. Digitalisation du domaine médical 1.1.1. Introduction
L‘utilisation des nouvelles technologies de l‘information dans le domaine médical a permis de muter la médecine traditionnelles vers d‘autres pratiques modernes où elle est plus proactive, précise, personnalisée et rapide pour diagnostiquer et soigner les maladies et ce, à un coût compétitif. La digitalisation numérique a facilité l‘accès, le transfert et le partage d‘informations médicales qui sont essentielle pour la prise de décision (Dumez, Minivielle, & Marrauld, 2015). Elle a créé un moyen de communication et de coopération rapide entre les différents acteurs du domaine de la santé (médecins généralistes, médecins spécialistes, les chirurgiens, les radiologues, le personnel soignant, les pharmaciens, etc.). En conséquence, cela a permis une meilleure prise en charge des patients.
Conscient de ce changement incontournable et stratégique, tous les organismes de santés publiques et libérales, à savoir les hôpitaux, les cliniques, les laboratoires, les centres de radiologies, les centres d‘analyse, etc., ont informatisé leurs documents et leurs processus. En effet, ils ont :
- Numérisé tous les documents papiers, surtout le dossier patient qui est le noyau de
cette digitalisation (Meingast, Roosta & Sastry, 2006)(Andonova & Vacher, 2013) (Ficatier, 2008)(Cordelier, 2012) ;
- Acquis des ordinateurs, des serveurs et des solutions de stockage ; - Installé des réseaux informatiques ;
- Intégré des outils et des équipements médicaux, numériques et connectés, pour
bénéficier d‘une plus grande précision et efficacité ;
- Développé des systèmes d‘information hospitaliers (SIH) pour faciliter la gestion
des informations médicales et la gestion administratives (Degoulet, 2007) ;
- etc.
Mais en contrepartie, à l‘instar des autres domaines, et grâce aux progrès technologiques qui ont réduits les coûts du stockage, la numérisation du domaine de la santé a provoqué une grande explosion des données (Béranger, 2016). Cela est dû aux :
- Les SIH, qui stockent toutes les données relatives aux patients admis à un
organisme médical (ANAP, 2016). A savoir : tous les résultats des diagnostiques, les données des essais cliniques, les données génétiques, les données biocliniques, les données pathologiques, les prescriptions de pharmacie, les résultats de laboratoires, les radiologies, les scanners, etc.
- Les objets médicaux connectés (IoT) génèrent aussi un grand flux de données en
permanence sur les patients et à n‘importe quel endroit (au travail, à domicile, en promenade, pendant le sommeil, etc.). Différents indices de mesures (telles que le taux de sucre dans le sang, le rythme cardiaque, la température corporelle, la tension, l‘alimentation, les déplacements, la prise des médicaments, le poids, les paramètres environnementaux, etc.) sont collectées et stockées dans de grande base de données.
- Etc.
De nos jours, avec le développement des techniques Data Mining, cette grande masse de données constitue un précieux trésor pour extraire de la connaissance, non visible, qui sera utilisée pour développer le secteur de la santé. Dans ce chapitre, nous allons décrire les avantages issus du croisement entre l‘intelligence artificielle et la science de la médecine.
1.1.2. Dossier patient (dossier médical)
La fouille des données biomédicales, pour concevoir des modèles Data Mining, passe obligatoirement par l‘usage du dossier patient. Ce dernier est un élément clé dans la médecine d‘aujourd‘hui. Il est au cœur de toute réflexion dans le domaine de la santé
(Lievre & Moutel, 2010). Pour cela, nous jugeons qu‘il est important de le définir brièvement.
1.1.2.1. Définition
Le dossier patient est le lieu de recueil et de conservation de toutes les informations médicales et administratives concernant un patient admis à un organisme de soin, publique ou libéral. Il retrace tout l‘historique des maladies et des soins du patient (Pelletier, 2003). Le dossier regroupe les informations suivantes :
Chapitre 1 Classification des données biomédicales
Les comptes rendus des consultations, des interventions subis, d'exploration ou d'hospitalisation,
La nature des soins prescrits,
Le dossier d‘anesthésie,
Les actes transfusionnels,
Les soins infirmiers,
Les résultats d‘analyses médicales,
Les résultats des radios, des scanners, IRM, etc.,
Les protocoles et les prescriptions thérapeutiques mis en œuvre,
Les médicaments prescrits,
Les feuilles de surveillance,
Les correspondances entre professionnels de santé,
Ainsi que toutes les autres informations médicales ou paramédicales, relatives aux patients qui doivent être consignées dans le dossier patient.
1.1.2.2. Objectif du dossier patient
Son enjeu est considérable dans le suivi des soins et la coordination pluridisciplinaire, ces avantages sont très nombreux (ANEAS, 2003) :
- C‘est un outil de communication, de coordination et d‘information entre les acteurs
de soin et avec les patients, car il permet de :
Mettre à disposition des informations utiles et nécessaires pour la prise en charge et le suivi du patient.
Faciliter et accélérer l‘échange d'informations entre les professionnels pour coordonner, éviter les actes redondants et agir contre les interactions médicamenteuses.
Assurer la continuité des soins.
- C‘est un outil d‘évaluation de soins.
- Aide à la décision thérapeutique par son contenu.
- Traçabilité des soins et des actions entreprises vis-à-vis du patient. - Lieu de recueil de tous les consentements du patient.
- Rôle juridique important dans le cas d‘une recherche de responsabilité.
- Fournis des informations nécessaires pour l‘enseignement et la recherche
- Etudes épidémiologiques.
- Fournis des informations nécessaires à l'analyse médico-économique de l'activité. - Etc.
1.1.2.3. Rôle des acteurs intervenant dans le dossier médical de patient
Le dossier patient constitue un vivier d‘information sûre pour effectuer un processus Data Mining, parce que ça gestion et sa tenue à jour est réglementée par des lois juridiques, à travers le monde entier. Tout contrevenant risque des mesures disciplinaires sévères. Par exemple : en France, un blâme a été infligé par la chambre disciplinaire de la première instance de l‘Ordre des Médecins PACA à un praticien pour ne pas avoir tenu des dossiers patients dans les conditions posées par l‘article R. 4127-45 CSP (Vade_mecum, 2012). Afin de maintenir le dossier patient à jour, le rôle de chaque intervenant est clairement défini par des instances de réglementation. Par exemple, la Haute Autorité de la Santé française (HAS3), a défini clairement les rôles et les responsabilités, de chacun des différents acteurs dans le domaine de la santé, pour la tenue du dossier patient comme suit
(ANAP, 2018) :
Les médecins hospitaliers, tous statuts confondus, doivent consigner toutes leurs constats, leurs observations, leurs interventions et leurs conclusions dans le dossier du patient.
Les sages-femmes doivent également tracer leurs interventions, observations et traitements instaurés
Les professionnels paramédicaux (infirmiers, kinésithérapeutes, diététiciennes, orthophonistes, etc.) constituent un dossier, appelé dossier de soins paramédical, contenant la trace de leurs observations, transmissions ciblées et actions de soins, assurant la continuité des soins.
Les rapports d'un psychologue ou d'un travailleur social font partie intégrante du dossier patient s‘ils ont été réalisés au sein d‘une équipe dirigée par un médecin et qu‘ils ont été joints au dossier du patient. Ces informations doivent pouvoir être accessibles aux autres professionnels, si elles sont utiles à la prise en charge du
3
La Haute Autorité de santé (HAS) est une autorité publique indépendante à caractère scientifique, créée par la loi du 13 août 2004 relative à l‘Assurance maladie. Depuis le 1er avril 2018, son périmètre s‘est élargi aux champs social et médico-social avec l‘intégration de l‘Agence nationale de l‘évaluation et de la qualité des établissements et services sociaux et médico-sociaux (Anesm). La HAS envisage ainsi la santé dans sa globalité (HAS, 2018).
Chapitre 1 Classification des données biomédicales
patient. Dans tous les cas, l‘intervention du psychologue ou du travailleur social doit être renseignée.
Etc.
En conclusion, le dossier patient est fiable et riche en informations précieuses qui peuvent servir pour tout type d‘études ou recherches scientifiques. Cependant, le partage d‘information sensibles est soumis à des lois juridiques strictes qui réglementent l‘accès au dossier et interdisent toute atteinte à la vie privée des patients.
1.2. Data Mining
1.2.1. Définition du Data Mining (fouille des données)
La première définition a été donnée en 1996 par USAMA Fayyad.
"a new generation of computational theories and tools to assist humans in extracting useful information (knowledge) from the rapidly growing volumes of digital data"
(Fayyad, 1996).
Le Data Mining consiste à rechercher et à extraire des informations utiles, non évidentes, à partir de gros volumes de données stockées dans des bases ou des entrepôts de données
(Preux, 2011). C‘est un outil qui permet de trouver des structures originales et des
corrélations informelles entre les données. Le Data Mining permet de mieux comprendre les liens entre des phénomènes en apparence distincts et d'anticiper des tendances encore peu discernables (ANAP, 2015).
1.2.2. Processus Data Mining
Le processus de la fouille de données, en anglais Knowledge Data Discovery (KDD), comprend les étapes suivantes (Preux, 2011) :
1. Collecte des informations et organisation de ces infos dans une base de données ; 2. Nettoyage de la base de données pour corriger les erreurs.
3. Sélection des attributs pertinents ;
4. Extraction de connaissances d‘une base de données (Data Mining) ;
6. Evaluation des résultats de l‘extraction de connaissance.
Le processus peut être résumé en trois phases successives : la phase prétraitement des données (étapes : 1, 2 et 3). La phase découverte des phénomènes fréquents par la fouille des données (étape 4). Finalement, la mise en forme et l‘évaluation des connaissances extraites (étape 5 et 6).
1.2.2.1. Prétraitement des données
Le prétraitement des données est décisif pour la construction des modèles. Un choix inapproprié d‘échantillons d‘apprentissage, dans le cas de la classification supervisée, peut faire échouer le processus Data Mining (Zighed & Rakotomalala, 2002). Le prétraitement des données permet d‘améliorer la qualité des données fouillées par les algorithmes d‘apprentissage automatiques. Il ‗agit du :
- Traitement des données manquantes.
- Traitement des données invalides et aberrantes. - La déduplication des données redondantes, - La normalisation des données,
- La sélection d‘attributs. - Etc.
Chapitre 1 Classification des données biomédicales
1.2.2.2. Types d’apprentissage
Les algorithmes d'apprentissage peuvent se catégoriser selon le mode d‘apprentissage employé. Ils existent trois méthodes d'apprentissage automatique (Brownlee, 2016) (Chapelle, Scholkopf & Zien, 2009) :
1.2.2.2.1. Apprentissage supervisé
L‘apprentissage est dit supervisé lorsque les données utilisées dans le processus sont déjà catégorisées, et que les algorithmes d‘apprentissage automatiques doivent s‘en servir pour prédire un résultat en vue de pouvoir le faire plus tard lorsque les données ne seront plus catégorisées (Bob, 2018).
Le processus se déroule en deux étapes : la phase d‘apprentissage et la phase de test. Avant la phase d‘apprentissage, un expert doit, au préalable selon un objectif bien défini, étiqueter les exemples soigneusement sélectionnés. Puis, un modèle data Mining est construit à partir de ces données en utilisant des algorithmes d‘apprentissage automatiques. La phase test consiste à tester les performances du modèle à prédire correctement les étiquettes de nouvelles instances (Metomo, 2017).
1.2.2.2.2. Apprentissage non supervisé
La classification non-supervisée consiste à apprendre sans superviseur. A partir d‘une population, il s‘agit d‘extraire des classes ou groupes d‘individus présentant des caractéristiques communes, le nombre et la définition des classes n‘étant pas données a priori (Cleuziou, 2004). Les méthodes d'apprentissage non supervisées peuvent examiner des données complexes, plus expansives et apparemment sans point commun, afin de les organiser de manière potentiellement significative.
1.2.2.2.3. Apprentissage semi-supervisé
C‘est une hybridation entre l‘apprentissage supervisée et le non supervisée. Il est utilisé lorsque la base d‘apprentissage est constituée d‘un petit nombre de données étiquetées et d‘un grand nombre de données non étiquetées. Ils existent différentes méthodes telles que : Generative models, Low-density separation, Graph-based methods, Heuristic approaches, etc (Zhu, 2006).
1.2.2.3. Les méthodes du processus Data Mining
Les différentes méthodes du Data Mining se résument, brièvement, comme suit
- Classification : examiner les caractéristiques d‘un objet et lui attribuer une classe. e.g. décision d‘attribution de prêt à un client.
- Prédiction : prédire la valeur future d‘un attribut en fonction d‘autres attributs. e.g. prédire la qualité d‘un client.
- Association : déterminer les attributs qui sont corrélés (associés). e.g. analyse du panier de la ménagère.
- Segmentation : former des groupes homogènes à l‘intérieur d‘une population.
1.2.3. Classification supervisée
La classification supervisée est une technique largement utilisée avec différentes applications dans la vie réelle (Kalmegh, 2015). Elle permet de générer des règles de classification (modèle) à partir d‘un jeu données classées à priori et d‘un algorithme d‘apprentissage automatique adéquat. Ces règles seront utilisées pour classer les nouvelles instances.
Soit D un ensemble de n exemples et de m attributs (n x m). Y= {y1, y2, .., yp} est l‘attribut classe avec p valeurs classes possibles. Chaque instance xi ∈ D est caractérisée par m attributs et par sa classe yi ∈ Y.
L‘objectif est, en s‘appuyant sur l‘ensemble d‘exemples étiqueté X et un classificateur CL, de prédire la classe des nouvelles instances.
Cl(x)=y, x est une nouvelle instance non étiquetée. y ∈ Y
Une classification est dite binaire, si le nombre de classes |Y| est égale à 2. Le classificateur doit prédire l‘une des deux classes pour les nouvelles instances. Une classification est dite multi classes si le nombre de classes |Y| > 2.
Pour évaluer un processus Data Mining, comparer les algorithmes d‘apprentissage et améliorer les performances sur un jeu de données, ils existent différentes techniques d‘évaluation, telles que holdout, k-cross validation et leave-one-out.
Chapitre 1 Classification des données biomédicales
Holdout:
La méthode holdout partitionne le data set Dn de n instances en deux parties. Une partie Dk pour l‘apprentissage et une partie Dt pour le test, généralement Dk=2/3 et Dt=1/3 (Kohavi,
1995).
K-cross validation :
Appelée estimation rotative (Kohavi, 1995), le dataset D est divisé aléatoirement en k sous ensemble mutuellement exclusifs D1,D2, ... ,Dk / D = D1 UD2 ..UDk. Le classificateur est testé k fois. A chaque t ∈ {1, 2, .., k}, l‘apprentissage se fait sur le dataset D-Dt et testé sur Dt. La performance finale P est la moyenne de toutes les performances Pt. Elle est calculée comme suit :
∑ (1. 1)
Leave-one-out :
La méthode leave-one-out est tout simplement la méthode k-cross validation avec k = n, n étant le nombre d‘instances.
1.2.4. La matrice de confusion
Dans le contexte de la classification supervisée, la matrice de confusion, appelée aussi matrice de contingence, est un outil qui sert à évaluer les performances d‘un algorithme de classification. Elle synthétise les informations sur les classes réelles et les classes prédites par le modèle. Les colonnes de la matrice représentent les classes estimées et les lignes représentent les classes réelles des instances testées. Différentes métriques sont calculées à partir de la matrice de confusion. Nous citons quelques-unes, calculées dans le cas d‘une classification binaire (Table 1.1), comme suit :
Table 1.1. Matrice de confusion pour une classification supervisée binaire
Classes Actuelles
Classes Prédites
Positif Négatif
Positif Vrai positif (TP) Faux Négatif (FN) Négatif Faux positif (FP) Vrai Négatif (TN)
(1. 3) (1. 4) (1. 5) (1. 6) (1. 7)
La mesure F-score, appelée également F-mesure ou F1, fournit une mesure plus réaliste sur les performances en fonction des classes. Elle calcule la moyenne harmonique et pondérée entre la précision et le rappel. La précision, également appelée valeur prédictive positive, correspond à la proportion de résultats positifs réellement positifs. Le rappel, également appelé sensibilité, est la capacité d'un test à identifier correctement les résultats positifs pour obtenir le taux de réponse positif réel.
1.2.5. Algorithmes de la classification supervisée
Ils existent différents algorithmes pour l‘extraction de la connaissance selon l‘objectif et le type d‘apprentissage. Bien que deux algorithmes d‘apprentissage puissent différer dans leur type d‘apprentissage, i.e. la nature de leur objectif, ils peuvent aussi se distinguer par la façon qu‘ils accomplissent cet apprentissage. Nous allons passer en revue quelques approches populaires utilisées dans l'apprentissage automatique.
1.2.5.1. Naïve Bayes
Naïve Bayes est un classificateur probabiliste simple. Il calcule un ensemble de probabilités en comptant la fréquence et les combinaisons de valeurs dans un jeu de données. L'algorithme utilise le théorème de Bayes et suppose que tous les attributs sont indépendants compte tenu de la valeur de la variable classe. Cette hypothèse d'indépendance conditionnelle est rarement valable dans les applications du monde réel, d'où la caractérisation naïve. Cependant, l'algorithme tend à bien fonctionner et à apprendre rapidement dans divers problèmes de classification supervisée (Patil & Shereker, 2013).
Compte tenu de la classe y et du vecteur de données (x1, x2, x3,…, xn), le théorème de Bayes énonce la relation suivante:
Chapitre 1 Classification des données biomédicales | | (1. 8) | ∏ | (1. 9) Pour tous les ‗xi‘, cette relation est simplifiée comme suit:
| ∏ |
(1. 10)
Puisque P (x1, x2, x3,…, xn) est constant pour l'entrée, nous pouvons utiliser la règle de classification suivante:
∏ |
(1. 11)
1.2.5.2. Support Vector Machine (SVM)
Le classificateur SVM, développée par Vladimir Vapnik en 1995, est un classificateur puissant, il a fait ses preuves dans plusieurs domaines. Le principe est de projeter les données qui sont non linéairement séparables dans un autre espace de dimension plus élevée où elles peuvent le devenir, en utilisant différents noyaux. Le but du SVM binaire est de trouver un hyperplan optimal qui sépare les deux classes en maximisant la distance. Cette distance est appelée marge. Dans le cas d‘une classification binaire, Figure 1.2, l‘hyperplan est une droite. Les points les plus proches, qui seuls sont utilisés pour la détermination de la marge, sont appelés vecteurs de support.
L‘hyperplan séparateur est représenté par l‘équation
(1. 12)
w est un vecteur de m dimensions et b est un terme (Djeffal, 2012). La fonction de décision, pour un exemple x, peut être exprimée comme suit :
{
(1. 13) Maximiser la marge revient maximiser
Figure 1.2. SVM classification binaire
SVM réduit le problème multi classe à une composition de plusieurs hyperplans bi-classe permettant de tracer les frontières de décision entre les différentes classes. Il décompose l‘ensemble d‘exemples en plusieurs sous-ensembles représentant chacun un problème de classification binaire. A chaque fois un hyperplan de séparation est déterminé par la méthode SVM binaire. On construit lors de la classification une hiérarchie des hyperplans binaires qui est parcourue de la racine jusqu‘à une feuille pour décider de la classe d‘un nouvel exemple (Djeffal, 2012).
On a donc une transformation d‘un problème de séparation non linéaire dans l‘espace de représentation en un problème de séparation linéaire dans un espace de redescription de plus grande dimension. Cette transformation non linéaire est réalisée via une fonction noyau. En pratique, quelques familles de fonctions noyau paramétrables sont connues et il revient à l‘utilisateur de SVM d‘effectuer des tests pour déterminer celle qui convient le mieux pour son application. On peut citer les exemples de noyaux suivants : polynomiale, gaussien, sigmoïde et laplacien (Zaiz, 2010).
1.2.5.3. Arbres de décision
Les arbres de décisions sont des techniques très populaires par leur efficacité et leur simplicité dans le domaine de la classification supervisée. Ils fournissent une représentation graphique du modèle facilement interprétable (Santos, 2015). Le modèle final est constitué d‘un nœud racine et des nœuds intermédiaires, des branches et des feuilles. La racine est le point d‘entrée à l‘arbre. Les feuilles représentent les valeurs
Chapitre 1 Classification des données biomédicales
classes à prédire. Les branches représentent les résultats de test relatif à chaque nœud. Pour effectuer une classification, l‘arbre est parcouru de la racine aux feuilles selon une série de tests à chaque niveau de l‘arbre. La théorie de Shannon est à la base de partitionnement de plusieurs arbres de décision. Elle est définie comme suit :
∑ (1. 14)
(1. 15)
nj représente le nombre d‘instances appartenant à la classe j et ns représente le nombre total d‘instance du nœud s.
Le gain d‘information est mesuré par la différence entre l‘impureté du nœud parent s et la somme des impuretés des p nœuds fils obtenus grâce à un attribut X (Taleb Zouggar, 2014).
∑
(1. 16)
nj représente le nombre d‘instances total du nœud s et n représente le nombre d‘instances total du nœud parent.
1.2.5.4. Réseaux de neurones
En apprentissage automatique, les réseaux de neurones constituent une famille d‘algorithmes d‘apprentissage statistique inspirés des réseaux de neurones biologiques. Ils sont généralement présentés comme des systèmes de "neurones" interconnectés, capables de calculer des valeurs à partir d'entrées/sorties. Le réseau reçoit les informations sur une couche réceptrice de neurones, traite ces informations avec ou sans l‘aide d‘une ou plusieurs couches « cachées » contenant un ou plusieurs neurones et produit un signal (ou plusieurs) sorties (Benyahia, 2012). Les réseaux de neurones sont très utilisés pour l'apprentissage automatique. Ils ont connu un grand succès et beaucoup d‘applications, surtout pour la reconnaissance des formes (Deep learning).
1.2.5.5. K plus proches voisins (KNN)
Le principe de la méthode KNN est de trouver k plus proches voisins, à partir de l‘échantillon d‘apprentissage, à une nouvelle instance qu‘on cherche à classer. La classe de
la nouvelle instance est la classe majoritaire (la plus représentée) parmi ces k voisins. Dans le cas d‘une régression, la valeur de sortie est une valeur continue qui peut être, par exemple, la moyenne des valeurs des k voisins.
Il existe plusieurs fonctions pour calculer la distance entre deux voisins, notamment, la distance euclidienne, la distance de Manhattan, la distance de Minkowski, la distance de Jaccard, etc. Dans ce qui suit, noud définissons la distance euclidienne et la distance de manhattan.
Soit un vecteur représentant les variables prédicatrices de l‘instance i. La distance euclidienne entre les deux instances est définie comme suit (Mathieu-Dupas, 2010) (Mulak & Talhar, 2015) :
( ) √∑ (1. 17) ( ) ∑ | | (1. 18)
Il est à noter que, KNN n‘a pas une phase d‘apprentissage où un modèle Data Mining est généré. Les exemples d'apprentissage sont des vecteurs dans un espace multidimensionnel avec un label de classe d'appartenance, ils sont stockés en permanence dans la mémoire lors de la phase de classification.
1.3. Data Mining biomédical 1.3.1. Définitions
1.3.1.1. Définition du biomédical
Selon le dictionnaire Larousse, c‘est un domaine qui concerne à la fois la biologie et la médecine.
La recherche biomédicale est un domaine d‘études qui vise à améliorer les connaissances en matière de santé. Pour ce faire, elle se base notamment sur le recueil d‘informations à caractère biologique et/ou médical (ARCAT, 2019).