HAL Id: tel-02821273
https://hal.inrae.fr/tel-02821273
Submitted on 6 Jun 2020HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
applications aux chromosomes de mammifères
Hélène Hayes
To cite this version:
Hélène Hayes. Cytogénétique classique moléculaire et comparée : applications aux chromosomes de mammifères. Sciences du Vivant [q-bio]. 2006. �tel-02821273�
UNIVERSITE de VERSAILLES SAINT-QUENTIN-EN-YVELINES
UFR Médecine/Biologie
Spécialité : Génétique CNU 4704
CANDIDATURE A L’HABILITATION A DIRIGER DES RECHERCHES
présentée par
Hélène HAYES
Cytogénétique classique, moléculaire et comparée :
applications aux chromosomes de mammifères
Unité de Génétique biochimique et Cytogénétique
Institut National de la Recherche Agronomique
Centre de Recherches de Jouy-en-Josas
78352 Jouy-en-Josas Cedex
Hélène HAYES
Née le 20 août 1957 Nationalité: française Unité de Génétique biochimique et Cytogénétique, INRA-CRJ78352 Jouy-en-Josas Cedex Tél. 01 34 65 26 73 Fax 01 34 65 24 78
Email helene.hayes@jouy.inra.fr Diplômes
Juin 1976 Baccalauréat C, Paris
Juin 1978 DEUG Sciences de la Nature et de la Vie, univ Paris VI Juin 1980 Maîtrise de Biochimie, université Paris VI
Juin 1981 DEA Biochimie Industrielle et Alimentaire, univ Paris VII
Mars 1984 Thèse de troisième cycle de Sciences Alimentaires, univ Paris VII Juin 1987 Maîtrise de Biologie et Génétique Appliquée, univ Paris VII
Octobre 1993 Thèse de doctorat de Biodiversité, Génétique et Evolution, univ Paris VII Stages et formations complémentaires
Janvier 1980 Laboratoire de Chimie Organique Biologique,univ Paris VI (A Marquet) Essais de synthèse de l’acide chrysanthémique
Janvier-Juin 1981 Laboratoire de Technologie Alimentaire, INRA Massy (MT Tollier) Composition en sucres de l’orge selon les conditions de croissance
Juillet-Août 1981 Institute of Cancer Research, Columbia university, New York, USA (P Feigelson) Essais de clonage du gène de la tryptophane oxygenase hépatique de rat Sept 1981-Mars 1984 Laboratoire de Microbiologie Laitière, INRA Jouy-en-Josas (JL Bergère)
Allocataire du Ministère de la Recherche et de l’Industrie Etude biochimique de la paroi de Clostridium tyrobutyricum
Mars 1984-Juin1986 Institut de Biologie Moléculaire et Cellulaire, univ Osaka, JAPON (Y Okada) Boursière du groupe pharmaceutique Takeda
Construction d’hybrides somatiques monochromosomiques hommexsouris Avril-Juillet 1987 Laboratoire de Structure et Mutagénèse Chromosomiques,
Institut Curie, Paris (B Dutrillaux)
Formation aux techniques d’analyse chromosomique en bandes R 1988-1993 Laboratoire de Cytogénétique, INRA Jouy-en-Josas (P Popescu)
Laboratoire de Structure et Mutagénèse Chromosomiques, I.Curie (B Dutrillaux) Analyse caryotypique et cartographie cytogénétique comparées chez les trois principaux bovidés domestiques : le bœuf (Bos taurus L.), le mouton (Ovis aries L.) et la chèvre (Capra hircus L.)
Carrière professionnelle
Juillet 1986 Recrutement au titre de chargée de recherches 2ème classe à l’INRA Laboratoire de Cytogénétique, INRA Jouy-en-Josas
Janvier 1991 Chargée de recherches 1ère classe INRA
Depuis 2002 Responsable de l’activité Cytogénétique dans l’équipe Génomique des bovins (A Eggen) de l’unité de Génétique biochimique et Cytogénétique (E Cribiu)
TABLE DES MATIERES
Première partie:
Renseignements administratifs et présentation
de la candidate ... 4
1. Lettre de sollicitation et de proposition des rapporteurs ... 4
2. Fiche récapitulative de demande d’HDR ... 5
3. Curriculum vitae ... 6
4. Itinéraire en recherche ... 7
5. Liste des publications ... 8
5.1. Publications scientifiques ... 8
5.1.1. Internationales avec comité de lecture ... 8
5.1.2. Nationales avec comité de lecture ... 12
5.1.3. Didactiques et non référencées ... 12
5.1.4. Chapitres de livres et livres ... 13
5.2. Listes des communications ... 13
5.2.1. Conférences sur invitation ... 13
5.2.2. Communications orales à des colloques ... 13
5.2.3. Communications par affiches à des colloques ... 14
6. Photocopie des diplômes ... 17
7. Liste des étudiants co-encadrés en thèse ... 18
8. Abréviations ... 19
Deuxième partie:
Synthèse des recherches sur le caryotype et la
carte cytogénétique d’animaux d’élevage ... 20
1. Introduction ... 20
2. Le caryotype : constante caractéristique d’un individu ou d’une espèce .... 20
2.1. De l’individu aux chromosomes ... 20
2.2. Du choix des bandes R pour identifier les chromosomes (1986-1988) ... 21
2.3. De la nécessité des caryotypes de référence (ISCNDA89, ISCNDB2000, lapin) .... 22
3. La carte cytogénétique : arrangement linéaire des loci sur les
chromosomes ... 24
3.1. Objectifs de la cartographie de gènes ... 24
3.2. Construction d’une carte cytogénétique ... 24
3.2.2. Hybridation in situ : « l’école française de B. Dutrillaux et son équipe 1989-1990 » . 27
3.3. Les progrès de la carte cytogénétique de la vache (1991-2000) ... 28
3.3.1. Contribution à la carte cytogénétique bovine ... 28
3.3.2. Intégration des cartes génétiques et cytogénétique de BTA1 ... 29
3.4. Première carte intégrée génétique et cytogénétique du lapin (2001-2005) ... 29
4. Cartographie comparée: intégration des connaissances d’espèces
différentes ... 31
4.1. Comparaison des caryotypes de ruminants domestiques et sauvages ... 31
4.1.1. Très grande similitude des caryotypes bovin, ovin et caprin ... 31
4.1.2 Le caryotype bovin comme référence pour la cytogénétique des ruminants sauvages 31 4.2. Comparaison des cartes humaine et animales ... 32
4.2.1. Peinture chromosomique hétérologue : vache/homme (1995) ... 33
4.2.2. Alignement des cartes cytogénétiques : homme/vache (2001-2003) ... 34
4.2.3. Carte comparée homme - lapin ... 35
5. Conclusion générale : du séquençage de génomes entiers ... 36
6. Références ... 37
Troisième partie :
Conception, animation, direction et
coordination des activités de recherche ... 40
1. Conception des activités de recherche ... 40
2. Animation des activités de recherche ... 41
3. Direction des activités de recherche ... 42
4. Coordination des activités de recherche ... 43
Quatrième partie :
Projet de recherche sur les régions
centromériques ... 45
1. Contexte du projet proposé ... 45
2. La région centromérique des chromosomes eucaryotes ... 47
2.1. Définitions du centromère ... 47
2.2. Rôle et fonctions de la région centromérique ... 48
2.3. Organisation de la région centromérique ... 49
2.3.1. Domaine central ... 49
2.3.2. Domaine kinétochorien ... 50
2.3.3. Domaine péricentromérique ... 51
3.1. Introduction
………….
... 513.2. ADN centromérique et fonction centromérique ... 51
3.3. Isolement et caractérisation de l’ADN centromérique ... 53
3.4. ADN satellite et évolution ... 54
3.5. ADN satellites humains ... 55
3.5.1. Différentes classes d’ADN satellite humain ... 55
3.5.2. ADN satellite alpha ... 55
3.5.3. ADN centromérique et détection individuelle des chromosomes humains ... 56
3.6. ADN satellites bovins ... 57
3.7. ADN satellites porcins ... 60
4. Présentation du projet ... 61
4.1. Approches méthodologiques envisagées pour les chromosomes bovins... 62
4.1.1. Analyse fine du clone de BAC bovin 231G08 ... 62
4.1.2. Isolement de séquences centromériques bovines connues ... 63
4.2. Approches méthodologiques envisagées pour les chromosomes porcins ... 64
4.3. Mise au point d’amorces centromériques spécifiques de chromosomes ... 64
5. Conclusion et perspectives ... 65
Première partie: Renseignements administratifs et présentation de
la candidate
1. Lettre de sollicitation et de proposition des rapporteurs
Hélène Hayes Commission HDR
LGbC, INRA-CRJ UFR Médecine/Biologie de l’UVSQ
78352 Jouy-en-Josas cedex CNU 4704 Génétique
hélène.hayes@jouy.inra.fr Tél. 01 34 65 26 73
Jouy-en-Josas le 21 octobre 2005 Mesdames, Messieurs,
Je soussignée Hélène HAYES, chargée de recherches 1ère classe à l’unité de Génétique biochimique et Cytogénétique de l’INRA Jouy-en-Josas, pose ma candidature à l’habilitation à diriger des recherches à l’UFR Médecine/Biologie de l’université de Versailles St Quentin dans la CNU de Génétique 4704.
Le document ci-joint comporte quatre parties :
- une première partie comportant la lettre de sollicitation et de proposition des rapporteurs, la fiche de demande d’HDR de médecine/Biologie à l’UVSQ, un curriculum vitae et mon itinéraire en recherche, la liste de mes publications, la photocopie du diplôme de thèse et la liste des étudiants encadrés
- une deuxième partie sur la synthèse de mes travaux de recherche
- une troisième partie sur la conception, l’animation, la direction et la coordination de mes activités de recherche
- une quatrième partie sur mon projet de recherche.
La liste des rapporteurs extérieurs proposés, ayant donné leur accord de principe, est la suivante :
- Professor Michael Schmid, Department of Human genetics, University of Würzburg, Biozentrum, Am Hubland, D-97074 Würzburg, ALLEMAGNE.
- Dr Bernard Dutrillaux, Laboratoire de Cytogénétique Animale, Muséum National d’Histoire Naturelle, Bâtiment de phanérogamie, 16 rue Buffon, 75005 Paris.
- Dr Françoise Lemeunier, Laboratoire Populations, Génétique et Evolution, CNRS, Bâtiment 13, avenue de la Terrasse, 91198 Gif-sur-Yvette.
- Dr Martine Yerle, Laboratoire de Génétique Cellulaire, INRA, BP 27, 31326 Castanet-Tolosan.
J’ai préparé une version en anglais (couverture verte) afin que le Dr M. Schmid puisse réaliser son évaluation.
Dans l’attente de votre réponse, veuillez Mesdames, Messieurs, accepter mes sincères salutations.
2. Fiche récapitulative de demande d’HDR
FICHE RECAPITULATIVE DE DEMANDE D’HABILITATION A DIRIGER DES
RECHERCHES
Nom :
HAYES
... Prénom :
Hélène
...
Date de naissance
:20 août 1957 UFR ou faculté : Médecine/Biologie CNU 4704 ....
Diplômes et titres acquis
Juin 1976 : Baccalauréat série C, Paris
Juin 1978 : DEUG Sciences de la Nature et de la Vie, université Paris VI Juin 1980 : Maîtrise de Biochimie, université Paris VI
Juin 1981 : DEA Biochimie Industrielle et Alimentaire, université Paris VII
Mars 1984 : Thèse de troisième cycle de Sciences Alimentaires, université Paris VII Juin 1987 : Maîtrise de Biologie et Génétique Appliquée, université Paris VII
Octobre 1993 : Thèse de Doctorat de Biodiversité, Génétique et Evolution, université Paris VII
Situation Professionnelle actuelle
Chargée de recherches 1ère classe
Laboratoire de rattachement
Unité de Génétique biochimique et Cytogénétique, UR 339, INRA-CRJ, Jouy-en-Josas 78350
Résumé du programme de recherche
De 1986 à 2005, mes travaux de recherche ont porté sur les caryotypes et les cartes cytogénétiques de la vache puis du lapin et sur leur comparaison avec ceux d’autres espèces domestiques, sauvages et humaine. La localisation de nombreux gènes pour la construction de cartes chromosomiques de ces espèces a permis d’intégrer les cartes génétiques et cytogénétiques, d’établir les liens entre données phénotypiques et
génotypiques et d’exploiter les données des cartes humaine et murine, beaucoup plus riches, dans les projets d’identification de gènes d’intérêt et de QTL. Avec l’avènement du séquençage global des génomes (bovin en 2006), la cartographie par hybridation in situ devient moins utile. Un nouveau projet est proposé sur
l’analyse des séquences d’ADN centromérique bovin et porcin afin d’une part, de pouvoir exploiter leur polymorphisme pour construire un panel de sondes centromériques spécifiques de chromosomes et d’autre part, d’améliorer la connaissance de ces séquences encore peu développée chez ces espèces pour à long terme développer des chromosomes artificiels, intéressant la transgénèse et l’étude du noyau en interphase.
Sélection des cinq publications les plus significatives
ISCNDA 1989: International System for Cytogenetic Nomenclature of Domestic Animals, DI BERARDINO D., HAYES H., FRIES R., LONG S. (eds). Cytogenet Cell Genet (1990) 53, 65-79.
HAYES H., PETIT E., LEMIEUX N., DUTRILLAUX B. Chromosomal localization of the ovine beta-casein gene by non-isotopic in situ hybridization and R-banding. Cytogenet Cell Genet (1992) 61, 286-288.
HAYES H. Chromosome painting with human chromosome specific DNA libraries reveals the extent and distribution of conserved segments in bovine chromosomes. Cytogenet Cell Genet (1995) 71, 168-174. HAYES H., ELDUQUE C., GAUTIER M., SCHIBLER L., CRIBIU E., EGGEN A. Mapping of 195 genes in cattle, and updated comparative map with man, mouse, rat and pig. Cytogenet Genome Res (2003) 102, 16-24.
CHANTRY-DARMON C., BERTAUD M., URIEN C., CHADI-TAOURIT S., PERROCHEAU M., ROGEL-GAILLARD C., HAYES H. Expanded comparative mapping between man and rabbit and detection of a new conserved segment between HSA22 and OCU4. Cytogenet Genome Res (2005) 111, 134-139.
3. Curriculum vitae
Hélène HAYES
Née le 20 août 1957Nationalité: française Unité de Génétique biochimique et Cytogénétique, INRA-CRJ
78352 Jouy-en-Josas Cedex Tél. 01 34 65 26 73 Fax 01 34 65 24 78
Email helene.hayes@jouy.inra.fr
Diplômes
Juin 1976 Baccalauréat C, Paris
Juin 1978 DEUG Sciences de la Nature et de la Vie, univ Paris VI Juin 1980 Maîtrise de Biochimie, université Paris VI
Juin 1981 DEA Biochimie Industrielle et Alimentaire, univ Paris VII
Mars 1984 Thèse de troisième cycle de Sciences Alimentaires, univ Paris VII Juin 1987 Maîtrise de Biologie et Génétique Appliquée, univ Paris VII
Octobre 1993 Thèse de doctorat de Biodiversité, Génétique et Evolution, univ Paris VII
Stages et formations complémentaires
Janvier 1980 Laboratoire de Chimie Organique Biologique, univ Paris VI (A Marquet) Essais de synthèse de l’acide chrysanthémique
Janvier-juin 1981 Laboratoire de Technologie Alimentaire, INRA Massy (MT Tollier) Composition en sucres de l’orge selon les conditions de croissance
Juillet-août 1981 Institute of Cancer Research, Columbia University, New York, USA (P Feigelson) Essais de clonage du gène de la tryptophane oxygenase hépatique de rat Septembre 1981- Laboratoire de Microbiologie Laitière, INRA Jouy-en-Josas (JL Bergère) Mars 1984 Allocataire du Ministère de la Recherche et de l’Industrie
Etude biochimique de la paroi de Clostridium tyrobutyricum
Mars 1984- Institut de Biologie Moléculaire et Cellulaire, univ Osaka, JAPON (Y Okada) Juin 1986 Boursière du groupe pharmaceutique Takeda
Construction d’hybrides somatiques monochromosomiques homme X souris Avril-juillet 1987 Laboratoire de Structure et Mutagenèse Chromosomiques
Institut Curie, Paris (B Dutrillaux)
Formation aux techniques d’analyse chromosomique en bandes R 1988-1993 Laboratoire de Cytogénétique, INRA Jouy-en-Josas (P Popescu)
Laboratoire de Structure et Mutagenèse Chromosomiques, I.Curie (B Dutrillaux) Analyse caryotypique et cartographie cytogénétique comparées chez les trois principaux bovidés domestiques : le bœuf (Bos taurus L.), le mouton (Ovis aries L.) et la chèvre (Capra hircus L.)
Carrière professionnelle
Juillet 1986 Recrutement au titre de chargée de recherches 2ème classe à l’INRA Laboratoire de Cytogénétique, INRA Jouy-en-Josas
Janvier 1991 Chargée de recherches 1ère classe INRA
Depuis 2002 Responsable de l’activité Cytogénétique dans l’équipe Génomique des bovins (A Eggen) de l’unité de Génétique biochimique et Cytogénétique (E Cribiu)
4. Itinéraire en recherche
Mes études universitaires ont été consacrées à deux disciplines principalement, la biochimie et la chimie organique et ont débouché sur un doctorat de troisième cycle en Sciences alimentaires à l’université Paris VII. Ce doctorat effectué dans le laboratoire de Microbiologie Laitière (J-L. Bergère) de l’INRA à Jouy-en-Josas (septembre 1981 - mars 1984) a porté sur la caractérisation biochimique de la paroi de Clostridium tyrobutyricum. Cette bactérie, présente dans certains lots de lait, est responsable du gonflement butyrique des fromages à pâte cuite entraînant un goût et une odeur désagréables et donc des pertes financières dans l’industrie fromagère. Ce sujet s’inscrivait dans un programme visant à mettre au point un test de détection spécifique de C. tyrobutyricum dans le lait.
Après la soutenance de mon doctorat en 1984, j’ai décidé de partir en stage post-doctoral à l’étranger. Ayant obtenu une bourse de la fondation japonaise Takeda, j’ai pu me rendre d’avril 1984 à juin 1986 à l’Institut de Biologie Moléculaire et Cellulaire de l’université d’Osaka (Japon). Le sujet de mon activité de recherche s’en est trouvé profondément modifié puisque le directeur de mon laboratoire d’accueil, Y. Okada, m’a proposé de travailler sur la mise au point d’un système de sélection positive pour la construction d’hybrides somatiques homme X souris par fusion entre cellules entières et fusion microcellulaire. Ce travail sera présenté brièvement dans la deuxième partie de ce mémoire. Mon stage post-doctoral a été une expérience scientifique et intellectuelle inoubliable. En effet, j’ai eu la chance d’être accueillie par une équipe de chercheurs et d’étudiants motivés et toujours disponibles pour répondre à toutes mes interrogations sur la science et sur le Japon.
Cette expérience japonaise a déterminé la suite de ma vie professionnelle après mon retour en France. C’est au cours de mon séjour au Japon que j’ai découvert la biologie eucaryote et la cytogénétique. Grâce à des contacts avec P. Popescu, directeur du laboratoire de Cytogénétique à l’INRA de Jouy-en-Josas, qui lors d’une visite au Japon m’a informée de l’ouverture d’un poste de chargée de recherche dans son laboratoire, j’ai décidé de me porter candidate au concours pour le profil « Cytogénétique des animaux d’élevage ». Reçue au concours en juin 1986 mais n’ayant pas de diplôme en génétique et sur les conseils du jury de recrutement, j’ai d’abord suivi la maîtrise de Biologie et Génétique Appliquée de l’université de Paris VII avant de rejoindre en juillet 1987, le laboratoire de P. Popescu.
Le travail de recherche que je devais mener au laboratoire de P.Popescu concernait l’analyse caryotypique et la cartographie génique comparées chez les trois principaux bovidés domestiques : le bœuf, le mouton et la chèvre. Pour répondre aux besoins de ces recherches, j’ai acquis des techniques de cytogénétique développées dans le laboratoire de Structure et Mutagenèse Chromosomiques de B. Dutrillaux (Institut Curie à Paris). Afin d’avoir un statut officiel durant mes stages de formation dans ce laboratoire, je me suis inscrite au diplôme de doctorat de Biodiversité Génétique et Evolution à l’université de Paris VII sous la direction scientifique de B. Dutrillaux. Après mon doctorat soutenu en octobre 1993, j’ai poursuivi mes activités de recherche en cytogénétique animale dans le laboratoire de Cytogénétique de P. Popescu devenu le laboratoire de Génétique biochimique et Cytogénétique en 1993 et dirigé aujourd’hui par E. Cribiu.
5. Liste des publications
5.1. Publications scientifiques
5.1.1. Internationales avec comité de lecture
HAYES H., COMMISSAIRE J., BERGERE J-L., 1984. A high-molecular-mass cell wall protein released from Clostridium tyrobutyricum by heat treatment. FEBS Letters 174, 284-288.
BERGERE J-L., HAYES H., COMMISSAIRE J., 1986. Major protein components in the cell envelope of Clostridium tyrobutyricum. Ann Inst Pasteur/Microbiol 137B, 271-282.
KANEDA Y., HAYES H., UCHIDA T., YOSHIDA M.C., OKADA Y., 1987. Regional assignment of five genes on human chromosome 19. Chromosoma 95, 8-12.
HAYES H., KANEDA Y., UCHIDA T., OKADA Y., 1987. Regional assignment of the gene for diphtheria toxin sensitivity using subchromosomal fragments in microcell hybrids. Chromosoma 96, 26-32. KOHNO K., HAYES H., MEKADA E., UCHIDA T., 1987. Mutant with diphtheria toxin receptor and
acidification function but defective in entry of toxin. Exp Cell Res 172, 54-64.
ISCNDA 1989: International System for Cytogenetic Nomenclature of Domestic Animals, DI BERARDINO D., HAYES H., FRIES R., LONG S. (eds), 1990. Cytogenet Cell Genet 53, 65-79.
YONEDA A., YONEDA Y., KANEDA Y., HAYES H., UCHIDA T., OKADA Y., 1991. Monoclonal antibodies specific for human chromosome 5 obtained with a monochromosomal hybrid can be used to sort out cells containing the chromosome with a FACS. Chromosoma 100, 187-192.
HAYES H., PETIT E., DUTRILLAUX B., 1991. Comparison of RBG-banded karyotypes of cattle, sheep and goats. Cytogenet Cell Genet 57, 51-55.
HAYES H., PETIT E., LEMIEUX N., DUTRILLAUX B., 1992. Chromosomal localization of the ovine beta-casein gene by non-isotopic in situ hybridization and R-banding. Cytogenet Cell Genet 61, 286-288.
HAYES H., PETIT E., BOUNIOL C., POPESCU P., 1993. Localization of the alpha-S2-casein gene (CASAS2) to the homoeologous cattle, sheep and goat chromosomes 4 by in situ hybridization. Cytogenet Cell Genet 64, 281-285.
HAYES H., PETIT E., 1993. Mapping of the beta-lactoglobulin gene and of an immunoglobulin M heavy chain-like sequence to homoeologous cattle, sheep and goat chromosomes. Mammal Genome 4, 207-210.
HAYES H., POPESCU P., DUTRILLAUX B., 1993. Comparative gene mapping of lactoperoxidase, retinoblastoma, and alpha-lactalbumin genes in cattle, sheep and goats. Mammalian Genome 4, 593-597.
CLARO F., HAYES H., CRIBIU E., 1993. The R- and G-banded karyotypes of the sable antelope (Hippotragus niger). J Hered 84, 481-484.
CLARO F., HAYES H., CRIBIU E., 1994. The C-, G-, and R-banded karyotype of the scimitar-horned oryx (Oryx dammah). Hereditas 120, 1-6.
VASSART M., SEGUELA A., HAYES H., 1995. Chromosomal evolution in gazelles. J Hered 86, 216-227. POPESCU P., BOSCHER J., HAYES H., BAN J., KETTMANN R., 1995. Chromosomal localization of the
LE CHALONY C., PIBOUIN L., HAYES H., APIOU F., DUTRILLAUX B., GOUBIN G., 1995. Partial nucleotide sequence and chromosomal localization of a bovine zinc finger gene ZNF164. Cytogenet Cell Genet 70, 192-194.
CLARO F., HAYES H., CRIBIU E., 1995. Identification of p and q arms of the blesbok (Damaliscus dorcas phillipsi, Alcelaphinae) RBG-banded chromosomes with comparison to other wild and domestic animals. Cytogenet Cell Genet 70, 268-272.
HAYES H., 1995. Chromosome painting with human chromosome specific DNA libraries reveals the extent and distribution of conserved segments in bovine chromosomes. Cytogenet Cell Genet 71, 168-174.
CLARO F., HAYES H., CRIBIU E., 1996. The karyotype of the addax and its comparison with karyotypes of other species of Hippotraginae antelopes. Hereditas 124, 223-227.
PFISTER-GENSKOW M., HAYES H., EGGEN A., BISHOP M., 1996. Chromosomal localization of the bovine obesity (OBS) gene. Mammalian Genome 7, 398-399.
HAWKINS G.A., EGGEN A., HAYES H., ELDUQUE C., BISHOP M., 1996. Tyrosinase-related protein-2 (DCT; TYRP2) maps to bovine chromosome 12. Mammalian Genome 7, 474.
PFISTER-GENSKOW M., HAYES H., EGGEN A., BISHOP M., 1996. The leptin receptor (LEPR) gene maps to bovine chromosome 3q33. Mammal Genome 8, 227.
ELDUQUE C., LAURENT P., HAYES H., RODELLAR C., LEVEZIEL H., ZARAGOZA P., 1996. PCR amplification and localization by FISH of the NGFB gene in cattle. Archivos de Zootechnia 45, 355-359.
FOLCH J.M., COLL A., HAYES H., SANCHEZ A., 1996. Characterization of a caprine beta-lactoglobulin pseudogene, identification and chromosomal localization by in situ hybridization in goat, sheep and cow. Gene 177, 87-91.
HAYES H., LE CHALONY C., GOUBIN G., MERCIER D., PAYEN E., BIGNON C., KOHNO K., 1996. Localization of ZNF164, ZNF146, GGTA1, SOX2, PRLR and EEF2 on homoeologous cattle, sheep and goat chromosomes by in situ hybridization and comparison with the human gene map. Cytogenet Cell Genet 72, 342-346.
ELDUQUE C., LAURENT P., HAYES H., RODELLAR C., LEVEZIEL H., ZARAGOZA P., 1997. Assignment of the beta-nerve growth factor (NGFB) to bovine chromosome 3 band q23 by in situ hybridization. Cytogenet Cell Genet 77, 306-307.
PAYEN E., PAILHOUX E., GIANQUINTO L., HAYES H., LE PENNEC N., BEZARD J., COTINOT C., 1997. The ovine SOX-2 gene: sequence, localization and gonadal expression. Gene 189, 143-147. SUN H.S., CAI L., DAVIS S.K., TAYLOR J.F., DOUD L.K., BISHOP M., HAYES H., BARENDSE W., VAIMAN
D., McGRAW R.A., HIRANO T., SUGIMOTO Y., KIRKPATRICK B.W., 1997. Comparative mapping of human chromosome 13 and bovine chromosome 12. Genomics 39, 47-54.
METENIER-DELISSE L., HAYES H., LEROUX C., GIRAUD-DELVILLE C., LEVEZIEL H., GUERIN G., MARTIN P., GROSCLAUDE F., 1997. Isolation and molecular characterization of bovine Rhesus-like transcripts and chromosome mapping of the relevant locus. Animal Genet 28, 202-209.
POPESCU C.P., HAYES H., MEINIEL R., CREVEAUX I., MEINIEL A., 1997. Localization of the SCO-spondin gene to cattle chromosome 4. Chromos Res 5, 276-277.
IANNUZZI L., HAYES H., DI MEO G.P., MERCIER D., KOHNO K., 1997. Chromosomal localization of alpha-galactosyltransferase 1 (GGTA1) and elongation factor 2 (EEF2) genes in river buffalo by FISH. Chromos Res 5, 274-277.
ROGEL-GAILLARD C., HAYES H., COULLIN P., CHARDON P., VAIMAN M., 1997. Swine centromeric DNA repeats revealed by primed in situ (PRINS) labelling. Cytogenet Cell Genet 79, 79-84.
LARSEN N.J., EGGEN A., HAYES H., BYLA B., DOUD L., BISHOP M.D., KIRKPATRICK B.W., 1997. UW54 and UW63, two polymorphic bovine microsatellites on chromosomes 6 and 8q18, respectively. Animal Genet 28, 377-378.
PFISTER-GENSKOW M., WEESNER G.D., HAYES H., EGGEN A., BISHOP M.D., 1997. Physical and genetic localization of the bovine cannabinoid receptor (CNR1) gene to bovine chromosome 9. Mammal Genome, 8, 301-302.
ELDUQUE C., HAYES H., BISHOP M., EGGEN A., 1998. Assignment of the presenilin 2 gene (PSEN2) to bovine chromosome 16. Animal Genet 29, 465-466.
FAURE M., HAYES H., BLEDSOE R.K., HUTSON S.M., PAPET I., 1998. Assignment of the gene of mitochondrial branched chain aminotransferase (BCAT2) to sheep chromosome band 14q24 and to cattle and goat chromosome bands 18q24 by in situ hybridization. Cytogenet Cell Genet 83, 96-97.
LAURENT P., HAYES H., PETIT E., LEVEZIEL H., 1999. PL44, a cosmid containing microsatellite INRA242 (DXS39), maps to bovine chromosome Xq25. Animal Genet 30,161.
LARSEN N.J., HAYES H., BISHOP M., DAVIS S.K., TAYLOR J., KIRKPATRICK B.W., 1999. A comparative linkage and physical map of bovine chromosome 24 with human chromosome 18. Mammal Genome 10, 482-487.
LAURENT P., HAYES H., EGGEN A., BISHOP M., LEVEZIEL H., 1999. Assignment of the amylase gene cluster to bovine chromosome 3 bands 3q23-q24 by in situ hybridization. Cytogenet Cell Genet 85, 315-316.
LE CHALONY C., HAYES H., FRELAT G., GEFFROTIN C., 2000. Identification and mapping of swine cyclin-dependent kinase inhibitor CDKN2A and CDKN2B exon 2 sequences. Cytogenet Cell Genet 88, 240-243.
HAYES H., BONFILS J., FAURE M., PAPET I., 2000. Assignment of BCAT1, the gene encoding cytosolic branched chain aminotransferase, to sheep chromosome band 3q33 and to cattle and goat chromosome bands 5q33 by in situ hybridization. Cytogenet Cell Genet 90, 84-85.
LAURENT P., ELDUQUE C., HAYES H., SAUNIER K., EGGEN A., LEVEZIEL H., 2000. Assignment of 60 human ESTs in cattle. Mammal Genome 11, 748-754.
HAYES H., DI MEO G.P., GAUTIER M., LAURENT P., EGGEN A., IANNUZZI L., 2000. Localization by FISH of the 31 Texas nomenclature type I markers to both Q- and R-banded bovine chromosomes. Cytogenet Cell Genet 90, 315-320.
IANNUZZI L., DI MEO G.P., HAYES H., PERUCATTI A., INCARNATO D., GAUTIER M., EGGEN A., 2001. FISH-mapping of the 31 Texas markers to river buffalo chromosomes. Chromos Res 9, 339-342. GAUTIER M., LAURENT P., HAYES H., EGGEN A., 2001. Development and assignment of bovine
specific PCR-system for the Texas nomenclature marker genes and isolation of homologous BAC probes. Genet Sel Evol 33, 191-200.
ISCNDB (2000) International System for Chromosome Nomenclature of Domestic Bovids. DI BERARDINO D., DI MEO G.P., GALLAGHER D.S., HAYES H., IANNUZZI I. (eds), 2001. Cytogenet Cell Genet 92, 283-299.
GAUTIER M., HAYES H., TAOURIT S., LAURENT P., EGGEN A., 2001. Assignment of eight additional genes from human chromosome 11 to bovine chromosome 15 and 29: refinement of the comparative map. Cytogenet Cell Genet 93, 60-64.
EGGEN A., GAUTIER M., BILLAUT A., PETIT E., HAYES H., LAURENT P., SORAVITO C., PFISTER-GENSKOW M., EILERTSEN K., BISHOP M.D., 2001. Construction and characterization of a new bovine BAC library with four genome-equivalent coverage. Genet Sel Evol 33, 543-548.
BERNARD L., LEROUX C., HAYES H., GAUTIER M., CHILLIARD Y., MARTIN P., 2001. Genomic organization, chromosomal localization and complete 5 kb cDNA sequence of the caprine SCD gene. Gene 281, 53-61.
GAUTIER M., HAYES H., TAOURIT S., LAURENT P., EGGEN A., 2001. Syntenic assignment of sixteen genes from the long arm of human chromosome 10 to bovine chromosomes 26 and 28: refinement of the comparative map. Chromos Res 9, 617-621.
ROY R., GAUTIER M., HAYES H., LAURENT P., OSTA R., ZARAGOZA P., EGGEN A., RODELLAR C., 2001. Assignment of the fatty acid synthase (FASN) gene to bovine chromosome 19 (19q22) by in situ hybridization and confirmation by somatic cell hybrid mapping. Cytogenet Cell Genet. 93, 141-142.
BONNET A., THEVENON S., CLARO F., GAUTIER M., HAYES H., 2001. Cytogenetic comparison between Vietnamese sika deer and cattle: R-banded karyotypes and FISH mapping. Chromos Res 9,673-687.
ROGEL-GAILLARD C., HAYES H., BOURGEAUX N., CHARDON P., 2001. Assignment of two new loci for gamma 1 porcine endogenous retroviruses (gamma 1 PERV) to pig chromosome bands 2q21 and 11q12 by in situ hybridization. Cytogenet Cell Genet 95, 112-113.
PAULOIN A., ROGEL-GAILLARD C., PIUMI F., HAYES H., FONTAINE M.L., CHANAT E., CHARDON P., DEVINOY E., 2002. Structure of the rabbit alpha S1 and beta casein gene cluster, assignment to chromosome 15 and expression of the alpha s1 casein gene in HC11 cells. Gene 283, 155-162. GAUTIER M., HAYES H., EGGEN A., 2002. An extensive and comprehensive radiation hybrid map of
bovine chromosome 15: comparison with human chromosome 11. Mammal Genome 13, 316-319. HAYES H., ROGEL-GAILLARD C., ZIJLSTRA C., DE HAAN N.A., URIEN C., BOURGEAUX N., BERTAUD M.,
BOSMA A.A., 2002. Establishment of an R-banded rabbit karyotype nomenclature by FISH localization of 23 chromosome-specific genes on both G- and R-banded chromosomes. Cytogenet Genome Res 98, 199-205.
ROY R., GAUTIER M., HAYES H., LAURENT P., ZARAGOZA P., EGGEN A., RODELLAR C., 2002. Assignment of mitochondrial glycerol-3-phosphate acyltransferase (GPAM) gene to bovine chromosome 26 (26q22) by in situ hybridization and confirmation by somatic cell hybrid mapping. Cytogenet Genome Res 97, 276.
ROY R., GAUTIER M., HAYES H., LAURENT P., ZARAGOZA P., EGGEN A., RODELLAR C., 2002. Assignment of maltase glucoamylase (MGAM) gene to bovine chromosome 4q34 by in situ hybridization and confirmation by radiation hybrid mapping. Cytogenet Genome Res 98, 311. DI MEO G.P., PERUCATTI A., GAUTIER M., HAYES H., INCARNATO D., EGGEN A., IANNUZZI L., 2003.
Chromosome localization of the 31 type 1 Texas bovine markers in sheep and goat chromosomes by comparative FISH mapping and R-banding. Anim Genet 34, 294-296.
ROY R., GAUTIER M., HAYES H., ZARAGOZA P., EGGEN A., RODELLAR C., 2003. Assignment of monoglyceride lipase (MGLL) gene to bovine chromosome 22q24 by in situ hybridization and confirmation by radiation hybrid mapping. Cytogenet Genome Res 101, 92.
ROY R., CALVO J.H., HAYES H., RODELLAR C., EGGEN A., 2003. Fine mapping of the bovine heart fatty acid-binding protein gene (FABP3) to BTA2q45 by fluorescence in situ hybridization and radiation hybrid mapping. Anim Genet 34, 466-467.
LE PROVOST F., CASSY S., HAYES H., MARTIN P., 2003. Structure and expression of goat GLYCAM1 gene: lactogenic-dependent expression in ruminant mammary gland and interspecies conservation of the proximal promoter. Gene 313, 83-89.
GAUTIER M., HAYES H., EGGEN A., 2003. A comprehensive radiation hybrid map of bovine Chromosome 26 (BTA26): comparative chromosomal organization between HSA10q and BTA26 and BTA28. Mamm Genome 14, 711-721.
BONNET-GARNIER A., CLARO F., THEVENON S., GAUTIER M., HAYES H., 2003. Identification by R-banding and FISH of chromosome arms involved in Robertsonian translocations in several deer species. Chromosome Res 11, 649-663.
HAYES H., ELDUQUE C., GAUTIER M., SCHIBLER L., CRIBIU E., EGGEN A., 2003. Mapping of 195 genes in cattle, and updated comparative map with man, mouse, rat and pig. Cytogenet Genome Res 102, 16-24.
GAUTIER M., HAYES H., BONSDORFF T., EGGEN A., 2003. Development of a comprehensive comparative radiation hybrid map of bovine chromosomes 7 (BTA7) versus human chromosomes 1 (HSA1), 5 (HSA5) and 19 (HSA19). Cytogenet Genome Res 102, 25-31.
CHANTRY-DARMON C., ROGEL-GAILLARD C., BERTAUD M., URIEN C., PERROCHEAU M., CHARDON P., HAYES H., 2003. 133 new gene localizations on the rabbit cytogenetic map. Cytogenet Genome Res 103, 192-201.
ORDOVAS L., ROY R., ZARAGOZA P., HAYES H., EGGEN A., RODELLAR C., 2005. Assignment of the solute carrier family 27 member 1 (SLC27A1) gene to bovine chromosome 17. Animal Genet 36(4): 352-353.
CHANTRY-DARMON C., BERTAUD M., URIEN C., CHADI-TAOURIT S., PERROCHEAU M., ROGEL-GAILLARD C., HAYES H., 2005. Expanded comparative mapping between man and rabbit and detection of a new conserved segment between HSA22 and OCU4. Cytogenet Genome Res 111:134-139.
POUSSIN K., HAYES H., PAULOIN A., CHANAT E., FONTAINE M-L., AUJEAN E., SUN J-S., DEBEY P., DEVINOY E., 2005. Interactions between the rabbit CSN1 gene and the nuclear matrix of stably transfected HC11 mammary epithelial cells vary with its level of expression. J Cell Biochem 96:611-621.
CHANTRY-DARMON C., URIEN C., HAYES H., BERTAUD M., CHADI-TAOURIT S., CHARDON P., VAIMAN D., ROGEL-GAILLARD C., 2005. Construction of a cytogenetically anchored microsatellite map in rabbit. Mamm Genome 16:442-459.
HAJJOUBI S., RIVAL-GERVIER S., HAYES H., FLORIOT S., EGGEN A., PIUMI F., CHARDON P., HOUDEBINE L-M., THÉPOT D., 2005. Ruminants’ genome no longer contains Whey Acidic Protein gene but only a pseudogene. Gene (sous presse).
5.1.2. Nationales avec comité de lecture
HAYES H., PETIT E., 1991. Comparison of RBG-banded karyotypes of cattle, sheep and goat. Genet Sel Evol 23, 144s-s147s.
HAYES H., 2000. ADN et chromosomes. Prod Anim hors série « Génétique moléculaire » 13-20.
5.1.3. Didactiques et non référencées
HAYES H., 1995. Peindre les chromosomes. JIR 93.
HAYES H., 1996. Peindre les chromosomes: Une nouvelle technique pour identifier les segments chromosomiques conservés entre espèces. INRA mensuel 87, 2-3.
5.1.4. Chapitres de livres et livres
BROAD T.E., HAYES H., LONG S., 1997. Cytogenetics: Physical chromosome maps in “The Genetics of Sheep” éditeurs L Piper et A Ruvinsky. CAB INTERNATIONAL (GB).
POPESCU P., HAYES H., DUTRILLAUX B., 1998. Techniques de Cytogénétique Animale. Editions INRA dans la collection « Techniques et Pratiques ».
POPESCU P., HAYES H., DUTRILLAUX B., 2000. Techniques in Animal Cytogenetics. Springer/INRA dans la collection « Principles and Practise ».
5.2. Listes des communications 5.2.1. Conférences sur invitation
HAYES H., 1993. Chromosomal and gene map comparisons between man and cattle. International Workshop on Animal Genome Analysis, Tsukuba, JAPON.
HAYES H., 1996. Hybridation in situ et génome bovin. Laboratoire de Génétique Moléculaire et cellulaire, INRA. Grignon, FRANCE.
HAYES H., 2000. ADN et chromosomes. Séminaire du département de Génétique Animale de l’INRA »La génétique moléculaire et ses applications ». Cap d’Agde, FRANCE.
HAYES H., 2002. Cartographie chromosomique du génome bovin : outils, résultats, applications. Colloque du Muséum National d’Histoire Naturelle « Contribution de la cytogénétique moderne à la biologie évolutive ». Paris, France.
HAYES H., CHANTRY-DARMON C., ALLAIN D., PENA B., URIEN C., BERTAUD M., DE ROCHAMBEAU H., ROGEL-GAILLARD C., 2004. Use of FISH-mapped genes to construct an integrated genetic map in rabbit. 15th International Chromosome Conference, Brunel, GRANDE BRETAGNE.
5.2.2. Communications orales à des colloques
HAYES H., KANEDA Y., UCHIDA T., OKADA Y., 1985. Isolation of mouse x human hybrid cells containing human chromosomes having integrated a dominant selectable marker and karyotype analysis. 38th Japanese Congress in Cellular Biology, Hiroshima, JAPON.
HAYES H., MATEJKA M., CRIBIU EP, 1988. Comparison of diagrammatic representations of the GTG-, RBA- and RHG- band patterns in sheep chromosomes. 8th European Colloquium on Cytogenetics of Domestic Animals, Bristol, GRANDE BRETAGNE.
HAYES H., PETIT E., 1992. Physical mapping of two different markers to homoeologous chromosomes in sheep, cattle and goat using non-isotopic in situ hybridisation. 10th European Colloquium on Cytogenetics of domestic Animals, Utrecht, PAYS-BAS.
HAYES H., PETIT E., LEMIEUX N., DUTRILLAUX B., 1992. Chromosomal localisation of ovine beta-casein gene by non-isotopic in situ hybridisation and R-banding. 10th European Colloquium on Cytogenetics of Domestic Animals, Utrecht, PAYS-BAS.
VASSART M., SEGUELA A., HAYES H., 1994. Chromosomal evolution in gazelles. 11th European Colloquium on Cytogenetics of Domestic Animals, Copenhagen, DANEMARK.
HAYES H., 1996. Cattle data obtained by ZOO-FISH. XXVth International Conference on Animal Genetics, Tours, FRANCE.
ROGEL-GAILLARD C., COULLIN P., HAYES H., VAIMAN M., CHARDON P., 1997. Centromeric motifs define subsets of swine chromosomes as revealed by Primed IN Situ labeling (PRINS). 1st European Cytogenetics Conference, Athènes, GRECE.
HAYES H., DI MEO G.P., GAUTIER M., LAURENT P., EGGEN A., IANNUZZI I., 2000. Localisation by FISH of the 31 Texas nomenclature type I markers to both Q- and R-banded bovine chromosomes. 14th European Colloquium on Cytogenetics of Domestic Animals, Brno, REPUBLIQUE TCHEQUE.
BONNET A., CLARO F., GAUTIER M., EGGEN A., HAYES H., 2002. Identification by R-banding and FISH-mapping of chromosome arms involved in robertsonian translocations in several species of deer. 15th European Colloquium on Animal Cytogenetics and Gene Mapping, Sorrento, ITALIE. HAYES H., ROGEL-GAILLARD C., ZIJLSTRA C., de HAAN N.A., URIEN C., BOURGEAUX N., BERTAUD M.,
BOSMA A., 2002. Contribution to the rabbit R-banded karyotype nomenclature by FISH localisation of 23 chromosome specific genes on both G- and R-banded chromosomes. 15th European Colloquium on Animal Cytogenetics and Gene Mapping, Sorrento, ITALIE.
CHANTRY-DARMON C., VAIMAN D., HAYES H., de ROCHAMBEAU H., ALLAIN D., URIEN C., BOURGEAUX N., BERTAUD M., CRIBIU E.P., CHARDON P., ROGEL-GAILLARD C., 2002. Towards the construction of an integrated cytogenetic and genetic map of rabbit. XXVIIIth International Conference on Animal Genetics, Göttingen, ALLEMAGNE.
HAYES H., ROGEL-GAILLARD C., ZIJLSTRA C., de HAAN N.A., URIEN C., BOURGEAUX N., BERTAUD M., BOSMA A., 2002. Contribution to the rabbit R-banded karyotype nomenclature by FISH localisation of 23 chromosome specific genes on both G- and R-banded chromosomes. XXVIIIth International Conference on Animal Genetics, Göttingen, ALLEMAGNE.
SAUNIER K., HAYES H., RAYNAUD P., EGGEN A., JULIEN R., WOMACK J.E., PETIT J.M., LEVEZIEL H., 2002. Physical mapping of the bovine 18q24-q26 chromosomal region. 7th World Congress on Genetics Applied to Livestock Production, Montpellier, FRANCE.
CHANTRY-DARMON C., URIEN C., DE ROCHAMBEAU H., ALLAIN D., PENA B., BOLET G., GARREAU H., HAYES H., BERTAUD M., GROHS C., CHADI-TAOURIT S., DERETZ-PICOULET S., LARZUL C., SAVE J.C., CRIBIU E. P., CHARDON P., ROGEL-GAILLARD C. 2005. Carte génétique du lapin: état des lieux et perspectives. 11èmes Journées Recherches Cunicoles, Paris, FRANCE.
5.2.3. Communications par affiches à des colloques
HAYES H., PETIT E., 1990. Comparison of RBG-banded karyotypes of cattle, sheep and goat. Ninth European Colloquium on Cytogenetics of Domestic Animals, Toulouse, FRANCE.
HAYES H., PETIT E., LEMIEUX N., DUTRILLAUX B., 1992. Chromosomal localisation of ovine beta-casein gene by non-isotopic in situ hybridisation and R-banding. 10th European Colloquium on Cytogenetics of Domestic Animals, Utrecht, PAYS-BAS.
HAYES H., MERCIER D., KOHNO K., 1994. Comparative gene mapping of alpha-galactosyltransferase 1 and elongation factor 2 genes in cattle, sheep and goat. 11th European Colloquium on Cytogenetics of Domestic Animals, Copenhagen, DANEMARK.
HAYES H., EGGEN A., DOUD L.K., LEVEZIEL H., BISHOP M.D., 1996. Physical mapping of YACs containing BTA1 microsatellites by FISH. XXVth International Conference on Animal Genetics, Tours, FRANCE.
EGGEN A., DOUD L.K., HAYES H., MURVKE B.T., JURGELLA G., PFISTER-GENSKOW M., DAVIS S.K., TAYLOR J.F., BISHOP M.D., 1996. Construction of a YAC contig in the vicinity of the polled locus in cattle. XXVth International Conference on Animal Genetics, Tours, FRANCE.
ELDUQUE C., EGGEN A., CHAING P.W., KURNIT D., HAYES H., LEVEZIEL H., 1996. Are human ESTs useful tools for bovine gene mapping? XXVth International Conference on Animal Genetics, Tours, FRANCE.
LARSEN N.J., HAYES H., EGGEN A., BISHOP M., KIRPATRICK B.W., 1996. Conservation of close linkage of pituitary adenylate cyclase activating peptide and thymidylate synthase loci between human and bovine. XXVth International Conference on Animal Genetics, Tours, FRANCE.
LAURENT P., ELDUQUE C., EGGEN A., HAYES H., LEVEZIEL H., 1996. The alpha-amylase gene is located on chromosome 3q23-q24 in cattle. XXVth International Conference on Animal Genetics, Tours, FRANCE.
PFISTER-GENSKOW M., HAYES H., EGGEN A., BISHOP M., 1996. Molecular mapping of genes involved in feed intake regulation. XXVth International Conference on Animal Genetics, Tours, FRANCE. SAUNIER K., LEVEZIEL H., PETIT E., GROHS C., NOE G., EGGEN A., HAYES H., JULIEN R., 1996.
Refined mapping of the bovine C blood group system (EAC) on BTA18. XXVth International Conference on Animal Genetics, Tours, FRANCE.
SUN H.S., GAI L., EGGEN A., DAVIS S.K., TAYLOR J., DOUD L.K., BISHOP M., HAYES H., BARENDSE W., VAIMAN D., Mc GRAW R.A., HIRANO T., SUGIMOTO Y., KIRKPATRICK B.W., 1996. A comparative linkage map of human chromosome 13 and bovine chromosome 12. XXVth International Conference on Animal Genetics, Tours, FRANCE.
METENIER-DELISSE L., EGGEN A., HAYES H., LEROUX C., GIRAUD-DELVILLE C., LEVEZIEL H., GUERIN G., MARTIN P., GROSCLAUDE F., 1996. Isolation and molecular characterisation of bovine Rhesus-like transcripts and chromosome mapping of the relevant locus. XXVth International Conference on Animal Genetics, Tours, FRANCE.
WERTH L.A., HAWKINS G.A., EGGEN A., O’BRIEN M., DOUD L.K., HAYES H., ELDUQUE C., KREIGESMANN B., JANSEN S., BISHOP M., 1996. Sequence and genotypic analysis of bovine melanogenic proteins: melanocyte stimulating hormone receptor and tyrosinase related protein 2. XXVth International Conference on Animal Genetics, Tours, FRANCE.
HAYES H., EGGEN A., DOUD L.K., LEVEZIEL H., BISHOP M., 1997. A comparative gene mapping study between bovine chromosome 1 and human chromosomes 3 and 21. 1st European Cytogenetics Conference, Athènes, GRECE.
LAURENT P., ELDUQUE C., HAYES H., JEULIN A., EGGEN A., LEVEZIEL H., 1999. Mapping of human ESTs on a bovine-rodent somatic cell hybrid panel: 58 new assignments in cattle. Plant and Animal Genome VII Meeting, San Diego, ETATS-UNIS.
ROGEL-GAILLARD C., HAYES H., BOURGEAUX N., LAFFONT P., CHARDON P., 1999. First cytogenetic map of porcine type C retroviral sequences. 2nd European Cytogenetics Conference, Vienne, AUTRICHE.
HAYES H., SCHIBLER L., CRIBIU E.P., 1999. Progress in the type I cytogenetic map of cattle. 2nd European Cytogenetics Conference, Vienne, AUTRICHE.
GAUTIER M., LAURENT P., HAYES H., EGGEN A., 2000. Identification of 31 bovine BACs containing the marker genes of the Texas standard. Plant and Animal Genome VII Meeting, San Diego, ETATS-UNIS.
ROY R., GAUTIER M., HAYES H., LAURENT P., EGGEN A., OSTA R., ZARAGOZA P., RODELLAR C., 2000. Isolation and chromosomal localisation of fatty acid synthase (FAS) in cattle. XXVIIth International Conference on Animal Genetics, Minneapolis, ETATS-UNIS.
HAYES H., URIEN C., LALOY E., VAIMAN D., ROGEL-GAILLARD C., 2001. FISH characterization of the R-banded rabbit (Oryctolagus cuniculus) karyotype. 3rd European Cytogenetics Conference, Paris, FRANCE.
HAYES H., SCHIBLER L., EGGEN A., CRIBIU E.P., 2001. Alignment of the bovine genome with the human autosomes. 3rd European Cytogenetics Conference, Paris, FRANCE.
BONNET A., THEVENON S., CLARO F., GAUTIER M., EGGEN A., HAYES H., 2001. Cytogenetic comparison between Vietnamese sika deer and cattle: R-banded karyotypes and FISH-mapping. 3rd European Cytogenetics Conference, Paris, FRANCE.
SAUNIER K., HAYES H., RAYNAUD P., EGGEN A., JULIEN R., WOMACK J.E., PETIT J.M., LEVEZIEL H., 2002. Physical mapping of the bovine 18q24-q26 chromosomal region. 7th World Congress on Genetics Applied to Livestock Production, Montpellier, FRANCE.
DI MEO G.P., PERUCATTI A., HAYES H., INCARNATO D., EGGEN A., IANNUZZI L., 2002. Chromosome localisation of the 31 type I Texas markers in sheep and goat chromosomes by fluorescent in situ hybridisation and R-banding. 15th European Colloquium on Animal Cytogenetics and Gene Mapping, Sorrento, ITALIE.
HAYES H., ELDUQUE C., GAUTIER M., SCHIBLER L., CRIBIU E.P., EGGEN A., 2002. Gene mapping progress in cattle and updated comparative map with man, mouse, rat and pig. XXVIIIth International Conference on Animal Genetics, Göttingen, ALLEMAGNE.
ROY R., GAUTIER M., HAYES H., LAURENT P., ZARAGOZA P., EGGEN A., RODELLAR C., 2002. Isolation and mapping of the mitochondrial glycerol-3-phosphate acyltransferase (GPAM) gene in cattle. XXVIIIth International Conference on Animal Genetics, Göttingen, ALLEMAGNE.
CHANTRY-DARMON C., HAYES H., URIEN C., BERTAUD M., CHARDON P., DE ROCHAMBEAU H., ROGEL-GAILLARD C., 2003. Cartographie chez le lapin: état des lieux. 10ème Journées de la Recherche Cunicole, Paris, FRANCE.
CHANTRY-DARMON C., ROGEL-GAILLARD C., BERTAUD M., URIEN C., PERROCHEAU M., CHARDON P., HAYES H., 2004. Progress in the rabbit cytogenetic map. 16th European Colloquium on Animal Cytogenetics and Gene Mapping, Jouy-en-Josas, FRANCE.
BONNET-GARNIER A., PINTON A., BERLAND H., HAYES H., EGGEN A., DUCOS A., DARRE R. (2004). Sperm nuclei analysis of several 1/29 Robertsonian translocation carriers using fluorescence in situ hybridization. 16th European Colloquium on Animal Cytogenetics and Gene Mapping, Jouy-en-Josas, FRANCE. (poster).
ORDOVAS L., ROY R., HAYES H., ROMERO A., ZARAGOZA P., RODELLAR C., 2004. Characterisation of bovine solute carrier family 27 member 1 (SLC27A1). XXIXth International Conference on Animal Genetics, Tokyo, JAPON.
CHANTRY-DARMON C., HAYES H., URIEN C., SAVE JC., TAOURIT T., GROHS C., DE ROCHAMBEAU H., ALLAIN D., CHARDON P., ROGEL-GAILLARD C., 2004. Establishment of a first generation microsatellite-based genetic map in the rabbit. XXIXth International Conference on Animal Genetics, Tokyo, JAPON.
CHANTRY-DARMON C., HAYES H., ALLAIN D., PENA B., URIEN C., BERTAUD M., DE ROCHAMBEAU H., ROGEL-GAILLARD C., 2004. Construction of an integrated genetic and cytogenetic map in the rabbit. VIIIth World Rabbit Congress, Mexico, MEXIQUE.
7. Liste des étudiants co-encadrés en thèse
Marc Vassart
(thèse soutenue en mai 1994)Evolution et diversité génétique chez les gazelles (Gazella) ; apports de l’électrophorèse des protéines, de la cytogénétique et des microsatellites
Publication:
VASSART M., SEGUELA A., HAYES H., 1995. Chromosomal evolution in gazelles. J Hered 86, 216-227.
Françoise Claro
(thèse soutenue en décembre 1994)Etude comparative du caryotype des espèces de la sous-famille des Hippotraginés – Application des résultats à la gestion des troupeaux en captivité et à la réintroduction de ces espèces dans le milieu naturel
Publications:
CLARO F., HAYES H., CRIBIU E., 1993. The R- and G-banded karyotypes of the sable antelope (Hippotragus niger). J Hered 84, 481-484.
CLARO F., HAYES H., CRIBIU E., 1994. The C-, G-, and R-banded karyotype of the scimitar-horned oryx (Oryx dammah). Hereditas 120, 1-6.
CLARO F., HAYES H., CRIBIU E., 1995. Identification of p and q arms of the blesbok (Damaliscus dorcas phillipsi, Alcelaphinae) RBG-banded chromosomes with comparison to other wild and domestic animals. Cytogenet Cell Genet 70, 268-272.
CLARO F., HAYES H., CRIBIU E., 1996. The karyotype of the addax and its comparison with karyotypes of other species of Hippotraginae antelopes. Hereditas 124, 223-227.
Amélie Bonnet-Garnier
(thèse soutenue en décembre 2001)Etude chromosomique et moléculaire de la sous-famille des Cervinae : Application des résultats à la gestion des troupeaux en captivité d’espèces menacées
Publications:
BONNET A., THEVENON S., CLARO F., GAUTIER M., HAYES H., 2001. Cytogenetic comparison between Vietnamese sika deer and cattle: R-banded karyotypes and FISH mapping. Chromos Res 9,673-687.
BONNET-GARNIER A., CLARO F., THEVENON S., GAUTIER M., HAYES H., 2003. Identification by R-banding and FISH of chromosome arms involved in Robertsonian translocations in several deer species. Chromosome Res 11, 649-663.
Céline Chantry-Darmon
(thèse soutenue en avril 2005)Construction d’une carte intégrée cytogénétique et génétique chez le lapin
Publications:
CHANTRY-DARMON C., ROGEL-GAILLARD C., BERTAUD M., URIEN C., PERROCHEAU M., CHARDON P., HAYES H., 2003. 133 new gene localizations on the rabbit cytogenetic map. Cytogenet Genome Res 103, 192-201.
CHANTRY-DARMON C., BERTAUD M., URIEN C., CHADI-TAOURIT S., PERROCHEAU M., ROGEL-GAILLARD C., HAYES H., 2005. Expanded comparative mapping between man and rabbit and detection of a new conserved segment between HSA22 and OCU4. Cytogenet Genome Res 111:134-139.
CHANTRY-DARMON C., URIEN C., HAYES H., BERTAUD M., CHADI-TAOURIT S., CHARDON P., VAIMAN D., ROGEL-GAILLARD C., 2005. Construction of a cytogenetically anchored microsatellite map in rabbit. Mamm Genome 16:442-459.
8.
Abréviations
ADN Acide DésoxyriboNucléique
APRT Adenine PhosphoRibosylTransferase ARN Acide RiboNucléique
BAC Bacterial Artificial Chromosome BrdU BromodeoxyUridine
BTA Bos taurus
CEA Commissariat à l’Energie Atomique CHI Capra hircus
CNP Cervus nippon pseudaxis CsCl Chlorure de césium
ENVT Ecole Nationale Vétérinaire Toulouse EST Expressed Sequence Tag
FISH Fluorescence In Situ Hybridization
GREG Groupement de recherche et d’études sur le génome HPRT Hypoxanthine-guanine PhosphorRibosylTransferase HSA Homo sapiens
IMCB Institute for Molecular and Cellular Biology INRA Institut National de la Recherche Agronomique kb kilobase
LGbC Laboratoire de Génétique biochimique et Cytogénétique LGC Laboratoire de Génétique Cellulaire
LREG Laboratoire de Radiobiologie et d ’Etude du Génome Mb Megabase
MMU Mus musculus OAR Ovis aries pb paire de bases
PCR Polymerase Chain Reaction PRINS Primed IN Situ labeling QTL Quantitative Trait Locus
RBG bandes R par incorporation de BrdU et coloration au Giemsa
RBP bandes R par incorporation de BrdU et coloration à l’iodure de Propidium RNO Rattus norvegicus
rob translocation robertsonienne TK Thymidine Kinase
SSC Sus scrofa
UMR Unité Mixte de Recherches YAC Yeast Artificial Chromosome
Deuxième partie:
Synthèse des recherches sur le caryotype et la
carte cytogénétique d’animaux d’élevage
1. Introduction
La génomique analyse la structure et le contenu du génome dans sa globalité, la génétique
recherche les mécanismes de l’hérédité et de la variabilité biologique et la cytogénétique étudie
l’architecture et le fonctionnement du génome dans le noyau, en métaphase (chromosomes) et en interphase. Les méthodes et les données de ces trois disciplines-clés de la biologie font partie intégrante des activités de recherche du département de génétique animale (DGA) de l’INRA. Le DGA a pour double mission de contribuer à la biologie intégrative animale en apportant une dimension génétique, et d'élaborer les méthodes et les outils d'aide à la gestion génétique des populations d'animaux d’élevage terrestres et aquatiques. A mon arrivée à l’INRA en 1986, la cartographie globale du génome de ces espèces était une des priorités du DGA et se déclinait en plusieurs volets dont la localisation de gènes ou de marqueurs par hybridation in situ sur leurs chromosomes. L’hybridation in situ était alors un outil incomparable pour apporter des informations précises de cartographie chromosomique, pour ancrer physiquement les groupes de liaison et les cartes génétiques sur les chromosomes et pour construire les premières cartes comparées inter-espèces. La cartographie comparée entre les espèces d’élevage et l’homme ou la souris, espèces aux cartes déjà denses, et la cartographie fine de régions candidates, étaient essentielles aux stratégies d’identification de gènes impliqués dans des caractères d’intérêt agronomique (stratégies de clonage positionnel). Le premier exemple de gène d’intérêt identifié est le gène PRKAG3 dont la mutation dite RN- est responsable d’une baisse de qualité de la viande chez les porcs de race Hampshire. Il a été déterminé et publié par un consortium européen incluant l’INRA, la Suède et l’Allemagne (Milan et al, 2000). Depuis, ~50 gènes majeurs ou responsables d’anomalies génétiques chez les ruminants domestiques, le porc, le cheval et le poulet ont été identifiés.
De 1986 à 2005, mes travaux de recherche ont porté sur les caryotypes et les cartes cytogénétiques de la vache puis du lapin et sur leur comparaison avec ceux d’autres espèces domestiques, sauvages et humaine. Les objectifs ont été tout d’abord de choisir et de mettre au point les techniques de préparation, d’analyse et d’hybridation in situ de ces chromosomes puis de fournir des données utiles à la communauté scientifique animale. Cette partie du rapport d’habilitation présentera la synthèse de ces travaux, avec une petite incursion dans ceux de mon stage post-doctoral au Japon de 1984 à 1986.
2. Le caryotype : constante caractéristique d’un individu ou d’une espèce
2.1. De l’individu aux chromosomes
Le noyau de toutes les cellules d’un individu possède le même patrimoine génétique dont le support physique, l’ADN, est contenu dans la chromatine formée de molécules d’ADN et de protéines. Au cours de chaque cycle cellulaire, la chromatine passe par deux états (figure 2.1): état
dispersé mais organisé en territoires chromosomiques durant l’interphase et état de compaction maximale sous la forme d’entités distinctes, les chromosomes, durant la mitose. Chaque territoire chromosomique ou chromosome renferme une seule molécule d’ADN continue et repliée de nombreuses fois sur elle-même. Chez toutes les cellules somatiques nucléées d’un individu normal d’une espèce donnée, le nombre, la taille et la forme des chromosomes sont constants et caractéristiques. Ils se regroupent par paires de chromosomes homologues sauf les chromosomes sexuels souvent de taille et de forme très différentes (figure 2.2). Par exemple, l’homme a 23
paires de chromosomes, la vache et la chèvre 30, le mouton 27, le lapin 22 et le porc 19.
Dans le noyau, les chromosomes ne sont présents qu’au moment de la division cellulaire. Pour les besoins de l’analyse caryotypique, les cellules cultivées in vitro doivent donc être récoltées en phase mitotique. Le nombre et la qualité des étalements chromosomiques obtenus dépendent de la capacité des cellules à (1) se diviser in vitro et (2) répondre efficacement à l’action d’agents de synchronisation du cycle cellulaire comme le méthotrexate ou la thymidine. Les cultures classiques de lymphocytes à partir de prélèvements de sang bovin, ovin et caprin ont un très faible indice mitotique. Confrontée à ce problème, je les ai remplacées par la culture de cellules de type fibroblastique décrite dans Hayes et al (1991) que j’ai mis au point à partir de poumon fœtal. Leur culture donne des résultats réguliers grâce à un indice mitotique élevé et un nombre potentiel de générations d’environ 50 contre 20 pour les cellules de tissu adulte. J’ai utilisé cette technique pour tous les résultats rapportés dans ce mémoire.
2.2. Du choix des bandes R pour identifier les chromosomes (1986-1988)
La propriété la plus exploitée et, paradoxalement, la moins bien comprise des chromosomes est de présenter le long de leurs bras, à la suite de colorations ou de traitements spécifiques, des bandes plus ou moins colorées. Pour une technique de marquage chromosomique donnée, la succession et la taille variable de ces bandes constituent un motif précis et reproductible (Hayes, 1998). Cette propriété, très précieuse, permet de distinguer et d’identifier les chromosomes donc de les assembler en un caryotype (figure 2.2) qui est l’image classée du contenu chromosomique du
noyau. L’analyse du caryotype en bandes rend possible la description de remaniements numériques ou structuraux, la localisation de gènes ou marqueurs à une bande donnée et la comparaison des chromosomes entre espèces proches ou éloignées. Les traitements et colorations utilisés pour observer les chromosomes agissent sur la chromatine, le complexe ADN-protéines qui compose leur structure. La chromatine se divise en hétérochromatine et euchromatine. L'hétérochromatine ne change pas d'état de condensation au cours du cycle cellulaire et contient principalement des séquences répétées d'ADN non codant. Par contre, l’euchromatine apparaît décondensée durant l’interphase et correspond à la majeure partie d’un génome. Les méthodes de marquage chromosomique sont apparues dans les années 1970 et les plus utiles pour l’analyse des chromosomes de mammifères marquent l’euchromatine. L’euchromatine possède une structure bipartite composée pour moitié de l’euchromatine des bandes G ou Q et pour moitié de celle des bandes R. A chacune de ces deux fractions, sont associées des propriétés biochimiques et fonctionnelles opposées (tableau 2.1) à l’origine des motifs de bandes visualisés au microscope
après traitement et coloration. Le motif des bandes R est le contretype de celui des bandes Q et G comme le montrent les exemples dans la figure 2.3 ci-dessous.
Le choix du type de bandes utilisé dans un laboratoire est en grande partie une question d’école, avec l’école anglo-saxonne des bandes Q et G et l’école française des bandes R. Suite à mon expérience avec les bandes Q au Japon (cf. figures 2.8 et 2.9) puis avec les bandes RBG et RBP
dans le laboratoire de B. Dutrillaux, j’ai souscrit au marquage en bandes R. En effet, les bandes R révélées après incorporation de BrdU dans l’ADN de la cellule vivante et colorées au Giemsa (bandes RBG) ou à l’iodure de propidium (bandes RBP) produisent des caryotypes d’une définition supérieure. Ce choix s’est trouvé pleinement justifié lorsque j’ai introduit l’hybridation in situ en fluorescence (FISH) au LGbC (paragraphe 3.3.2).
En 1986, les caryotypes prométaphasiques en bandes R obtenus après synchronisation des cultures et incorporation de BrdU pour les espèces autres que l’homme étaient rares dans la littérature. Après avoir adapté la technique de Viegas-Péquignot et Dutrillaux (1978) aux chromosomes de bovidés domestiques (figure 2.4a) (Hayes et al, 1991), elle a été appliquée avec
succès à d’autres espèces de bovidés sauvages [Hippotragus niger, Oryx dammah, Damaliscus dorcas phillipsi, Addax nasomaculatus (Claro et al, 1993 ; 1994 ; 1995 et 1996)], de cervidés [Axix axis (figure 2.4b), Cervus nippon pseudaxis, C. albirostris, C. timorensis russa, C. unicolor, C. eldi siamensis (Bonnet-Garnier et al, 2001 et 2003)] et au lapin (figure 2.4c) (Hayes et al, 2002), mais
aussi au porc, au cheval et à la poule. La plupart des caryotypes ainsi publiés ont apporté des précisions quant à la description des motifs des bandes et ont contribué aux études de cytogénétique comparée, en particulier ceux des bovidés sauvages comparés au caryotype bovin pris comme référence (cf. paragraphe 4.1).
2.3. De la nécessité des caryotypes de référence (ISCNDA89, ISCNDB2000, lapin)
En 1988, la préparation de caryotypes en bandes RBG bovin, ovin et caprin se pratiquait en routine au LGbC mais le problème de leur classement selon la nomenclature internationale en cours se posait et retardait leur publication. En effet, à cette date, les seuls caryotypes standards reconnus au niveau international pour ces trois espèces étaient ceux établis en bandes G en 1976 lors de la conférence de Reading (publication connue sous le nom Reading 1976 mais éditée en 1980). Ces caryotypes, produits à partir de chromosomes métaphasiques donc de moins bonne définition que les chromosomes prométaphasiques, ne permettaient pas de déterminer avec certitude toutes les correspondances entre chromosomes en bandes G et en bandes R. Il faut préciser ici, que les chromosomes de bovidés (figure 2.4a) présentent des difficultés
supplémentaires par rapport à ceux de l’homme (2n=46), du porc (2n=38) ou du lapin (2n=44): ils
BTA9 : chromosome bovin 9 (BTA pour Bos taurus)
sont plus nombreux (vache et chèvre, 2n=60 ; mouton, 2n=54), ils sont tous ou presque tous acrocentiques, c’est à dire dépourvus de différences morphologiques entre eux, et ils forment un gradient de taille continu du plus grand au plus petit. Le problème de l’absence de nomenclature standard en bandes R allait devenir encore plus critique avec la localisation de gènes par hybridation in situ qui débutait en parallèle au LGbC chez ces espèces. Dans ce contexte, il était absolument nécessaire de trouver un accord international sur le classement et la nomenclature de la numérotation des bandes des chromosomes bovins, caprins et ovins en bandes R.
Avec P. Popescu, nous avons donc décidé d’organiser une deuxième conférence de standardisation des caryotypes bovin, ovin et caprin qui s’est tenue à l’INRA en mai 1989 et qui a réuni 29 représentants de la communauté cytogénétique animale. Les objectifs suivants ont été atteints pour l’espèce bovine et publiés dans un document intitulé ISCNDA1989 (International System for Cytogenetic Nomenclature of Domestic Animals) :
• création de caryotypes standards en bandes R à partir de chromosomes prométaphasiques (400 bandes) respectant le classement des chromosomes métaphasiques (250 bandes) de la nomenclature Reading 1976,
• établissement des correspondances entre chromosomes en bandes G et bandes R, • description détaillée du motif des bandes G et R,
• création d’une nomenclature des bandes avec leur numérotation et de représentations schématiques des chromosomes indispensables pour caractériser les anomalies chromosomiques et donner la position des gènes sur les chromosomes en cartographie.
Pour la chèvre et le mouton, les images de qualité ont manqué pour atteindre ces objectifs. Cependant, les caryotypes des trois espèces étant très similaires (cf. paragraphe 4.1.1), il a été
décidé d’extrapoler les données bovines aux deux autres espèces en attendant de nouveaux documents.
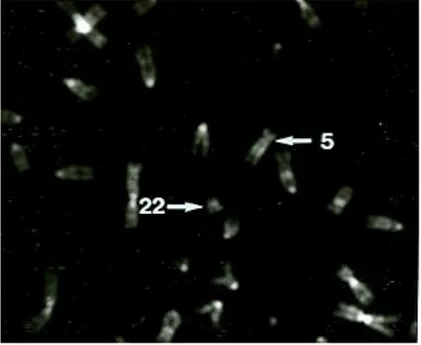
La nomenclature ISCNDA1989 marquait un progrès certain pour la cytogénétique des bovidés domestiques mais elle n’était pas complète et contenait quelques erreurs par rapport à la nomenclature de 1976. Rapidement identifiées, ces erreurs n’ont été officiellement corrigées qu’en 2000. En effet, j’ai bâti une stratégie consistant à hybrider des sondes spécifiques à chacun des chromosomes bovins dans les deux systèmes de bandes G/Q et R. Ceci a permis d’éliminer tous les points d’achoppement. Tout d’abord, le comité de standardisation (Texas,1995) a établi une liste de 31 gènes représentatifs de chacun des 31 chromosomes bovins (Popescu et al, 1996). Au LGbC, nous avons isolé des clones de BAC bovin contenant ces gènes (Gautier et al, 2001), qui ont ensuite servi de sondes en FISH pour les localiser d’une part sur les chromosomes bovins en bandes R au LGbC et d’autre part sur les chromosomes en bandes Q (équivalentes aux bandes G) par L. Iannuzzi en Italie (Hayes et al, 2000). La figure 2.5 illustre cette démarche avec l’exemple du gène de la
lactoglobuline bêta (LGB) sur le chromosome bovin 11. Ces 31 gènes ont été aussi localisés sur les chromosomes de buffle (Iannuzzi et al, 2001) et les chromosomes ovins et caprins (Di Meo et al, 2003). Ces résultats ont permis de finaliser la nomenclature des chromosomes bovins, ovins et caprins en bandes G et R en 2000. Publiée en 2001 (ISCNDB2000 International System for the Nomenclature of Domestic Bovids), cette nomenclature comprend des caryotypes en bandes Q/G et
R ainsi que les idiogrammes correspondants pour les trois espèces (exemple du caryotype caprin dans la figure 2.6). Elle constitue un langage commun et fiable pour toute la communauté
internationale et représente l’aboutissement de nombreuses années de travail et de discussions pour convaincre les cytogénéticiens étudiant ces espèces de l’intérêt des bandes R.
Lorsque nous avons décidé de construire une carte cytogénétique du lapin, les mêmes impératifs techniques rendaient nécessaire une nomenclature chromosomique en bandes R pour cette espèce. D’emblée, j’ai appliqué la même stratégie de localisation de gènes sur chromosomes en bandes G et R avec la collaboration de C. Rogel-Gaillard (LREG-INRA) et de A. Bosma (université d’Utrecht, Pays-Bas). Un consensus international a été atteint rapidement grâce aux données de FISH et à la taille plus restreinte de la communauté cytogénétique lapin (Hayes et al, 2002).
3. La carte cytogénétique : arrangement linéaire des loci sur les chromosomes
3.1. Objectifs de la cartographie de gènes
La cartographie des génomes a pour but ultime de définir l'inventaire complet des gènes d'un génome. Aujourd’hui, ce but est presque atteint pour les génomes dont la séquence complète est en cours d’annotation. C’est le cas des génomes de l’homme, du chimpanzé, de la souris, du rat, du chien, de la vache, de la poule et de plusieurs poissons (cf. le site http://www.ensembl.org/index.html). Dans les années 1980, l’objectif de la cartographie était plus modeste, il s’agissait de se donner un outil pour repérer et isoler des gènes intéressants et consistait à déterminer la position de loci (gènes ou marqueurs) le long des chromosomes. La position des loci pouvait être définie soit par un repère physique sur les chromosomes (les bandes), c’est la carte cytogénétique avec un pouvoir de résolution de 10 à quelques mégabases, soit par une distance relative déduite des fréquences de recombinaison entre loci, c’est la carte génétique exprimée en centimorgans avec un pouvoir de résolution légèrement supérieur. La carte cytogénétique peut inclure tout locus, gène ou marqueur anonyme pour lequel une sonde donc une séquence est disponible. Par contre, la carte génétique nécessite des loci polymorphes bien répartis sur le génome pour pouvoir suivre leur transmission dans des familles sur trois générations au moins, ce qui est le cas des marqueurs microsatellites mais pas de la majorité des gènes. L’enjeu était alors d’établir des ponts entre ces cartes afin de les intégrer entre elles et de faire le lien entre données phénotypiques et génotypiques. Un autre aspect complémentaire de la cartographie des génomes est de comparer les cartes entre espèces différentes comme je l’ai déjà mentionné. C’est dans ce contexte que la cartographie cytogénétique s’est avéré essentielle pour constituer un réseau de relations entre les cartes de différents types et de différentes espèces.
3.2. Construction d’une carte cytogénétique
La construction des cartes cytogénétiques utilise soit l’analyse d’hybrides cellulaires somatiques soit l’hybridation in situ sur les chromosomes.