HAL Id: tel-01359183
https://tel.archives-ouvertes.fr/tel-01359183
Submitted on 2 Sep 2016HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Protéine kinase GCN2 et phosphorylation de l’intégrase
du VIH-1
Anaïs Jaspart
To cite this version:
Anaïs Jaspart. Protéine kinase GCN2 et phosphorylation de l’intégrase du VIH-1. Immunologie. Université de Bordeaux, 2014. Français. �NNT : 2014BORD0405�. �tel-01359183�
THESE
Pour le
DOCTORAT DE L’UNIVERSITE DE BORDEAUX
Mention : Sciences, Technologie, SantéOption : Microbiologie
Présentée et soutenue publiquement Le 12 Décembre 2014
Par
JASPART Anaïs
Née le 10 Juillet 1987 à Mont Saint Martin
Protéine Kinase GCN2 et phosphorylation de l'intégrase
du VIH-1
Membres du Jury
Mme Marie Edith Lafon………..……….Présidente du Jury
Professeur, Bordeaux
Mme Angela Ciuffi……….Rapporteur
Maître d'enseignement et de recherche, Université de Lausanne
Mr Marc Lavigne……….Rapporteur
Chargé de recherche, Institut Pasteur, Paris
Mr Jean-Marie Schmitter ………Examinateur
Professeur, Bordeaux
Mme Anne-Catherine Maurin……….Examinateur Chargée de Recherche, INRA, Clermont Ferrand
Mme Marie-Line Andréola ………..………………Directrice de Thèse Directrice de recherche, CNRS, Bordeaux
Protéine Kinase GCN2 et phosphorylation de l'intégrase du
VIH-1
Résumé
L’intégrase (IN) du VIH-1 est une enzyme clé qui catalyse l’insertion stable du génome viral dans celui de la cellule infectée. D’autre part, l’IN participe également à de nombreuses étapes du cycle viral telles que la transcription inverse ou la maturation virale. La compréhension des mécanismes impliqués dans la régulation de l’intégration cellulaire au cours de l’infection est un enjeu important. L’IN fait partie du complexe de préintégration composé de facteurs cellulaires et viraux. La dynamique des interactions au sein de ce complexe régule les activités catalytiques mais également non catalytiques de l’IN. C’est dans ce contexte de recherche de nouveaux cofacteurs de l’intégrase que nous avons identifié une interaction entre l’IN et la protéine Kinase GCN2.
Mon travail de thèse s’est orienté sur trois questions autour de l’étude du rôle de cette interaction IN/GCN2. Dans un premier temps, le rôle de la protéine kinase cellulaire GCN2 au cours du cycle viral a été étudié. Nous avons pu montrer que l’infection par le VIH-1 provoque un stress activant GCN2. Cette activation aboutit à une inhibition de la traduction dès les premières heures de l’infection.
GCN2 est capable de phosphoryler l’IN du VIH-1 sur deux positions : les sérines en position 24 et 255. L’étude du rôle de la phosphorylation de l’IN par GCN2 a permis de montrer que l’absence de phosphorylation de l’IN entraîne une stimulation de l’infection. GCN2 via la phosphorylation de l’IN a donc un effet restrictif sur l’infection par le VIH-1.
L’étude du domaine d’interaction entre l’IN et GCN2 a permis d’identifier un résidu essentiel de l’IN, l’acide glutamique en position 85 (E85). En effet, la mutation E85A de l’IN entraîne la production de virus non infectieux, suite à un défaut de maturation.
MOTS-CLES : VIH-1, intégrase, GCN2
Kinase GCN2 and the phosphorylation of HIV-1 integras
e
Abstract
HIV-1 integrase (IN) catalyzes the integration of the viral DNA into the cellular genome. Besides, IN is also involved in other steps of the viral life cycle like the reverse transcription or the viral maturation. Our group is interested in the mechanisms involved in the regulation of cellular integration during the infection. IN is part of a pre-integration complex composed of cellular and viral proteins. The dynamic of these interactions regulates IN activities but also non catalytic activities such as nuclear import and tethering of the complex to the integration site. During the
identification of news partners of IN, an interaction between IN and the kinase GCN2 was characterized My PhD project is composed of 3 topics:
The role of the cellular kinase GCN2 was studied during the viral cycle. We showed that GCN2 is activated upon HIV-1 infection. This activation leads to a general decrease of cellular translation during the first hours of infection. GCN2 is able to phosphorylate IN on two positions: the serines in position 24 and 255. The absence of IN phosphorylation causes a stimulation of HIV-1 infection. We demonstrate that GCN2 affects the viral cycle via the phosphorylation of IN.
Binding domain analysis between IN and GCN2 led to the identification of a critical residue of IN, the glutamic acid in position 85 (E85). Mutations E85A of IN impair the viral production by inhibiting the viral maturation.
Remerciements
Je tiens, tout d’abord à remercier le Dr Marie-Line Andréola pour m’avoir accepté dans son laboratoire. Merci de m’avoir soutenu ces cinq années et notamment pour la préparation du concours de l’école doctorale ce qui m’a permis d’obtenir une bourse pour pouvoir conduire ma thèse dans ton équipe. J’ai beaucoup appris à tes côtés mais surtout tu m’as montré qu’en science la rigueur était essentielle. Cela peut paraître évident qu’il faut répéter les expériences plus d’une fois pour en tirer des conclusions, mais on se rend vite compte que tout le monde ne possède pas cette rigueur dont tu fais preuve. J’ai apprécié l’autonomie et la confiance que tu m’as donné ce qui m’a permis de m’épanouir dans ce que je faisais. Nos longues discussions scientifiques ou non et nos sorties shopping vont me manquer. Je ne dirai jamais assez que j’ai eu énormément de chance de t’avoir comme directrice de thèse. Et même si comme toutes les thèses, il y a eu des bons comme de mauvais moments, je suis heureuse d’avoir eu cette expérience et tu es en grande partie responsable de la bonne finalité de la chose. Merci pour cette aventure passionnante et n’oublie pas « ça se tente ».
Une autre personne importante durant ma thèse fut Christina Calmes alias Christinette. Travailler à tes côtés fut un plaisir, tu es toujours prête à rendre service. Ta motivation m’a toujours impressionné et j’espère que tu la garderas. Je pense que l’on peut dire que tu es une assistante ingénieure en or. Merci de ton implication dans le laboratoire mais surtout dans mon projet de thèse. Je te souhaite bonne chance pour la suite.
Merci Mathieu pour la gentillesse dont tu fais toujours preuve. Certaines personnes sont toujours disponibles pour aider les autres et tu en fais partie. J’espère que tu vas prendre le projet en main et en faire de belles choses ce dont je ne doute pas.
Merci également aux autres membres de l’équipe actuels Vincent, Moh, Juju, Pantxika, Sandrine et Paty ou encore anciens Ludi et Ophélie pour leurs conseils et leur contribution au projet.
Je souhaite remercier le Dr Michel Ventura. Michel est toujours de bonne humeur et à l’écoute et il faut savoir qu’il n’a jamais. Mais effectivement on finit toujours par dire « eh oui michel V avait raison ». Merci pour les nombreux conseils que tu m’as donnés et pour les discussions-débats qui ont animé bien des pauses café.
Merci aux autres membres de l’équipe HCV, notamment Chloé. Nos attaques contre Cyril ou nos délires du vendredi avant le week end vont me manquer.
Egalement un petit remerciement à Cyril alias cyrilou. J’ai toujours pu compter sur toi, la preuve tu es même venu m’ouvrir lorsque je me suis retrouvée enfermée chez moi par inadvertance . Tu as été un peu mon grand frère dans ce laboratoire toujours le premier pour me taquiner, mais aussi pour me donner des conseils, m’écouter, m’encourager. Nos discu-blagues vont me manquer. Tu as largement contribué aux bonnes conditions dans lesquelles j’ai réalisé ma thèse.
pour parler des potins, des bons plans, de top chef avant de partir du boulot.
Merci à Simon pour ton soutien durant ces trois ans. Le labo et les gens du labo sont importants pour toi et ça se voit. Tu es toujours disponible pour partager ton expérience et donner un coup de main à qui en a besoin. En tout cas, tu as été présent pour moi et je te remercierai toujours pour cela. Tu es une personne généreuse et j’ai eu beaucoup de chance de connaître et te côtoyer. Merci à toutes mes amies, Popo, Lolo, Nouga, Fieur, Méli, Marie et Strich qui ont été présentes durant toutes ces années. Votre amitié depuis tout ce temps est essentielle pour moi, vous êtes ma deuxième famille.
Merci à mes collègues de master Rémi, Adrien, Alice et Elodie pour les bons moments que l’on a passés en dehors du labo, avec une spéciale dédicace pour Rémi et nos pizzas-bières du lundi. Je souhaite remercier la personne qui me supporte tous les jours, mon Marco. Merci de toujours trouver les mots pour me faire rire, de m’avoir épaulé et soutenu lors de ma thèse. Tu es ma pépite et merci d’être là pour moi.
Enfin, je ne serai pas là où j’en suis aujourd’hui sans mes parents et mon petit frère. Donc merci pour tout ce que vous avez fait et faites encore pour moi. Vous m’avez toujours soutenu dans mes choix et j’espère que vous êtes fiers de moi aujourd’hui. En tout cas vous pouvez être fiers de vous parce que vous êtes des parents et un petit frère formidables.
SOMMAIRE ... 2
ABRÉVIATIONS ... 1
INTRODUCTION ... 1
1.
L’INFECTION PAR LE VIH-1 ... 3
1.1.
L
E SYNDROME DE L’
IMMUNODEFICIENCE ACQUISE OUSIDA ... 3
a. Historique ... 3
b. Epidémiologie : situation actuelle et tendance ... 4
c. Variabilité génétique du VIH-1 ... 5
d. L’ origine du VIH ... 7
e. Physiopathologie de l’infection par le VIH-1 ... 7
f. Diagnostic ... 8
1.2.
L
EVIH-1 ... 8
a. Classification ... 8 b. Structure du virus ... 9 c. Le génome viral ... 10 d. Protéines virales ... 111.3.
L
E CYCLE REPLICATIF... 17
a. Etapes précoces ... 18 b. Etapes tardives ... 221.4.
L
ES FACTEURS DE RESTRICTIONS... 26
a. Le facteur de restriction TRIM5α... 27
b. Le facteur de restriction APOBEC3G ... 27
c. Le facteur de restriction BST-2 ... 27
d. Le facteur de restriction SAMHD1 ... 27
1.5.
L
A LUTTE ANTIRETROVIRALE... 28
a. Les inhibiteurs de l’entrée ... 28
b. Les Inhibiteurs de la transcription inverse ... 29
Les NRTIs ... 29
Les NNRTIs ... 29
c. Les inhibiteurs de la protéase (IPs) ... 30
d. Les inhibiteurs de l’intégrase (INIs) ... 30
a. Le domaine N-terminal (1-49) ... 31
b. Le domaine central (50-212) ... 32
c. Le domaine C-terminal (213-288) ... 33
d. L’intasome fonctionnel de l’IN ... 33
2.2.
M
ECANISME D’
ACTION DE L’
INTEGRASE... 35
a. Le 3’-end processing ... 35
b. Le transfert de brin ... 35
c. La réparation post-intégration ... 35
d. Oligomérisation de l’IN ... 36
2.3.
A
CTIVITES DE L’
INTEGRASE IN VITRO... 37
a. Le 3’-end processing et le transfert de brin ... 37
b. La désintégration ... 37
c. L’intégration concertée (IC) ... 38
2.4.
L
A SELECTIVITE DE L’
INTEGRATION... 39
2.5.
L
ES COFACTEURS DE L’
INTEGRATION... 41
a. Les cofacteurs viraux ... 41
b. Les cofacteurs cellulaires ... 41
2.6.
A
CTIVITES NON CATALYTIQUES DE L’
INTEGRASE... 44
a. Rôle de l’IN lors de la transcription inverse ... 44
b. Rôle de l’IN lors de l’import nucléaire ... 45
c. Rôle de l’IN lors des étapes tardives ... 45
3.
LES MODIFICATIONS POST-TRADUCTIONNELLES DES
INTEGRASES ... 47
3.1.
L’
ACETYLATION... 47
3.2.
L’
UBIQUITINILATION... 48
3.3.
L
ASUMO
YLATION(S
MALLU
BIQUITIN-
LIKEM
ODIFIER) ... 49
3.4.
L
A PHOSPHORYLATION... 50
4.
LA PROTEINE KINASE CELLULAIRE GCN2 ... 52
4.2.
S
TRUCTURE ET FONCTION DEGCN2 ... 53
4.3.
L
ES CONSEQUENCES DE L’
ACTIVATION DEGCN2 ... 56
4.4.
GCN2
ET LES VIRUS... 57
5.
LES OBJECTIFS DE L’ETUDE ... 60
MATERIELS ET METHODES ... 62
1.
MATERIELS ... 64
1.1.
C
ULTURE CELLULAIRE... 64
a. Bactéries ... 64
b. Cellules de mammifères ... 64
1.2.
V
IRUS ET LENTIVIRUS PSEUDO-
TYPESVSV-
G... 65
1.3.
P
LASMIDES... 65
a. Plasmide d’expression dans la bactérie ... 65
b. Plasmide d’expression dans les cellules humaines... 65
c. Plasmides pour la production des lentivirus pseudo-typés VSV-g ... 66
1.4.
O
LIGONUCLEOTIDES... 67
1.5.
L
ES PROTEINES COMMERCIALES... 69
1.5.
L
ES ANTICOPRS... 69
2.
METHODES ... 70
2.1.
C
ULTURES BACTERIENNES... 70
2.2.
C
ULTURES DE CELLULES MAMMIFERES... 70
a. Les virus : VIH-1 ... 71
b. Les virus : les lentivirus pseudo-typés VSV-g ... 72
c. Transfection ... 73
d. Préparation des cellules pour le microscope ... 75
2.3.
M
ANIPULATIONS DES ACIDES NUCLEIQUES... 75
a. Extraction des acides nucléiques de cellules infectées ... 75
e. Séquençage haut débit des sites d’intégration ... 76
2.4.
L
A MANIPULATION DES PROTEINES... 79
a. Production de l’IN recombinante ... 79
b. Production d’eIF2α recombinante... 80
c. Immunoprécipitation ... 81
d. Digestion trypsique des protéines ... 82
e. Gels de protéines et Western Blot ... 82
f. Détection des protéines néosynthétisées ... 83
2.5.
T
EST IN VITRO... 84
a. Activité ADN polymérase ARN dépendante de la RT ... 84
b. Activités de l’intégrase ... 85
c. Tests de phosphorylation ... 86
RESULTATS - DISCUSSIONS ... 88
PARTIE 1 : ROLE DE GCN2 DURANT L’INFECTION PAR LE VIH-1 . 90
1.
OBJECTIFS ... 90
2.
INTERACTION IN/GCN2 ... 91
2.1.
I
NTERACTION DE L’IN
AVECGCN2
CHEZ LA LEVURE... 91
2.2.
I
NTERACTION DE L’IN
AVECGCN2
HUMAINE... 92
3.
GCN2 ET L’INFECTION PAR LE VIH-1 ... 93
3.1.
A
CTIVATION DEGCN2
LORS DE L’
INFECTION PAR LEVIH-1 ... 93
3.2.
R
OLE DEGCN2
AU COURS DE L’
INFECTION... 96
3.3.
T
RADUCTION DES PROTEINES AU COURS DE L’
INFECTION PAR LEVIH-1 ... 98
3.4.
R
ESTAURATION DE LA TRADUCTION CELLULAIRE... 99
4.1.
I
NTERACTIONIN/GCN2 ... 104
4.2.
A
CTIVATION DEGCN2
PAR L’
INFECTION VIRALE... 104
4.3.
GCN2
EST UN FACTEUR DE RESTRICTION DUVIH-1 ... 105
4.4.
L
A TRADUCTION DE LA CELLULE EST INHIBEE APRES L’
INFECTION... 105
4.5.
R
ESTAURATION DE LA TRADUCTION... 106
PARTIE 2 : PHOSPHORYLATION DE L’INTEGRASE DU VIH-1 ... 109
1.
OBJECTIFS ... 109
2.
PHOSPHORYLATION DE L’IN DU VIH-1 ... 110
2.1.
P
HOSPHORYLATION DE L’IN
PARGCN2
IN VITRO... 110
2.2.
I
DENTIFICATION D’
UN SITE PUTATIF D’
INTERACTIONIN/GCN2 ... 112
2.3.
I
DENTIFICATION DES SITES DE PHOSPHORYLATION... 113
2.4.
P
HOSPHORYLATION DES SERINES24
ET255
DE L’IN
DUVIH-1 ... 115
3.
ROLE DE LA PHOSPHORYLATION DE L’IN ... 117
3.1.
E
FFET DES MUTATIONS SUR L’IN
IN VITRO... 118
3.2.
R
OLE DE LA PHOSPHORYLATION SUR LE CYCLE VIRAL... 120
a. Effet des mutations sur l’infectivité ... 120
b. Effet des mutations sur l’étape d’intégration ... 123
c. Effet des mutations de l’IN sur la transcription inverse ... 124
3.3.
R
OLE DELEDGF/
P75
SUR LA PHOSPHORYLATION DE L’IN ... 125
a. Effet des mutations sur la localisation cellulaire de l’IN ... 127
b. Effet des mutations sur la stabilité de l’IN ... 128
c. Sélectivité d’intégration ... 129
4.
PHOSPHORYLATION DES IN RETROVIRALES ... 134
a. Phosphorylation des IN rétrovirales ... 134
b. Identifications des sites de phosphorylations ... 135
5.2.
R
OLE DE LA PHOSPHORYLATION DE L’IN
DUVIH-1 ... 137
5.3.
R
OLE DE LA PHOSPHORYLATION DELEDGF/
P75 ... 140
PARTIE 3 : CARACTERISATION DU MUTANT E85A DE L’IN... 143
1.
OBJECTIFS DE L’ETUDE ... 143
2.
EFFET DE LA MUTATION E85A SUR LES PROPRIETES DE
L’IN IN VITRO ... 144
2.1.
E
TAT D’
OLIGOMERISATION... 144
2.2.
C
HANGEMENT DE CONFORMATION... 144
2.3.
A
CTIVITES CATALYTIQUES... 145
3.
EFFET DE LA MUTATION E85A SUR LA REPLICATION
VIRALE ... 146
3.1.
E
FFET SUR L’
INTEGRATION ET LA TRANSCRIPTION INVERSE... 147
3.2.
E
FFET SUR LA PRODUCTION VIRALE... 149
3.3.
E
FFET SUR LA SYNTHESE DUP
R55
GAG ET LA MATURATION DE LA P24 ... 151
3.4.
A
CTIVITE DE LA PROTEASE VIRALE... 152
4.
CONCLUSIONS ET DISCUSSIONS ... 155
4.1.
C
ARACTERISTIQUES DE L’IN
E85A ... 155
4.2.
E
FFET DE LA MUTATIONE85A
SUR L’
INFECTIVITE VIRALE... 155
CONCLUSIONS GENERALES ... 158
2.
PHOSPHORYLATION DE L’INTEGRASE DU VIH-1 ... 160
3.
CARACTERISATION DU MUTANT E85A DE L’IN ... 162
BIBLIOGRAPHIES ... 165
A
APOBEC3G Apolipoprotein B mRNA-Editing enzyme Catalytic polypeptide-like 3G ARNt Acide RiboNucléique de transfert
ARNv Acide RiboNucléique viral ARV Agents antiRétroViraux ASV Avian Sarcoma Virus
ATF4 Activating Transcription Factor 4 ATP Adénosine-5'-triphosphate
B
BSA Bovine Serum Albumine
BAF Barrier to Auto Integration Factor
C
CA Capside
CCD Catalytic Core Domain (Domaine catalityque) CCR CC Chemokine Receptor
CD4 Cluster of Differentiation 4
CHAPS 3-[(3-cholamidopropyl)-dimethylammonio]-1-propanesulfonate CMH Complexe Majeur d'Histocompatibilité
CMV CytoMegaloVirus
CPI Complexe de pré-intégration CRF Circulating Recombinant Form
CTD C-Terminal Domain (Domaine C-terminal) CUL2/VHL Cullin2 / Von Hippel–Lindau
CXCR CXC Chemokine Receptor
D
DB Double Brin
DIS Dimerization Initiation Sequence
DMEM Dulbecco’s modified minimal essential medium DNA-PK DNA-dependent Protein Kinase
DO Densité Optique
DSS Disuccinimidyl Subérate DTT DiThioThréitol
E
EDTA EthyleneDiamineTetraacetic Acid eIF2α Eukaryotic Initiation Factor 2 α
Env Enveloppe
GCN2 General Control Nonderepressed 2 GFP Green Fluorescent Protein
H
HAT Histone Acetyl Transferase HDAC Histone DeACetylase
HDGF Hepatoma-Derived Growth Factor HEK Human Embryonic Kidney
HeLa Henrietta Lacks
HisRD Histidyl tRNA synthétase like
HMGA1 High Mobility Group Chromosomal Protein A1 HRI Heme-Regulated Inhibitor
HSP Heat-Shock Proteins HTH Hélice-Tour-Hélice
HTLV Human T-Lymphotropic Virus
I
IBD Integrase Binding Domain
IN Integrase
INI Integrase Inibitor INI1 Integrase interactor 1
InR Initiator
IPTG Isopropyl β-D-1-ThioGalactopyranoside IRES Internal Ribosome Entry Sequence
J
JNK JuN Kinase
K
KAP1 Kidney Androgen regulated Protein 1
L
LAV Lymphadenopathy-Associated Virus LEDGF Lens Epithelium-Derived Growth Factor LT CD Lymphocyte T CD
LTR Long Terminal Repeat
M
MA Matrice
MAP Microtubule Associated Protein MAPK Mitogen-Activated Protein Kinase MEF Mouse Embryonic Fibroblasts MOI Multiplicity Of Infection
MP Membrane plasmique
MLV Moloney Murine Leukemia Virus
N
NCINI Non-Catalytic site Integrase Inhibitor (inhibiteurs non catalytiques de l’IN) NED NTD Extension Domain (domaine extension N-terminal)
Nef Negative factor NES Nuclear Export Signal
NFAT Nuclear factor of Activated T-cells NF-κB Nuclear Factor-kappa B
NLS Nuclear Localisation Signal
NNRTI Non Nucleoside Reverse Transcritase Inhibitor
NPC Nuclear Pore Complex (Complexe de pores nucléaires) NRTI Nucleoside Reverse Transcritase Inhibitor
NTD N-terminal Domain
O
ODN Oligonucléotide
OMS Organisation Mondiale de la Santé
ORF Open Reading Frame (Cadre ouvert de lecture)
P
PBS Primer Binding Site PEG PolyEthylène Glycol
PERK Protein kinase-like ER Kinase PFV Prototype Foamy Virus PI Protease Inihibitor PKR Proteine Kinase R PPT PolyPurine Tract
PR Protéase
P-TEFb Positive Transcription Elongation Factor b PTM PostTranslational Modification
PWWP Pro-Trp-Trp-Pro motif
R
RE Réticulum Endoplasmique Rev Regulator of Virion
RRE Rev Response Element RSV Rous Sarcoma Virus RT Reverse transcritase
RTC Reverse Transcription Complex (Complexe de transcription inverse) RT-PCR Reverse Transcription Polymerase Chain Reaction
RMN Résonance Magnétique Nucléaire RWD RING finger WD Repeat
S
SDS Sodium Dodécyl Sulfate SH3 Src Homology Domain 3
SIDA Syndrome de l’ImmunoDéficience Acquise siRNA Small interfering RNA
SV Sindbis Virus
SVF Sérum de Veau Fœtal
T
Tat Transactivateur de la transcription TAR Transactivating-responsive region TD Targeting Domain
TCA TriChloroacetic-Acid TNPO3 Transportine 3
TRIM5 Tripartite Motif-containing protein 5 TRN-SR2 Transportine-SR2
U
Ub Ubiquitine
URF Unique Recombinant Forms UV UltraViolet
V
VBP1 Von Hippel-Lindau Binding Protein 1
VIH Virus de l'Immunodéficience Humaine (HIV) VIS Virus de l'Immunodéficience Simienne VPL Virus-Like Particle
Vpr Viral protein r
VSV Virus de la Stomatite Vésiculeuse
Vul3/VHL complexe ubiquitine ligase cullin 2/ von hippel lindau
« Chacun d’entre nous est un génie. Mais si vous jugez un poisson à sa
capacité à grimper un arbre, il va croire toute sa vie qu’il est stupide »
Albert Einstein
L’infection par le VIH-1
Introduction
1. L’infection par le VIH-1
1.1.
Le syndrome de l’immunodéficience acquise ou SIDA
Le syndrome de l’immunodéficience acquise est une maladie virale s’accompagnant de la destruction des cellules du système immunitaire. Il existe plusieurs rétrovirus responsables du SIDA, chacun infectant une espèce particulière. Le plus connu, le virus de l'immunodéficience humaine (VIH), infecte l’homme.
a.
HistoriqueLe 5 juin 1981, le docteur Michael S. Gottlieb et son équipe décrivent dans le Morbidity and
Mortality Weekly Report cinq patients atteints de grave pneumonie à Pneumocystis carinii. Le
mois suivant, le New York Times se fait l’écho de 41 nouveaux cas de pneumocystose à New York et en Californie. Très vite d’autres cas intriguent les chercheurs, des patients homosexuels atteints d’une tumeur de la peau appelée sarcome de Kaposi. Points communs entre tous ces patients : jeunes, homosexuels et présentant un déficit immunitaire et plus particulièrement en lymphocytes T CD4+ (LT CD4+). Cette maladie sera alors surnommée le « cancer gay ».
Plusieurs arguments épidémiologiques évoquaient la possibilité d’un agent infectieux transmissible par voie sexuelle et/ou sanguine. Deux équipes de chercheurs l’une à l’Institut Pasteur à Paris et l’autre au National Cancer Institut aux Etats-Unis travaillent sur l’identification de l’agent pathogène. Le 4 janvier 1983, un prélèvement de ganglion lymphatique provenant d’un patient homosexuel parisien appelé BRU est confié à l’équipe de Luc Montagnier pour être analysé. Une fois les ganglions mis en culture, Françoise Barré-Sinoussi démontre la présence d’une activité de transcription inverse. Cette activité est la preuve de la présence d’un rétrovirus. Ce nouveau virus sera appelé lymphadenopathy-associated virus (LAV) [1]. De l’autre côté de l’Atlantique, la ministre américaine, Margaret Heckler, annonce que l’équipe de Gallo a identifié l'agent causal du SIDA, un rétrovirus baptisé HTLV-III [2]. En 1985, les résultats de séquençage des deux virus LAV et HTVL-III ont permis de montrer que ces deux virus ne forment qu’un seul et unique virus. En 1986, une commission a décidé de renommer le virus isolé par VIH pour virus de
l’immunodéficience humaine. En 2008, Luc Montagnier ainsi que Françoise Barré-Sinoussi reçurent le prix Nobel de médecine pour avoir identifié l’agent responsable du SIDA.
b.
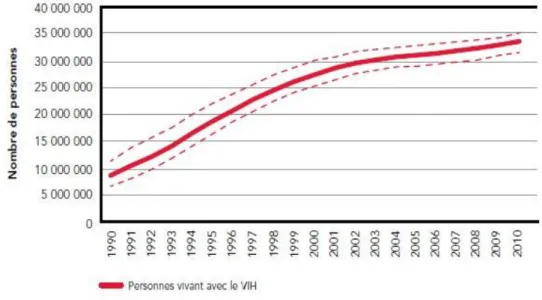
Epidémiologie : situation actuelle et tendanceDepuis le début de la pandémie, plus de 60 millions de personnes ont été infectées par le VIH et près de 30 millions sont décédées des causes de la maladie. D’après le rapport ONUSIDA de septembre 2013, 35,3 millions de personnes sont séropositives en 2012 et le nombre de personnes nouvellement infectées est de 2.3 millions par an. L’Afrique subsaharienne est la région la plus touchée. Elle concentre 69% des personnes vivant avec le VIH dans le monde (figure 1). L’épidémie après avoir connu un pic dans les années 1990, semble s’être stabilisée dans la plu part des régions du monde et ceci notamment grâce à la mise en place de thérapies antirétrovirales efficaces (figure 2).
Figure 1. Répartition des personnes (adultes et enfants) vivant avec le VIH en 2012 (rapport de
l’ONUSIDA 2013). Environ 34 millions de personnes vivent avec le SIDA. La région la plus touchée reste l’Afrique. En effet 70 % des personnes infectées se trouvent dans cette région.
L’infection par le VIH-1
Figure 2. Evolution du nombre de personnes vivant avec le VIH de 1990 à 2010 (rapport de
l’ONUSIDA 2011). Le taux annuel de nouvelles infections a chuté de 21% entre 1997 et 2010, selon l’Onusida. Cependant, le nombre de personnes vivant avec le SIDA n’a jamais été aussi important ces dernières, en raison d’un meilleur accès aux traitements.
c.
Variabilité génétique du VIH-1Le virus de l’immunodéficience humaine appartient à la famille des rétrovirus et à la sous famille des lentivirus. Les lentivirus sont des virus qui provoquent des maladies à évolution lente et qui sont cytopathogènes en culture. Deux types de virus responsables du SIDA ont été identifiés; le VIH-1 répandu dans l’ensemble des continents et le VIH-2 surtout présent en Afrique de l’ouest. Des virus apparentés tel que le SIV (Simian immunodeficiency virus) ont été détectés chez les singes. Une analyse comparative entre le VIH et le SIV a révélé une proximité génétique entre le VIH-1 et le SIV du gorille d’une part, et entre le VIH-2 et le SIV du mangabey d’autre part [3]. Sur la base des distances génétiques entre les VIH-1 retrouvés chez les patients une classification en quatre groupes distincts a été établie : les groupes M, N, O et P (figure 3). Le groupe M (majoritaire) est responsable de la pandémie et regroupe 6 sous types non recombinants (A, B, C, D, F, G, H, J et K), et de nombreux Circulating Recombinant Forms (CRFs) et Unique Recombinant Forms (URFs). Les formes CRFs et URFs proviennent de phénomènes de recombinaison génétique chez des sujets co-infectés par différents types de VIH-1 [4]. En France, la présence du sous-type B prédomine mais la circulation d’autres sous-sous-types ou de recombinant augmente tel que le CRF02_AG (figure 4). Les groupes N, O et P sont retrouvés principalement au Cameroun et sont beaucoup plus rares [5].
Cette diversité génétique entraîne l’émergence perpétuelle de nouvelles formes et peut aboutir à la sélection de formes plus adaptées. Bien entendu, ce phénomène de variabilité constitue un frein à l’élaboration d’un vaccin efficace.
Figure 3. Arbre phylogénétique du VIH-1 et du SIV basé sur les séquences des enzymes réplicatives rétrovirales (transcritase inverse (RT)) [4] SIVcpz correspond aux virus retrouvé chez
le champanzé tandis que le SIVgor correspond à celui retrouvé chez le gorille.
L’infection par le VIH-1
d.
L’ origine du VIHSelon des analyses phylogéniques, il est admis que l’origine du VIH serait simienne (figure 3). Différentes hypothèses ont été proposées pour expliquer la transmission du singe à l’homme. Les hypothèses les plus communément admises sont une contamination lors des parties de chasse via une morsure par un singe infecté, une écorchure consécutive au dépeçage de la proie ou à l’ingestion d’une viande insuffisamment cuite.
e.
Physiopathologie de l’infection par le VIH-1Le VIH infecte les LT CD4+ et les cellules présentatrices d’antigène telles que les macrophages et les cellules dendritiques, induisant un déficit profond de l’immunité cellulaire. La physiopathologie de l’infection en absence de traitement se déroule classiquement en trois phases: la primo-infection, la phase asymptomatique et la phase d’apparition des symptômes du SIDA (figure 5).
La primo-infection est la phase précoce survenant durant les premières semaines après l’infection. Les premiers symptômes sont peu spécifiques et sont apparentés à un syndrome pseudo-grippal. La fièvre est présente chez 90 % des sujets et d’autres symptômes tels que la dysphagie, les céphalées, les myalgies, l’asthénie et l’amaigrissement sont également fréquents. Cette phase se caractérise par une réplication virale intense, avec une charge virale plasmatique pouvant atteindre fréquemment 106 copies d’ARN viral par millilitre de sang. Cette virémie élevée est associée à une diminution de la quantité des LT CD4+ circulants qui reste faible en l’absence de traitement antirétroviral.
La phase de latence ou asymptomatique peut durer de quelques mois à quelques années. La réplication du virus diminue et se stabilise à un niveau plus ou moins élevé en fonction des patients.
Sans traitement, la phase SIDA apparait en général dans les 10 ans après l’infection et se caractérise par l’apparition des premiers signes cliniques de l’infection. La phase SIDA est déclarée lorsque la virémie est élevée, le taux de LT CD4+ est bas et enfin lors de l’apparition de maladies opportunistes. Le déficit immunitaire est sévère favorisant l’apparition de maladies opportunistes et de tumeurs provoquant le décès des sujets.
Primo-infection Phase asymptomatique SIDA Figure 5. Phases caractéristiques de l’infection du VIH-1 en absence de traitement antivirale http://georges .dolisi.free.fr/Physiopatho logie/SIDA_evolution.htm
f.
DiagnosticL’approche diagnostique la plus accessible est une méthode indirecte. Ce test de dépistage est basé sur la mise en évidence de la présence des anticorps anti-VIH par ELISA dans le sérum ou dans le plasma des individus. Si ce test est négatif, l’individu est considéré comme séronégatif. Si le résultat est positif, un test de confirmation est réalisé. A partir du même prélèvement, la présence d’anticorps contre les protéines virales du VIH-1 est détectée par Western Blot à l’aide de bandelettes commerciales. Selon les critères de l’organisation mondiale de la Santé (OMS), ce test est considéré positif si au moins deux des glycoprotéines d’enveloppe gp41 et gp120 ou la forme précurseur gp160 sont révélées. Dans le cas de primo-infection, la recherche de la présence de l’antigène p24 ou de l’ARN viral du VIH-1 peut s’avérer nécessaire.
1.2.
Le VIH-1
a.
ClassificationLe VIH-1 appartient à la famille des rétrovirus. Ce sont des virus enveloppés qui se caractérisent par la présence de deux copies d’ARN simple brin de polarité positive. Les rétrovirus possèdent une enzyme spécifique la transcritase inverse (RT) qui catalyse la synthèse d’ADN double brin à partir de l’ARN.
Les rétrovirus peuvent être endogènes ou exogènes. Les rétrovirus endogènes résultent de l’intégration d’ADN viral lors d’infections survenues il y a des millions d’années. Ils sont donc transmis de manière verticale au sein d’une population donnée et sont devenus des séquences
L’infection par le VIH-1
stables dans le génome d’un organisme. Les rétrovirus endogène humain représentent 8 % du génome. Les rétrovirus exogènes quant à eux ne sont pas présents naturellement dans l'organisme.
La famille des rétrovirus exogènes est divisée en deux sous-familles: les Orthoretovirinae et les
Spumaretovirinae (figure 6). Les Spumaretovirinae sont composés d’un seul genre les Spumavirus
contenant le virus de Foamy Humain non pathogène. La sous-famille d’Orthoretovirinae est constituée de 6 genres : les Alpharetrovirus (ex :Virus du Sarcome du Rous), les Betaretrovirus (ex :Virus de la Tumeur Mammaire de la Souris), les Gammaretrovirus (ex :Virus de la Leucémie Murine), les Deltaretrovirus (ex : Virus-T lymphotropique Humain), les Epsilonretrovirus (ex : Walley Dermal Sarcoma Virus), les Lentivirus (ex : Virus de l’Immunodéficience Humaine) [6].
Figure 6. Analyse phylogénique des régions conservées des gènes de polymérases des rétrovirus. basé sur
l’alignement des séquences partielles de la transcritase inverse [6].
b.
Structure du virusLes rétrovirus forment une particule sphérique d’environ 80 à 100 nm de diamètre. La membrane virale est composée de lipides issus des cellules productrices des virions et de glycoprotéines virales nécessaires à l’attachement et l’entrée dans la cellule cible.
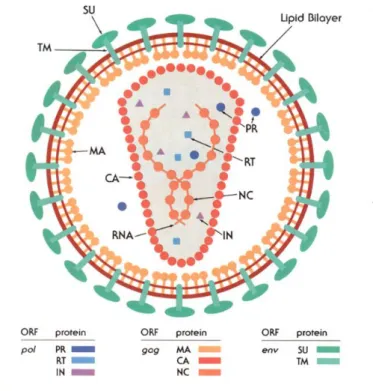
Au niveau de la surface interne de l’enveloppe, la matrice (MA) forme un réseau stabilisant la particule virale. Au sein de cette enveloppe se trouve le « cœur » ou core viral composé d’environ 1500 copies de capside (CA) contenant l’ARN viral, la nucléocapside (NC) ainsi que les enzymes virales la protéase (PR), la RT et l’intégrase (IN) (figure 7).
Figure 7. Structure schématique d’une particule mature. http://www.chm.tcu.edu/reinecke.html
c.
Le génome viralLe génome du VIH-1, une fois intégré, est appelé provirus, il a une longueur d’environ 9200 nucléotides. Les extrémités du provirus sont flanquées par des séquences répétées nommées LTRs (long terminal repeats) qui se divisent en trois éléments : U3, R et U5. Ces régions contiennent des signaux importants pour l’intégration, la transcription, la transcription inverse et l’encapsidation du génome viral.
L’ADN proviral code pour neuf gènes qui peuvent être classés en 3 groupes fonctionnels : les gènes codant pour les protéines structurales et non structurales Gag, Pol et Env, les gènes codant pour les protéines régulatrices Tat et Rev et enfin les gènes codant pour les protéines accessoires Vpu, Vpr, Vif, et Nef [7] (figure 8).
L’ORF gag code pour une protéine précurseur Pr55gag de 55 kDa qui est clivée par la protéase virale en 4 protéines : la matrice MA, la capside CA, la nucléocapside NC, le peptide C-terminal P6, ainsi que deux spacers : le peptide N-terminal P2 ainsi que le peptide C-terminal P1.
L’ORF pol code trois enzymes virales : la protéase (PR), la reverse transcritase (RT) et l’intégrase (IN) qui sont issus du clivage du précurseurs Pr150 gag-pol.
Et enfin le gène env code pour une polyprotéine d’enveloppe gp160 synthétisée au niveau du réticulum endoplasmique. Cette polyprotéine est glycosylée puis clivée par une protéase cellulaire
L’infection par le VIH-1
gag
pol
env
U3 R U5 U3R U5
gp160
en deux protéines, la glycoprotéine de surface (SU) gp120 et la glycoprotéine transmembranaire (TM) gp41.Figure 8. Organisation génomique du VIH-1 et représentation schématique des précurseurs protéiques et des protéines issues de leur maturation. Adapté de http://hire.uohbif.in/.
d.
Protéines viralesLes glycoprotéines d’enveloppe
Les glycoprotéines de surface de l’enveloppe (Env) du VIH interagissent avec le récepteur cellulaire CD4 et les corécepteurs CCR5 et CXCR4 permettant l’entrée du virus dans la cellule cible. A la surface de la membrane virale, gp120 et gp41 s’assoxient sous forme de trimère. Le nombre de trimère par virion peut varier entre 10 et 100.
Gp120 est composée d’un domaine interne qui interagit avec la sous unité gp41 et d’un domaine externe hautement glycosylé interagissant avec le récepteur CD4 et les corécepteurs [8]. Gp120 contient cinq domaines relativement conservés (C1-C5) et cinq boucles variables (V1-V5). Ces boucles variables jouent un rôle dans l’échappement au système immunitaire et dans la liaison aux corécepteurs.
La protéine gp41, quant à elle, est constituée de trois domaines majeurs, le domaine extracellulaire, le domaine transmembranaire et le domaine cytoplasmique [9]. Elle est impliquée dans la fusion entre les membranes cellulaires et virales.
Les protéines structurales La matrice (p17, MA)
La matrice forme le domaine N-terminal du précurseur Pr55gag et elle recouvre la surface interne de l’enveloppe virale. La structure tridimensionnelle de la MA a été déterminée par spectrométrie résonance magnétique nucléaire (RMN) et par cristallographie aux rayons X [10, 11]. Ces résultats ont permis de montrer que la MA s’organise en tétramère. Cette protéine est composée de trois séquences de localisation, une de transport à la membrane, une de localisation nucléaire et une d’export nucléaire [12]. Le signal de transport à la membrane est en N-terminal et est myristoylé. Ce signal joue un rôle dans l’assemblage des particules virales en permettant la liaison de la MA à la membrane et donc le recrutement correct du précurseur Pr55gag au site d’assemblage. La matrice est donc une protéine multifonctionnelle qui est impliquée dans la régulation des étapes précoces et tardives du cycle.
La capside (p24, CA)
Les protéines de capside s’assemblent sous forme d’hexamère pour former le core viral qui renferme l’information génétique (figure 9 (a)). L’interaction entre les molécules de CA au sein de chaque hexamère est permise grâce au domaine N-terminal de la CA. Tandis que le domaine C-terminal est impliqué dans l’interaction entre les hexamères permettant la formation de la structure cylindrique (figure 9 (b)). La capside a un rôle surtout lors des étapes précoces du cycles tel que la reverse transcription et l’import nucléaire mais également lors des étapes tardives comme l’assemblage [13].
L’infection par le VIH-1
Figure 9. Structure de la capside de VIH-1 et d’un hexamère de capside [14]. (a) Un modèle
moléculaire de la capside du VIH-1 avec en orange les hexamères de CA et en beige les pentamères. (b) La structure d’un hexamère de CA (PDB : 3H47).
La nucléocapside (p7, NC)
La nucléocapside est une petite protéine fortement basique caractérisée par la présence de deux domaines à doigts de zinc CCHC connectés par un peptide de liaison flexible (figure 10 (a)) [15]. La structure de la NC a été résolue par RMN (figure 10 (b)) [16].
La présence du motif en doigt de Zinc confère à la NC une forte affinité pour les acides nucléiques. Elle joue ainsi un rôle de protéine chaperonne et de protection de l’ARN viral [17]. Ces propriétés de protéines chaperonnes sont importantes lors de l’assemblage des virus pour l’internalisation et la dimérisation de l’ARN viral. La NC est également impliquée lors de l’étape de transcription inverse [18].
Figure 10. Séquence et structure de la nucléocapside (a) La séquence de la protéine NC avec ses
deux doigts de zinc. Les lettres vides correspondent aux résidus en interaction avec l’ARN viral. Les étoiles indiquent les résidus impliqués dans les liaisons hydrogènes intermoléculaires [15]. (b) Structure de la nucléocapside du VIH-1 complexée avec l’ARN Ψ de SL3 (en blanc) du VIH-1. Les doigts de zinc sont représentés par les sphères grises [16].
La protéine p6
Située en C-terminal du Pr55, cette petite protéine de 51 acides aminés est importante pour l’incorporation de la protéine Vpr durant l’assemblage viral. Elle facilite également la formation de nouveau virus [19].
Les protéines non structurales La protéase (p12, PR)
La protéase appartient à la famille des aspartates protéases. La forme active de la protéase est dimérique. Son site actif se situe à l’interface des dimères et est constitué d’une triade catalytique Asp25-Thr26-Gly27, conservée au sein de la famille des protéases aspartiques. Sa structure a été obtenue en 1989 [20] (figure 11). La protéase catalyse la maturation des précurseurs protéiques nécessaires à la production de particules virales matures et infectieuses. En plus de cliver les précurseurs viraux, la PR clive également des protéines cellulaires. La protéolyse par la PR de protéines cellulaires telles que Bcl2 ou encore la procaspase 8 pourrait être responsable du processus de mort cellulaire entraînant la déplétion des LT CD4+ [21].
Figure 11. Structure cristallographique d’un homodimère de protéases Les résidus Asp 25 de
chaque monomère sont représentés en rouge [22] (PDB : 3HVP)
La transcritase inverse (p51/66, RT)
La RT possède trois activités enzymatiques, une activité ADN polymérase ARN dépendante et ADN dépendante et une activité RNase H. Ces trois activités sont nécessaires à l’étape de transcription inverse permettant la synthèse d’un ADN double brin à partir de l’ARN viral. La RT est un hétérodimère composé de deux sous-unités, p66 et p51. La sous-unité p51 est issue d’un clivage
L’infection par le VIH-1
par la PR du dimère p66/p66. C’est la sous-unité p66 qui contient le site actif, tandis que la sous unité p51 a un rôle structural.
Dans [23] et [24], la structure 3D de la RT a permis de montrer que la sous-unité p66 est composée de deux domaines spatialement distincts (polymérase et RNAse H), tandis que la sous-unité p51 possède seulement le domaine DNA polymérase (figure 12). Le domaine polymérase est constitué de quatre sous domaines : les doigts (résidus 1-85 et 118-155), la paume (résidus 86-117 et 156-236), le pouce (237-318) et le domaine de connexion (319-426). Chacun de ces sous-domaines a un rôle unique dans l’incorporation des nucléotides au cours la synthèse de l’ADN [22]. Le site actif de la polymérase dans la sous-unité p66 est composé de trois arginines hautement conservées en position 110, 185 et 186 de la paume de p66 [25]. Dans la sous-unité p51 ces résidus sont présents mais dans une structure qui les rendent non accessibles.
La sous-unité p51 permet de stabiliser l’hétérodimère p66/p51 mais participe aussi à la liaison du primer tRNA servant d’amorce pour la transcription inverse. La p51 possède également les quatre sous-domaines mais la position relative de chacun est différente entre p66 et p51.
Figure 12. Représentation en ruban du complexe RT avec un acide nucléique. [22]. Le monomère
p51 est représenté en gris. Le monomère p66 est en : bleu (doigts), en vert (pouce), rouge (paume) et en rose (domaine RNAseH). L’acide nucléique matrice est en jaune et l’amorce en orange.
L’intégrase (p32, IN)
L’IN est une protéine de la famille des polynucléotidyl transférases. Cette enzyme est composée de trois domaines : N-terminal, core catalytique et C-terminal. Elle catalyse l’étape d’intégration et son oligomérisation est nécessaire à son activité. Cette protéine étant l’objet d’étude principal de ma thèse, sa structure ainsi que son rôle seront plus approfondis dans la partie B : Intégrase.
Les protéines régulatrices Tat
Tat est une petite protéine de 80 à 103 résidus en fonction des isolats viraux. Il est supposé que Tat adopte différentes conformations en fonction de ses interactions avec ses différents partenaires. Le rôle principal de Tat est de contribuer à la transactivation de la transcription des gènes viraux et cellulaires [26].
Rev
Rev est une protéine de liaison à l’ARN possédant un signal de localisation nucléaire et un autre d’export nucléaire. Rev se fixe sur la séquence RRE présente au niveau des ARNm viraux mono épissés ou non épissés, cette interaction stimule le transport nucléocytoplasmique de ces ARNm. Rev est également impliqué lors de la polyadénylation des ARNm, la traduction, l’encapsidation et la stabilité de l’ARN [27] .
Les protéines accessoires
Quatre protéines accessoires sont issues du génome du VIH-1 : Vpu, Vpr, Vif, Nef. La présence ou l’absence de ces protéines peut interférer sur la vitesse et la sévérité de l’infection virale.
Vpu
Vpu est une protéine membranaire qui active le bourgeonnement de nouveaux virions [28]. De plus, suite à l’interaction avec le récepteur CD4 dans le réticulum endoplasmique, Vpu induit l’ubiquitinylation de CD4 qui sera alors dégradé par le protéasome [29].
Vpr
La protéine Vpr est très conservée entre le VIH-1, le VIH-2 et le SIV [28]. Vpr est impliqué dans l’import nucléaire du complexe de pré-intégration (CPI). En effet, il a été montré que Vpr est important pour la translocation nucléaire du CPI dans des macrophages ne se divisant pas [30].
L’infection par le VIH-1
Etapes précoces Etapes tardives
1 2 3 4 5 5 6 7
6
7
8
Vpr est également impliqué dans le passage de la phase S à G2 lors du cycle cellulaire, dans la régulation de l’apoptose et enfin dans la transactivation des LTR viraux.
Vif
Vif est une protéine hautement exprimée dans les cellules infectées. Il a été montré que Vif interagit avec la cytosine désaminase APOBEC3G qui est un facteur cellulaire de restriction à l’infection par le VIH-1 dans les cellules non permissives [31]. Vif en se liant à APOBEC3G induit sa dégradation par le protéasome permettant ainsi l’infection dans des cellules non permissives [32].
Nef
Nef est hautement conservée au sein des lentivirus de primate. Ses principales fonction est d’inhiber l’expression de plusieurs récepteurs cellulaires : CD4, CD8, CD28, le CMH-I et le CMH-II ce qui modifie la transduction de signaux cellulaires [28].
1.3.
Le cycle réplicatif
Le cycle réplicatif du VIH-1 se déroule en plusieurs étapes divisées en deux parties : les étapes précoces (figure 13 de 1 à 4) et les étapes tardives (figure 13 de 5 à 7).
a.
Etapes précocesL’entrée virale.
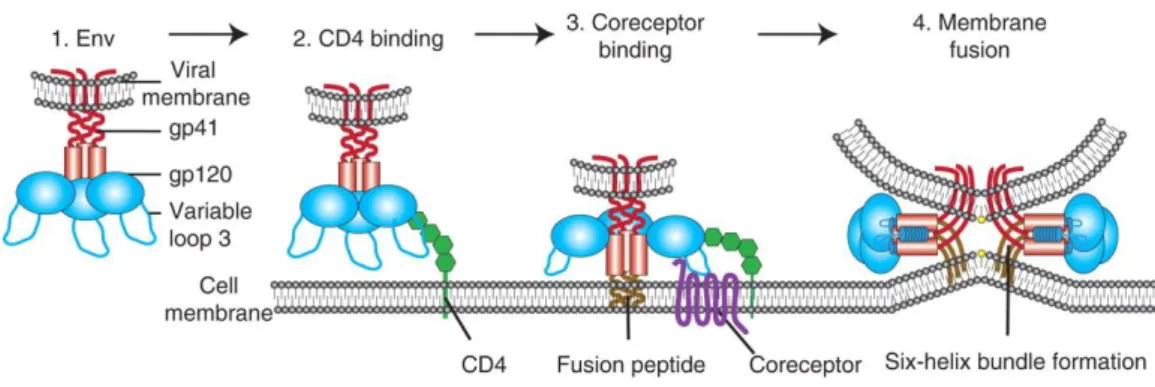
La première étape du cycle est l’entrée du virus dans la cellule hôte (figure 13 - étape 1). Cette étape commence par une interaction entre le virus et la cellule, suivie d’une fusion entre la membrane virale et la membrane cellulaire libérant le core viral dans le cytoplasme. L’entrée virale peut être divisée en plusieurs phases (figure 14).
Dans un premier temps a lieu une interaction entre la protéine virale gp120 et le récepteur CD4 [34]. Cette liaison entraîne un réarrangement des domaines variables de gp120 essentiel à l’étape suivante qui est l’interaction avec le corécepteur transmembranaire aux chémokines. Deux types de corécepteurs peuvent être impliqués : CCR5 et CXCR4. Les virus sont ainsi classés VIH X4, VIH R5 ou VIH X4/R5 en fonction du type de corécepteur qu’ils utilisent. En général seuls les virus R5 et X4/R5 peuvent être transmis entre les individus. Plus tard, lors de l’infection les virus X4 seront séléctionés, c’est ce que l’on nomme le tropisme.
La dernière étape de l’entrée est la fusion membranaire. La liaison avec le corécepteur induit l’exposition d’un peptide de fusion de gp41 qui s’insère dans la membrane de la cellule cible. La fusion complète se produit alors entre les membranes virale et cellulaire libérant la capside virale dans le cytoplasme [35]. Ces interactions entre le virus et les récepteurs cellulaires activent de multiples voies de signalisation intracellulaires impliquées en autre dans le réarrangement du cytosquelette, l’activation de la transcription (activation de NF-ΚB et NFAT), dans la survie cellulaire [36]
Figure 14. Etapes d’entrée du VIH-1 dans la cellule-cible. 1. L’entrée nécessite les protéines Env
comprenant les sous unités gp120 (bleus) et gp41 (rouges). 2. La liaison entre gp120 et CD4. 3. Le changement conformationnel de gp120 permettant son rapprochement avec le corécepteur. 4. Ce qui induit un changement de conformation permettant au peptide fusion de gp41 de s’insérer dans la membrane de la cellule induisant la fusion des deux membranes [35].
L’infection par le VIH-1
La transcription inverse
La transcription inverse consiste en une série de réactions enzymatiques permettant la synthèse d’un ADN double brin à partir de l’ARN viral (figure 13-étape 2). Cette étape est catalysée par la RT et a lieu dans le cytoplasme au sein d’un complexe appelé le complexe de transcription inverse (RTC) (figure 15).
Pour initier la transcription inverse, la RT du VIH-1 a besoin d’une matrice et d’une amorce qui est un ARNt cellulaire, l’ARNtlys3. Cet ARNtlys3 se fixe au niveau de la séquence PBS (primer binding site) située en 5’ de l’ARN viral (ARNv) et sert d’amorce à la RT pour la synthèse de l’ADN négatif jusqu’à l’extrémité 5’ de l’ARN. Un duplex ADN/ARN est ainsi généré [37]. L’activité RNase H de la RT dégrade l’extrémité 5’ de l’ARNv, exposant l’ADN négatif nouvellement synthétisé. Cette région d’ADN est alors transférée à l’extrémité 3’ de l’ARNv. Ce premier saut de brin a lieu grâce à la présence des séquences R répétées aux extrémités de l’ARNv. La synthèse de l’ADN négatif peut alors se poursuivre sur tout le brin, puis la RNase H dégrade le reste de l’ARN excepté les séquences PolyPurine Tract (PPT) riches en purines situées en 3’ et dans la région centrale. Ces régions PPT servent d’amorce pour la synthèse du brin positif d’ADN et seront éliminées au cours de l'élongation. Un second saut de brin permet l’hybridation des séquences PBS des deux brins d’ADN. La fin de la synthèse aboutit à un ADN double brin linéaire comprenant à ses extrémités les LTR composés des séquences U3/R/U5. Ces extrémités LTR étant le substrat pour l’étape d’intégration, la spécificité de clivage des extrémités U3 en 5’ et U5 en 3’ par la RNase H est importante pour la suite.
La transcription inverse dans la cellule se produit au sein du RTC en présence des protéines virales MA, CA, RT, Vpr et IN. Ces protéines virales pourront avoir un rôle de cofacteur au cours de cette étape. Il a notamment été montré que la NC est un régulateur important de la transcription inverse (pour une revue voir [38]).
Figure 15. Etapes de la transcription inverse du VIH-1 (figure issue de la revue [39])
La décapsidation
Suite à la fusion des membranes cellulaires et virales, la capside virale est libérée dans le cytoplasme (figure 13 – étape 3). A l’heure actuelle, les données tendent à dire que la décapsidation a lieu après l’entrée et avant l’import nucléaire, mais les avis divergent quant au moment et au lieu précis de cette étape.
Une des hypothèses est que le désassemblage de la capside a lieu immédiatement après la fusion à proximité de la membrane plasmique, sachant qu’il n’est pas exclu que ce désassemblage soit partiel [40]. La décapsidation serait alors déclenchée par un changement soudain d’environnement lié à l’entrée du virus dans la cellule hôte.
Dans un autre modèle, la décapsidation est séquentielle au cours du transport vers le noyau. Dans ce cas, l’activation du désassemblage serait due à des changements successifs de l’environnement cellulaire et à des interactions avec différents cofacteurs cellulaires.
Un troisième modèle propose que la décapsidation à lieu à proximité du pore nucléaire lorsque la transcription inverse est terminée [41]. La capside intacte offrirait un environnement propice à la transcription inverse avec une concentration élevée de RT tout en permettant des échanges de
L’infection par le VIH-1
petites molécules telles que les nucléotides [42]. Dans ce modèle, une transcription inverse complète est nécessaire pour induire la décapsidation.
De nombreuses études montrent que si la décapsidation a lieu trop tôt, la transcription inverse est inhibée. En effet, un facteur de restriction cellulaire TRIM5α est connu pour cibler la CA au protéasome. TRIM5α agit dans l’heure suivant l’infection et entraîne un désassemblage prématuré inhibant la RT. De plus des mutants de CA induisant une instabilité de la capside virale provoquent une décapsidation prématurée et une transcription inverse abortive. Ces données prouvent que le core viral doit rester plus ou moins intact au cours de la transcription inverse [13].
Le transport intracellulaire et l’import nucléaire
Le cytoplasme est un milieu très visqueux ne permettant pas le déplacement des macromolécules et notamment des particules virales par simple diffusion. Les virus doivent donc détourner les mécanismes cellulaires pour se déplacer dans le cytoplasme et atteindre le site de réplication. Dans le cas du VIH, il semblerait que le RTC se déplace le long des microtubules pour atteindre la périphérie nucléaire au niveau du centrosome.
Lorsque la transcription inverse est complète le RTC devient le complexe de pré-intégration (CPI). Il a été montré que ce complexe est composée de l’ADNc, de protéines cellulaires et virales (RT, IN, MA, Vpr, CA et NC) même si la composition exacte du CPI reste controversée [19]. Ce CPI une fois transporté à proximité du noyau doit franchir la membrane nucléaire. Le transport via les pores nucléaires (NPCs) est très sélectif, les molécules de diamètre de moins de 39 nm peuvent passer par diffusion passive, tandis que le passage de molécules de diamètre supérieur à 39 nm nécessite un transport actif [43]. Sachant que le CPI est un complexe de grande taille dont le diamètre a été estimé supérieur à 50 nm, son entrée dans le noyau nécessite un transport actif et donc l’interaction avec des protéines carrier spécifiques. Les protéines virales et la structure viral « ADN flap » ont été proposées comme importantes pour l’import nucléaire du CPI [44].
La structure ADN Flap de l’ADN complémentair reverse transcrit est décrit comme impliquée dans l’import nucléaire, cependant la mutation de cette structure ne bloque pas totalement l’entrée du CPI dans le noyau [45].
De plus, les signaux de localisation nucléaires (NLSs) présents dans la MA, dans l’IN et dans Vpr ont été décrits comme importants pour le recrutement de protéines impliquées dans le transport nucléaire [46–48]. Il semblerait que plusieurs mécanismes soient impliqués pour l’import nucléaire du CPI.
L’intégration du génome viral
L’intégration est une étape clef du cycle viral permettant l’insertion stable de l’ADN viral dans le génome cellulaire rendant possible l’expression des gènes viraux et le maintien de l’information génétique lors de la division cellulaire. Cette étape est catalysée par l’IN. Le cofacteur cellulaire le plus étudié de l’intégration est LEDGF/p75. Les différentes étapes de l’intégration seront d’avantages décrites ainsi que les partenaires impliqués dans le la partie 2 intégrase du VIH-1.
b.
Etapes tardivesUne fois intégré, le provirus sert de matrice pour la transcription des ARNm viraux qui seront pour certains traduits en protéines et d’autres seront encapsidés pour devenir les génomes des nouvelles particules virales.
Transcription et export nucléaire des ARNm viraux
Les LTR contiennent des sites de liaisons pour plusieurs facteurs de transcriptions incluant NF-Kb, NFAT, Sp1 et AP1 ainsi que des signaux de polyadénylations. En amont du site d’initiation de la transcription (InR), des signaux types TATA box et des sites de liaison pour SP1 permettent le recrutement correct de l’ARN polymérase II au site d’initiation de la transcription [49]. En 3’ de cette région est présente une structure ARN, l’élément transactivating-responsive region (TAR) sur lequel se fixe la protéine Tat [50]. Tat recrute au niveau de TAR l’activateur de la transcription P-TEFb (positive transcription elongation factor b) constituée des deux sous unités CDK9 et CycT1. L’interaction entre Tat et P-TEFb induit un changement de conformation de CDK9 activant P-TEFb. Une succession de phosphorylations se produit alors et CDK9 phosphoryle le domaine CTD de l’ARN pol II permettant de passer d’un état d’initiation à un état d’élongation de la transcription et donc de finir l’élongation de l’ARN viral [51].
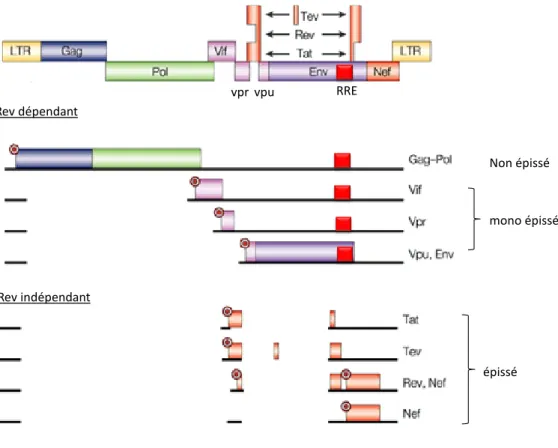
L’ARN polymérase II produit des transcrits ARN coiffés en 5’ et polyadénylés en 3’. Contrairement à beaucoup d’ARNm cellulaires, une partie des transcrits pour le VIH-1 sont non épissés (figure
16). Les ARN multi épissés sont exportés vers le cytoplasme de manière indépendante de Rev via
le système d’export des ARN messagers cellulaires, ce qui permet la production dans le cytoplasme des protéines régulatrices Rev, Nef et Tat [50]. Quant à l’export nucléaire des ARNm non épissés ou mono-épissés, il nécessite la présence de la protéine Rev qui se lie à l’élément Rev Responsive Element (RRE) permettant l’export des ARNm viraux [33]. L’ARNm non épissé, par la
L’infection par le VIH-1
suite servira aussi bien comme matrice pour la traduction ou comme ARN génomique qui sera encapsidé dans les nouveaux virons produits [53].
Figure 16. Transcription du VIH-1. Le génome viral contient plusieurs ORFs codant pour seize
protéines, issus d’au moins dix transcrits. Les lignes noires correspondent aux transcrits épissés et non épissés. Le point rouge correspond au codon d’initiation de la traduction. Adapté de [49].
Traduction
La traduction de l’ARN génomique peut se faire de manière coiffe-dépendante ou Internal Ribosome Entry Sequence (IRES) dépendante.
L’initiation coiffe-dépendante est utilisée communément par les ARNm eucaryotes. Le complexe eIF4F se fixe à la structure coiffe en 5’ de l’ARNm permettant le recrutement de la machinerie de traduction. La sous unité 40S du ribosome procède alors au scan de l’ARNm de 5’ en 3’ jusqu’au codon d’initiation AUG. Mais la traduction coiffe-dépendante est ralentie par la présence de la région 5’UTR de l’ARN viral qui est longue et hautement structurée [54]. L’ARNm gag non épissé ainsi que les ARNm mono épissés partageant le même 5’UTR, tous deux sont donc de mauvais substrats pour la traduction coiffe dépendante.
épissé Non épissé mono épissé vpr vpu RRE Rev dépendant Rev indépendant
Le VIH-1 a donc développé un mécanisme d’initiation de la traduction coiffe-indépendante. Le virus possède plusieurs séquences IRES une en 5’UTR et une autre dans la région codante de gag. Ces IRES permettent le recrutement de la sous unité 40S indépendamment de la liaison du complexe eIF4F à la structure coiffe.
Une des hypothèses serait que les rétrovirus utilisent les deux modes d’initiation, coiffe-dépendant et IRES-coiffe-dépendant, selon les conditions cellulaires pour favoriser la traduction de leurs protéines et leur réplication. En effet la traduction coiffe-dépendante peut être inhibée lors de différents stress cellulaires tels que l’hypoxie, les infections virales, l’apoptose. Il a été montré que dans ces conditions de stress la traduction des ARNm du VIH n’est pas perturbée et se fait de façon IRES-dépendante [55].
L’ARNm génomique entier sera traduit en deux précurseurs le Pr55gag et le Pr160gag-pol. La synthèse du précurseur gag-pol se produit suite à un décalage de lecture en -1. Ce décalage existe dans environ 5% des cas et permet la production d’un précurseur gag-pol pour 20 précurseurs gag. Deux éléments sont essentiels pour ce phénomène. L’héptanucléotide UUUUUUA et une structure tige-boucle situés à environ 200 nucléotides en amont du codon de terminaison [56].
Assemblage et bourgeonnement
La formation de nouvelles particules virales se produit selon plusieurs étapes faisant intervenir le précurseur Gag. Ce processus inclut la liaison de Gag à la membrane plasmique (MP), suivi de sa multimérisation, l’encapsidation de l’ARN génomique et l’incorporation des protéines Env [57]. Le ciblage de Gag à la MP nécessite la présence d’un domaine hautement basique myristilé en N-terminal de la MA.
L’assemblage se poursuit par le bourgeonnement des particules virales à la MP de la cellule. Ce bourgeonnement est initié par l’oligomérisation de Gag et est complété par la machinerie cellulaire incluant notamment le complexe Endosomal Sorting Complex Required for Transport-I et III (ESCRT) ainsi que les facteurs associés (figure 17) [58].
Si le Pr55Gag est suffisant pour la production de Virus-Like Particules (VPLs) immatures, la production de particule mature nécessite l’encapsidation d’ARN et l’incorporation des glycoprotéines Env et du précurseur Gag-Pol. L’encapsidation de l’ARN est possible grâce à une interaction entre la NC et le signal d’encapsidation Psi (Ψ) présent sur l’ARNv en 5’ [57]. Deux copies d’ARNv sont sélectivement encapsidées et sont liées de manière non covalente via une séquence située dans le 5’UTR appelée signal d’initiation de dimérisation (DIS). La dimérisation de l’ARNv est importante pour l’infectivité des particules virales produites [14].
L’infection par le VIH-1
Figure 17. Assemblage et bourgeonnement des virus à la membrane plasmique. [58]
Maturation des particules virales
Les virus immatures non infectieux produits subissent une ultime étape de maturation pour devenir infectieux (figure 18). Cette étape passe par le clivage des précurseurs par la protéase virale. La protéase fonctionne comme un homodimère. Suite à l’interaction entre deux molécules gag-pol, le dimère de PR formé est instable et faiblement actif. Dû à cette faible activité, le premier clivage effectué par la PR est intramoléculaire permettant ainsi la libération du domaine N-terminal en amont de la PR [59]. Ceci augmente la stabilité de la PR ainsi que son activité favorisant les clivages intermoléculaires. Les clivages réalisés par la protéase se déroulent dans un ordre précis afin de former des virions infectieux (figure 19). En effet des mutations de la PR altérant l’ordre de clivage inhibent de manière drastique l’infection virale [60].
Une activation prématurée de la PR comme lors de la surexpression du précurseur gag-pol entraîne un processing précoce conduisant à une diminution de la production virale. D’autre part, l’expression d’une PR inactive aboutit à la production de virus non infectieux démontrant que des protéases cellulaires ne peuvent pas prendre le relais. L’activation de la PR est donc une étape critique et qui doit être hautement régulée.
L’assemblage, le bourgeonnement et la maturation sont étroitement liés. La cinétique de ces évènements n’est pas résolue à l’heure actuelle. Suite à l’initiation de l’assemblage à la MP, des
![Figure 4. Répartition des différents sous-types et CRFs dans le monde de 2004 à 2007 [5]](https://thumb-eu.123doks.com/thumbv2/123doknet/14741185.755098/30.892.182.698.690.1055/figure-répartition-types-crfs-monde.webp)
![Figure 9. Structure de la capside de VIH-1 et d’un hexamère de capside [14]](https://thumb-eu.123doks.com/thumbv2/123doknet/14741185.755098/37.892.293.630.64.344/figure-structure-la-capside-vih-un-hexamère-capside.webp)
![Figure 12. Représentation en ruban du complexe RT avec un acide nucléique. [22]. Le monomère p51 est représenté en gris](https://thumb-eu.123doks.com/thumbv2/123doknet/14741185.755098/39.892.217.652.573.893/figure-représentation-ruban-complexe-acide-nucléique-monomère-représenté.webp)
![Figure 13. Représentation schématique du cycle réplicatif du VIH-1 [33]](https://thumb-eu.123doks.com/thumbv2/123doknet/14741185.755098/41.892.111.769.687.1098/figure-représentation-schématique-du-cycle-réplicatif-du-vih.webp)
![Figure 15. Etapes de la transcription inverse du VIH-1 (figure issue de la revue [39])](https://thumb-eu.123doks.com/thumbv2/123doknet/14741185.755098/44.892.77.827.70.590/figure-etapes-transcription-inverse-vih-figure-issue-revue.webp)
![Figure 19. Clivage du précurseur gag. Processus séquentiel de clivage de gag par la protéase virale [58]](https://thumb-eu.123doks.com/thumbv2/123doknet/14741185.755098/50.892.227.602.495.844/figure-clivage-précurseur-processus-séquentiel-clivage-protéase-virale.webp)