Développement d’un bioessai moléculaire pour le
diagnostic des sept principaux gènes d’avirulence chez
Phytophthora sojae
Mémoire
Chloé Dussault-Benoit
Maîtrise en biologie végétale - avec mémoire
Maître ès sciences (M. Sc.)
Ré sumé
L’une des principales maladies attaquant le soya est la pourriture phytophthoréenne, causée par l’agent pathogène Phytophthora sojae. La méthode de lutte la plus efficace à ce jour pour contrer cet agent pathogène est la lutte génétique. Des gènes de résistance (Rps) se trouvant naturellement dans certaines lignées de soya sont introgressés dans des cultivars ayant un attrait pour l’agriculture. Cependant, pour définir quel gène Rps utiliser, il est essentiel de connaître les pathotypes de P. sojae se trouvant dans le sol, puisque les gènes Rps reconnaissent les gènes Avr caractérisant les différents pathotypes. Actuellement, les méthodes d’identification des nombreux pathotypes de l’agent pathogène sont des techniques de phénotypage longues et parfois imprécises. Cette étude présente donc le premier outil moléculaire ayant pour but de diagnostiquer rapidement et précisément les pathotypes de P. sojae se trouvant dans un échantillon de sol ou de tissus végétaux infecté. Une étude exhaustive de 31 isolats de P. sojae préalablement réalisée a permis d’identifier des marqueurs discriminants entre les haplotypes de virulence et d’avirulence pour les sept principaux gènes Avr retrouvés en Amérique. Des amorces spécifiques aux différents marqueurs ont été créées. Elles ont par la suite été adaptées afin de pouvoir être utilis.es simultanément dans une PCR multiplexe. Un taux d’efficacité à identifier les gènes d’avirulence présents chez différents isolats de P. sojae de 96% a été atteint lors de l’étude, Avr3a étant le seul gène à présenter des résultats aléatoires. Cela a donc ouvert la porte à d’éventuelles études plus approfondies sur l’interaction entre les gènes Rps3a et Avr3a. Le test sera de plus un outil précieux dans la prise de décision du cultivar à semer pour les producteurs, qui auront désormais accès à plus d’informations quant aux souches de P. sojae se trouvant dans leurs champs.
Tablé dés matié rés
Résumé ... iii
Liste des tableaux ... vii
Liste des figures ... viii
Remerciements ... ix
Avant-propos ... x
Introduction ... 1
Hypothèse et objectifs ... 3
Chapitre 1 — Revue bibliographique ... 4
Le soja ... 4
Phytophthora sojae ... 5
Cycle évolutif ou infectieux ... 5
Symptômes ... 7
Méthodes de lutte ... 9
Diversité des races de P. sojae et les gènes de résistance ... 10
Résistance verticale contre Phytophthora sojae ... 12
PCR multiplexe ... 14
Réaction Polymérase en Chaine (PCR) ... 14
PCR multiplexe ... 16 Chapitre 2 — Manuscrit ... 20 Running head ... 20 Keywords ... 20 Résumé ... 20 Summary ... 21 Introduction ... 21
Experimental Procedures ... 24 Results ... 27 Discussion ... 37 Acknowledgements ... 40 References ... 41 Conclusions ... 46 Bibliographie ... 50
Listé dés tabléaux
Chapitre 1 – Revue de littératureTableau 1 Les pathotypes des différentes races de Phytophthora sojae et leur importance de 1980 à 1989 et de 2010-2012, ainsi que la différence entre ces deux périodes. ... 11 Tableau 2 Exemples de pathotypes de certaines races de Phytophthora sojae identifiés avec un différentiel de huit cultivars de soya chacun possédant un unique gène Rps (d’après Sugimoto et al. (2012)) ... 13
Chapitre 2 — Manuscrit
Table 1. Primer sequences, with their genomic positions, amplicon size and concentrations, used for the detection of seven Avr genes in the Phytophthora sojae genome ... 28 Table 2. Comparative results of predicted pathotypes between the molecular assay and the hydroponic assay for 25 isolates of Phytophthora sojae ………..36
Listé dés figurés
Chapitre 1 — Revue de littérature
Figure 1 Répartition de la culture du soya dans les différentes provinces du Canada. ... 4 Figure 2 Illustration du cycle évolutif et infectieux de Phytophthora sojae. ... 6 Figure 3 Pourriture des graines de soya causée par Phytophthora sojae. ... 7 Figure 4 Taches noires sur les cotylédons et mort des plantules du soya causées par Phytophthora sojae. ... 8 Figure 5 Mort de plants de soya infectés par Phytophthora sojae dans un champ. ... 8 Figure 6 Illustration des trois étapes d’un cycle de réaction polymérase en chaine (PCR) : dénaturation, hybridation et élongation. ... 16 Figure 7 Illustration d’une PCR multiplexe comportant quatre paires d’amorces dans un même tube de réaction permettant d’amplifier quatre gènes différents. ... 17
Chapitre 2 — Manuscrit
Figure 1 Uniplex PCR amplifications of each of the Avr gene among different P. sojae isolates. ... 30 Figure 2 Multiplex PCR analyses for the 31 isolates with a known haplotype. ... 32 Figure 3 Genotyping results from multiplex PCR analyses and hydroponic phenotyping obtained for isolate 2012-82………..34
Rémérciéménts
Tout d’abord, j’aimerais remercier mon directeur de recherche, monsieur Richard R. Bélanger, de m’avoir accueillie dans son équipe de recherche et de m’avoir soutenue et encouragée tout au long de ma maîtrise. Merci aussi de m’avoir proposé ce projet stimulant et rempli de défis. J’adresse également ma gratitude à mon codirecteur, monsieur François Belzile.
Ensuite, mes remerciements vont à Geneviève, Huma, Caroline et Benjamin, pour m’avoir aidée à résoudre tous les problèmes que j’ai rencontrés lors de mon projet. Leur expertise en génomique, PCR, etc. a été essentielle au développement de mes compétences lors de ma maîtrise.
Merci aussi à tout le reste de l’équipe, Aliyeh, Daphné, Maxime, Amandine, Rupesh, Partha, Chiva, Krishna, Devrim, ainsi qu’aux étudiants d’été, pour avoir rendu ces deux années aussi enrichissantes et amusantes.
Un merci bien particulier à tous les partenaires du projet SoyaGen pour leur support financier tout au long du projet.
Avant-propos
Ce mémoire débute avec une revue bibliographique portant sur la culture du soya au Canada, la maladie causée par Phytophthora sojae, des informations sur l’agent pathogène et les dégâts engendrés par ce dernier, la résistance verticale appliquée à P. sojae, ainsi que sur les réactions PCR. Ceci clôt le premier chapitre. Pour ce qui est du deuxième chapitre, il est écrit sous forme de manuscrit pour publication dont je suis l’auteure principale. L’article a été soumis à la revue Molecular Plant Pathology dans la semaine du 29 avril 2019. Ce deuxième chapitre porte sur la méthode utilisée lors du développement d’une PCR multiplexe pour diagnostiquer la présence ou l’absence des sept principaux gènes d’avirulence présents chez P. sojae. Ce travail a été réalisé grâce à la collaboration de Geneviève Arsenault-Labrecque et d’Humira Sonah, qui ont apporté une aide indispensable dans l’analyse des différents pathotypes de P. sojae, de François Belzile et de Richard R. Bélanger, pour la mise en place du projet de maîtrise et l’écriture de l’article et du mémoire. Finalement, le troisième chapitre présente les conclusions de ce qui a été réalisé grâce à cette étude, ainsi que des ouvertures possibles suite au projet.
Introduction
Le soya, Glycine max (L.) Merr., est devenu au fil des années la troisième culture en importance au Canada. En effet, les superficies cultivées en soya sont passées de 1,2 million d’hectares en 2006 à plus de 3,5 millions en 2017 (Dorff, 2007, Statistique Canada, 2017). De plus, la superficie semée en soya au Québec dépassait pour la première fois les superficies semées en maïs-grain en 2017. Cette importante augmentation a aussi entraîné son lot de problèmes. L’intensification de l’agriculture et l’utilisation de rotations de cultures de plus en plus courtes pour tenter de semer des cultures plus rentables comme le soya a favorisé l’apparition de nouvelles maladies et l’augmentation des populations. Ces dernières sont donc de plus en plus présentes et difficiles à réprimer. Parmi celles-ci, la pourriture phytophthoréenne des racines du soya, causée par l’oomycète Phytophthora sojae Kaufm. & Gerd. 1958, est l’une des plus importantes en matière de pertes économiques. Effectivement, ce serait plus de 20 millions de tonnes métriques qui seraient détruites annuellement dans le monde par cette maladie (Dorrance, 2018). Cela représente jusqu’à 2 milliards de dollars de pertes par année à travers le monde (Tyler, 2007).
Le soya est important pour l’agriculture canadienne, en raison de ses usages multiples possibles. Il existe différents cultivars de soya, ayant chacune son propre usage. Une partie de ces cultivars est destinée à la consommation humaine pour le lait de soya, le tofu, la sauce soya, le miso, le tempeh, l’huile, etc. Une autre partie est pour l’alimentation animale sous forme de tourteau de soya (sous-produit de l’extraction de l’huile) et de fèves de soya grillées. Ensuite, il y a les cultivars destinés aux produits industriels tels que l’encre d’imprimerie, le biodiésel, la paraffine, les solvants, les lubrifiants, etc. (Dorff, 2007).
La pourriture phytophthoréenne a été identifiée pour la première fois en 1946 en Indiana aux États-Unis (Kaufmann et Gerdemann, 1958). Depuis ce temps, elle ne cesse de s’étendre sur le territoire américain et canadien et de causer des dégâts importants. Les chercheurs s’efforcent depuis à développer des moyens de limiter ses dégâts et son expansion. Malheureusement, P. sojae est un agent pathogène qui mute facilement et s’adapte rapidement à des environnements non favorables ou au déploiement de gènes de résistance
(Chen et Wang, 2017, Dorrance et al., 2003). Il existe donc maintenant plus de 200 souches, ou combinaison de pathotypes, différentes de P. sojae simplement sur le territoire canadien (Dorrance et al., 2016). Les phytopathologistes tentent donc d’étudier en profondeur cet agent pathogène afin d’en connaître davantage sur son cycle évolutif ou infectieux, les conditions favorables à son développement, les symptômes engendrés, les moyens d’attaque sur les plants de soya, etc. Cela permet de mettre en place des études sur les moyens de lutte potentiellement efficaces pour contrer P. sojae. Parmi les méthodes testées, l’enrobage de semences avec des fongicides comme le métalaxyl est assez efficace ; les rotations de cultures par contre le sont peu, puisque l’agent pathogène peut survivre plusieurs années sous forme de spores de conservation nommées oospores (Schmitthenner, 2000).
Pour trouver une alternative plus écologique aux fongicides, les chercheurs se sont penchés sur la présence d’une résistance chez certains cultivars de soya. Ils ont découvert des gènes de résistance à P. sojae (Rps) qui permettaient au soya de contrecarrer les attaques de l’oomycète qui contient les gènes d’avirulence (Avr) correspondants (Athow et al., 1974, Bernard et al., 1957). Depuis ce temps, plusieurs gènes Rps ont été introgressés à différentes lignées de soya pour lutter contre P. sojae (Abeysekara et al., 2016, Dorrance et Schmitthenner, 2000). Cependant, comme mentionné précédemment, P. sojae possède une grande flexibilité génétique, comme plusieurs autres oomycètes, et a rapidement contourné les gènes de résistance du soya (Tyler, 2007). Cela demande donc un travail récurrent pour la découverte et le développement de nouveaux gènes Rps, puisqu’il s’agit de la méthode la plus efficace à ce jour pour diminuer les effets de cette maladie sur les champs de soya.
Le principal atout d’un outil tel que celui sera de pouvoir conseiller les producteurs de soya sur le cultivar à utiliser en fonction du profil de virulence des isolats de P. sojae présents dans leurs champs. De plus, pour aider les phytogénéticiens à développer de nouvelles
sélectionneurs doivent connaître la nature précise des gènes d’avirulence (Avr). À cette fin, un test PCR multiplexe serait un outil potentiellement très utile pour un diagnostic rapide et précis des gènes Avr présents dans un isolat de P. sojae.
Hypothé sé ét objéctifs
Grâce à l’analyse génomique de différentes souches de Phytophthora sojae réalisée par Arsenault-Labrecque et al. (2018), il a été possible de trouver des zones discriminantes, appelées marqueurs, au sein des différents allèles. Cette découverte a permis de soumettre l’hypothèse qu’il était possible de développer des amorces se basant sur ces marqueurs pour amplifier les principaux gènes d’avirulence (Avr) de P. sojae et servir ainsi dans un test diagnostic pour la mise en évidence des allèles avirulentes de ces gènes.
Cette hypothèse pourra être validée grâce aux quatre objectifs suivants : 1) identifier des zones discriminantes du génome de P. sojae permettant de créer des amorces discriminantes pour les sept principaux gènes d’avirulence (1a, 1b, 1c, 1d, 1k, 3a, 6) ; 2) développer des amorces spécifiques à ces marqueurs pour chacun des sept gènes d’avirulence ; 3) combiner ces amorces dans le contexte d’un test PCR multiplexe ; et 4) valider la fiabilité du test en comparant des résultats de phénotypage et de PCR multiplexe pour la caractérisation d’isolats inconnus.
Chapitré 1 — Révué bibliographiqué
Le soja
Le soya, de son nom latin Glycine max, est une plante annuelle de la famille des Fabaceae (Fabacées). Il a été introduit au Canada vers le milieu des années 1800, mais proviendrait originairement de l’Asie du Sud-Est, où il serait cultivé depuis des milliers d’années déjà. Jusqu’en 1970, la culture du soya se faisait principalement en Ontario, là où le climat le permettait. Par la suite, de nouvelles lignées de soya adaptées à des climats plus froids furent créées (Dorff, 2007) et la culture de cette légumineuse s’est étendue au Manitoba, en Saskatchewan et au Québec (Figure 1). Les superficies dépassaient déjà les 3,5 millions d’hectares en 2017 (Statistique Canada, 2017). Cette popularité qui a mené le soya au troisième rang en importance des superficies semées au Canada est principalement due à sa haute teneur en protéines (35 %) et en huile (18 %), ainsi qu’au fait qu’il fixe l’azote atmosphérique grâce à une bactéries vivant en symbiose sur les racines (Bradyrhizobium japonicum) (Albrecht et al., 1979), réduisant ses besoins en amendements azotés (Dorff, 2007). Cette rapide expansion de la culture a par contre entraîné une augmentation des populations d’insectes ravageurs et des maladies, telles que la pourriture phytophthoréenne des racines du soya (PPR).
Phytophthora sojae
La pourriture phytophthoréenne des racines du soya, causée par l’oomycète Phytophthora sojae M. J. Kaufmann & J. W. Gerdemann, est l’une des maladies les plus limitantes pour la culture du soya actuellement au Canada (Wrather et al., 2010). Le soya est son hôte principal et celui ayant la plus grande importance économique. L’agent pathogène est toutefois capable d’infecter les plantes du genre Lupinus. Il aurait été observé pour la première fois en Indiana en 1948, mais n’aurait été identifié qu’en 1954 en Ohio et en Caroline du Nord. Par la suite, la maladie aurait été aussi identifiée en Argentine, en Australie, au Brésil, au Canada, en Hongrie, en Chine, au Japon, en Italie, etc. (Schmitthenner, 2000).
Les oomycètes sont des organismes eucaryotes filamenteux aquatiques s’apparentant à des algues, mais qui ne réalisent pas de photosynthèse. Morphologiquement, ils ressemblent fortement aux champignons. C’est entre autres pour cela qu’ils sont appelés pseudo-champignons, malgré qu’ils appartiennent au règne des Straménopiles, qui représente une branche distincte de celle des champignons (Eumycota) dans l’arbre phylogénétique. Les oomycètes sont dans le même règne que les algues brunes (Tyler, 2007). Leurs hyphes, contrairement aux champignons, sont non cloisonnés et constitués de cellulose plutôt que de chitine (Bartnicki-Garcia, 1968).
Cycle évolutif ou infectieux
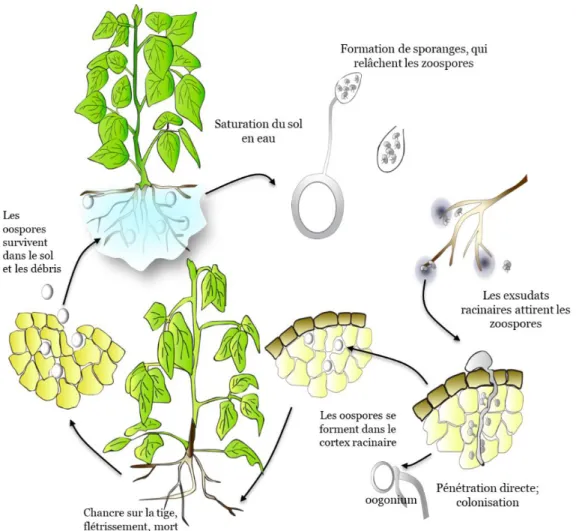
Les conditions favorables au développement de cette maladie sont un sol argileux et mal drainé qui peut facilement être inondé pendant une bonne période de temps. En effet, c’est à ce moment que les oospores, la forme de conservation de P. sojae, vont germer en sporanges contenant des zoospores. Ces zoospores sont des spores motiles pouvant se déplacer facilement dans l’eau grâce à deux flagelles. Ces spores motiles sont attirées par les exsudats des racines du soya par chimiotactisme. Une fois les racines atteintes, les zoospores s’enkystent et adhèrent à ces dernières en formant un tube germinatif, puis des hyphes intercellulaires et un haustorium (Lebreton et al., 2018, Tyler, 2007). Suite à la reproduction sexuée dans le cortex racinaire, de nouvelles oospores sont formées et peuvent survivre dans la plante ou les tissus morts pendant plusieurs années, même lors de conditions défavorables (Tyler, 2007). Le cycle recommence par la suite, lorsque les conditions au champ sont de
cycle évolutif ou infectieux de P. sojae est présenté à la Figure 2. De plus, P. sojae est un oomycète homothallique, c’est-à-dire que les hyphes des deux sexes proviennent du même thalle et qu’il est donc autofertile. Phytophthora sojae est hémibiotrophe, donc il commence son cycle infectieux en étant biotrophe, puis lorsque le plant ou les tissus meurent, il y a transition vers la phase nécrotrophe (Dorrance, 2018).
Figure 2 Illustration du cycle évolutif et infectieux de Phytophthora sojae. Tirée de Lebreton (2015)
Symptômes
Phytophthora sojae est capable d’infecter le soya à tous ses stades de développement. En autant que les conditions favorables mentionnées précédemment soient rencontrées, la maladie peut atteindre les graines et causer la pourriture des semences (Figure 3), ainsi que la fonte des semis des plantules (Figure 4). Chez les lignées sensibles, P. sojae peut entraîner un brunissement, appelé chancre, et la mort des racines et de la tige du plant (Figure 5). Il arrive parfois que le feuillage soit aussi atteint et brunisse et flétrisse. Les symptômes de la maladie apparaissent souvent une à deux semaines après une forte pluie (Dorrance et Berry, 2007) et sont très difficiles à différencier des symptômes causés par d’autres agents pathogènes appartenant aux oomycètes comme Rhizoctonia solani.
Figure 3 Pourriture des graines de soya causée par Phytophthora sojae. © Laboratoire d’expertise et de diagnostic en phytoprotection — MAPAQ https://www.iriisphytoprotection.qc.ca/Fiche/Champignon?imageId=3065
Figure 4 Taches noires sur les cotylédons et mort des plantules du soya causées par Phytophthora sojae.
© Laboratoire d’expertise et de diagnostic en phytoprotection — MAPAQ
Figure 5 Mort de plants de soya infectés par Phytophthora sojae dans un champ. © Pierre Lachance — MAPAQ
Méthodes de lutte
Parmi les principales méthodes de lutte utilisées actuellement, les traitements de fongicides sur les semences, particulièrement de métalaxyl, sont utilisés pour prévenir les dégâts causés à la graine. En revanche, cette substance active fongicide est communément utilisée pour lutter contre les oomycète et quelques espèces ont déjà été rapportées pour avoir développé une résistance (Dorrance et al., 2007). Deux autres fongicides ont aussi démontré une efficacité contre P. sojae. Il s’agit de l’éthaboxam en traitement de semences (Dorrance et al., 2012, Radmer et al., 2017, SAgE pesticides, 2019) et l’oxathiapiproline(Miao et al., 2016). D’autres fongicides sont utilisés en combinaison en traitement de semences selon SAgE pesticides (2019), tels que fludioxonil / métalaxyl-M ; fludioxonil / métalaxyl-M / sédaxane et métalaxyl + pyraclostrobine.
Comme autres méthodes, l’utilisation de certaines pratiques culturales peut aider à diminuer l’effet de la pourriture phytophthoréenne sur les plants de soya. Entre autres, puisque la maladie se développe dans un sol compacté et mal drainé, le drainage et les travaux du sol, permettant un décompactage, peuvent aider à diminuer les sites d’infestation de la maladie (Dorrance et al., 2007, Workneh et al., 1998). Les rotations de cultures avec des cultures non-hôtes peuvent être utilisées pour limiter l’augmentation de l’inoculum dans le sol, mais sur de longues périodes de temps, puisque P. sojae peut survivre dans le sol sous forme d’oospore plusieurs années (Schmitthenner, 2000).
La méthode de lutte ayant démontré la plus grande efficacité pour réprimer P. sojae est l’utilisation de gènes de résistance Rps (Résistance à Phytophthora sojae), ainsi que de la résistance partielle (Dorrance et al., 2009). Les gènes Rps procurent au soya une résistance verticale, complète, à l’agent pathogène possédant le gène d’avirulence (Avr) correspondant. Cette notion sera discutée dans la prochaine section de la revue bibliographique. La résistance partielle (Abeysekara et al., 2016) ou quantitative (Dorrance, 2018), quant à elle, est non différentielle et conférée par plusieurs gènes. Sans être complète, la résistance partielle et quantitative est surtout efficace contre toutes les races (“provides resistance to all physiological races”) de P. sojae (Abeysekara et al., 2016). Elle permet de réduire la colonisation des racines par l’agent pathogène et diminue l’apparition de lésions pouvant
causer la mort du plant (Dorrance et al., 2007). Une combinaison de gènes Rps de résistance complète et de gènes de résistance partielle ou quantitative est ce qui permet le mieux de lutter contre P. sojae pour le moment (Dorrance, 2018 ; Dorrance et al., 2009).
Diversité des races de P. sojae et les gènes de résistance
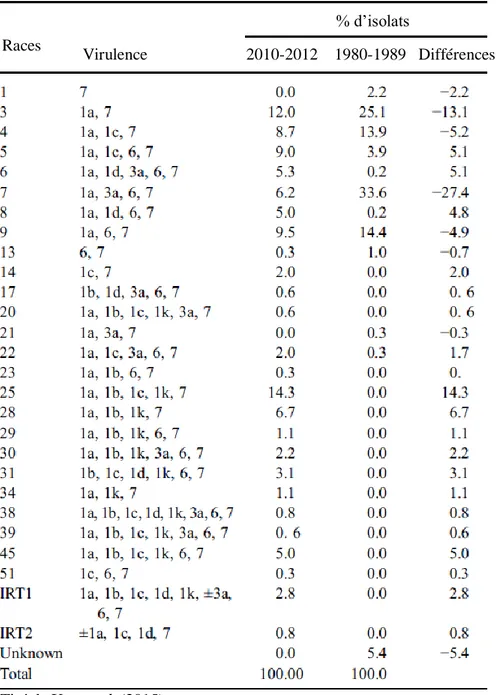
Dans les années 1960, seules des races de P. sojae ayant des pathotypes simples ont été identifiées au départ (Grau et al., 2004). À partir des années 2000, des races plus complexes ont été identifiées en Amérique du Nord pour contourner les gènes de résistance utilisés dans le soya (Dorrance et al., 2003, Jackson et al., 2004, Kaitany et al., 2001, Malvick et Grunden, 2004, Nelson et al., 2008). Les gènes Rps peuvent ne plus être effectif lorsque, par exemple, l’agent pathogène perd la capacité de produire la protéine codante pour un gène d’avirulence fonctionnel suite à une mutation de nucléotide ou d’une délétion. Ces variations dans le gène peuvent entrainer une baisse de l’expression du gène d’avirulence ou le rendre non reconnaissable par la plante (Tyler and Gijzen, 2014). Une race se caractérise par des isolats ayant le même pathotype et donc le même profil de virulence. Xue et al. (2015) (Tableau 1) montrent un ensemble des pathotypes des différentes races retrouvées au Canada, ainsi que leur proportion (%). Selon le plus récent recensement, on retrouverait plus de 200 pathotypes
Tableau 1 Les pathotypes des différentes races de Phytophthora sojae et leur proportion (%) de 1980 à 1989 et de 2010-2012, ainsi que la différence entre ces deux périodes.
Tiré de Xue et al. (2015)
Grâce à ce tableau, tiré de Xue et al. (2015), on voit l’évolution des races de P. sojae au fil des années et leur proportion courante. Cependant, cette étude a été réalisée sur le territoire de l’Ontario seulement, donc les proportions des races présentes dans les sols ailleurs au Canada peuvent varier, même si les pathotypes retrouvés sont similaires dans les autres provinces.
% d’isolats Races
Jusqu’à présent, on connaît environ 28 gènes Rps dans le soya (Lebreton et al., 2018), se trouvant sur huit loci différents (Abeysekara et al., 2016, Sandhu et al., 2004, Zhang et al., 2010). Parmi ces 28 gènes, huit ont été introgressés dans les cultivars de soya : Rps1a, Rps1b, Rps1c, Rps1k, Rps2, Rps3a, Rps6 et Rps7 (Abeysekara et al., 2016, Dorrance et Schmitthenner, 2000). À chacun de ces gènes de résistance doit correspondre un gène Avr chez P. sojae et chacune des races peut porter une combinaison unique de ces gènes d’avirulence.
Actuellement, très peu d’informations sont disponibles pour les producteurs de soya quant aux souches de P. sojae se trouvant dans leurs sols. Cela s’ensuit donc par une mauvaise utilisation des gènes Rps disponibles dans les cultivars de soya. Lorsqu’un cultivar est non adapté à la souche présente dans le sol, cela créer une pression de sélection des souches virulentes sur le territoire. Ces souches contourneront rapidement le gène Rps introduit et il sera nécessaire d’en introgresser de nouveaux pour contrer la maladie. Si les producteurs de soya avaient accès plus d’informations à ce sujet, ils pourraient faire un choix éclairé de cultivars et réduire la pression de sélection exercée sur P. sojae. Une bonne gestion de la résistance au champ permettra d’augmenter la durée d’efficacité des gènes Rps.
Résistance verticale contre Phytophthora sojae
Comme mentionné précédemment, la résistance verticale est procurée par la présence d’un seul gène Rps et consiste en une résistance complète du soya à certaines races de P. sojae. Il y a donc absence complète du développement de la maladie par les races concernées. Ces gènes Rps ont pour fonction d’activer des effecteurs ayant un rôle dans la résistance du soya contre les agents pathogènes (Dong et al., 2011). En revanche, pour que la plante active sa résistance complète, elle doit être en contact avec un agent pathogène possédant un gène Avr correspondant aux gènes Rps qu’elle possède. Par exemple, un plant de soya possédant le gène Rps1a activera sa résistance complète contre une souche de P. sojae possédant le gène
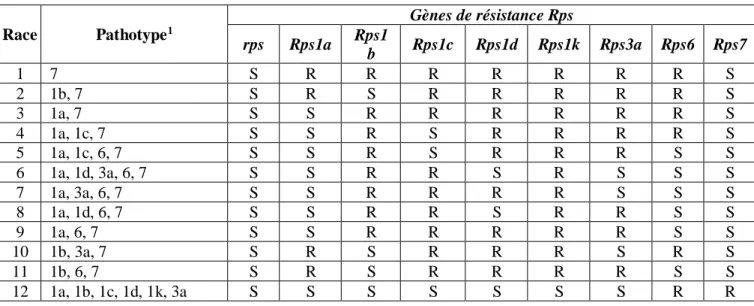
Tableau 2 Exemples de pathotypes de certaines races de Phytophthora sojae identifiés avec huit lignées différentielles de soya chacune possédant un unique gène Rps (d’après Sugimoto et al. (2012)) Race Pathotype1 Gènes de résistance Rps rps Rps1a Rps1 b Rps1c Rps1d Rps1k Rps3a Rps6 Rps7 1 7 S R R R R R R R S 2 1b, 7 S R S R R R R R S 3 1a, 7 S S R R R R R R S 4 1a, 1c, 7 S S R S R R R R S 5 1a, 1c, 6, 7 S S R S R R R S S 6 1a, 1d, 3a, 6, 7 S S R R S R S S S 7 1a, 3a, 6, 7 S S R R R R S S S 8 1a, 1d, 6, 7 S S R R S R R S S 9 1a, 6, 7 S S R R R R R S S 10 1b, 3a, 7 S R S R R R S R S 11 1b, 6, 7 S R S R R R R S S 12 1a, 1b, 1c, 1d, 1k, 3a S S S S S S S R R
1 Un pathotype représente un ensemble d’isolats (ou races), de Phytophthora sojae dans ce cas-ci,
présentant les mêmes profils de virulence contre le soya. Les pathotypes décrivent donc la capacité de ces isolats (ou races) à rendre susceptible sensible un cultivar de soya possédant le gène de résistance (Rps) correspondant.
Présentement, pour diagnostiquer le profil de virulence de P. sojae, deux bio-essais sont utilisés. Tout d’abord, le test encore reconnu officiellement est le test de l’hypocotyle, consistant à injecter une bouillie mycélienne d’un isolat de P. sojae dans l’hypocotyle après l’avoir incisé au scalpel. On regarde par la suite si le plant meurt ou survit (Dorrance et Berry, 2007). Suite à l’obtention de résultats contradictoires, Lebreton et al. (2018) ont mis en place un test phénotypique par bassin hydroponique, consistant à faire pousser les plantules flottantes dans une suspension de jus de sol et d’eau, laquelle est inoculée d’une ou plusieurs souches de P. sojae. Ce test étant plus fidèle aux conditions favorables au développement de l’agent pathogène au champ ; il est aussi plus fiable que le test de l’hypocotyle, puisqu’il permet d’obtenir moins de fausses réponses sensibles (faux positifs) qu’avec l’ancienne méthode. En revanche, ce test peut tout de même prendre jusqu’à 3 mois à réaliser, entre la croissance de l’agent pathogène, la préparation de l’inoculum, la croissance des plantes, la mise en bassin et l’obtention des résultats. De plus, il nécessite un grand espace en serre pour disposer les bassins hydroponiques. En ce moment, il est principalement utilisé en recherche, mais n’est pas utilisé pour conseiller les producteurs sur le cultivar à choisir en fonction des souches trouvés dans leurs champs.
Arsenault-Labrecque et al. (2018) ont récemment analysé le génome de 31 isolats de P. sojae et découvert les variations génétiques des sept principaux gènes Avr. À partir des signatures génomiques identifiées, il serait par la suite possible d’identifier des marqueurs permettant de discriminer les allèles virulents (causant la maladie) des allèles avirulents (ne causant pas la maladie) pour les sept gènes d’avirulence étudiés. Ces marqueurs permettraient de créer des amorces discriminantes entre les allèles et ainsi pouvoir caractériser le profil de virulence des isolats de P. sojae pour ces sept gènes Avr.
PCR multiplexe
nombre important de copies d’une région précise d’intérêt (Mullis, 1990), à partir d’une quantité minime d’ADN.
Pour réaliser une réaction PCR, quelques produits sont indispensables, tels qu’un échantillon d’ADN d’intérêt, des nucléotides (Adénine, Thymine, Cytosine, Guanine), une enzyme polymérase et des amorces spécifiques à la région que l’on veut amplifier. L’échantillon d’ADN est celui qui contient les séquences qui seront amplifiées. Les nucléotides permettent de construire les copies amplifiées d’ADN grâce à l’enzyme polymérase. Cete dernière enzyme est celle qui permet la synthèse des brins complémentaires d’ADN aux brins de l’échantillon d’ADN. Les amorces, quant à elles, sont de courts fragments d’ADN (environ 20 paires de bases en général) complémentaires à l’ADN que l’on veut amplifier et qui permettent à l’enzyme d’amplifier exactement le fragment d’ADN voulu. Les amorces se lient au brin d’ADN et l’enzyme polymérase s’y lie à son tour pour synthétiser le brin d’ADN complémentaire. Les amorces permettent ainsi d’obtenir une région spécifique amplifiée (Garibyan et Avashia, 2013).
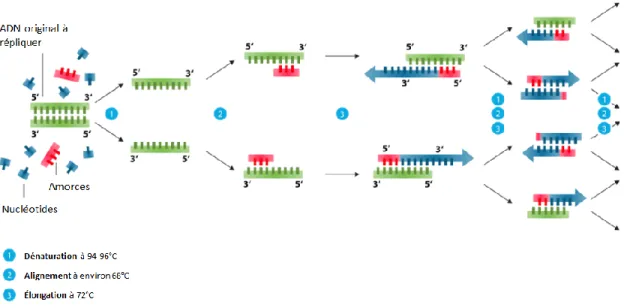
Cette réaction PCR est réalisée grâce à un appareil nommé thermo-cycleur, qui permet de répéter un même cycle à plusieurs reprises. Il permet aussi d’élever la température du bloc, ainsi que de la diminuer à des températures très précises et pour des temps bien précis. Tous les produits mentionnés précédemment sont mis dans un même tube et disposés dans le thermo-cycleur. Les principales étapes d’un cycle d’une réaction PCR sont les suivantes. La première étape est l’étape de dénaturation et consiste à élever la température au point de fusion des deux brins d’ADN complémentaires. Cette température est aux alentours de 90-95°C pendant 30 secondes. La seconde étape est nommée l’alignement (ou l’hybridation), puisqu’il s’agit du moment où les amorces spécifiques s’alignent aux brins d’ADN complémentaires dénaturés. Cette partie de la réaction est permise grâce à une baisse de température à environ 50-60°C, dépendamment de la température d’alignement des amorces choisies. Lors de la troisième étape, la température s’élève de nouveau pour permettre à l’enzyme polymérase de construire les nouveaux fragments complémentaires d’ADN à partir des amorces en ajoutant des nucléotides. Cette étape est nommée l’élongation et se produit à des températures avoisinant 68°C. Ces trois étapes forment un cycle, et la réaction PCR peut
nécessiter un nombre élevé de ce même cycle. À chacun de ces cycles, le nombre de copies du fragment d’ADN reconnu par les amorces double (Garibyan et Avashia, 2013). La Figure 6 montre un cycle d’une réaction PCR.
Figure 6 Illustration des trois étapes d’un cycle de réaction polymérase en chaine (PCR) : dénaturation, hybridation et élongation.
Adaptée de https://en.wikipedia.org/wiki/Polymerase_chain_reaction (2019)
PCR multiplexe
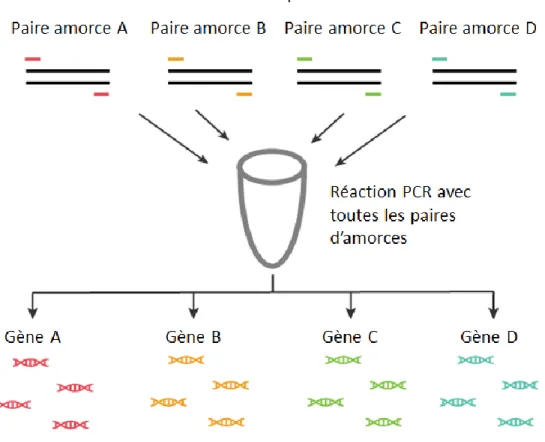
Une PCR multiplexe est une réaction PCR comportant plusieurs régions à amplifier dans une même réaction grâce à plusieurs paires d’amorces. Les PCR multiplexes permettent entre autres de détecter plusieurs régions simultanément dans une seule et même réaction. On gagne ainsi en temps et en coûts de réactifs et de main-d’œuvre lorsqu’on utilise ce type de PCR. La Figure 7 montre les étapes et composantes d’une PCR multiplexe.
Figure 7 Illustration d’une PCR multiplexe comportant quatre paires d’amorces dans un même tube de réaction permettant d’amplifier quatre gènes différents.
© ABM
https://www.abmgood.com/marketing/knowledge_base/polymerase_chain_variation_system.php
Dans ce type de réaction PCR plus complexe, les amorces se doivent d’être encore plus spécifiques et les conditions entourant la réaction se doivent d’être précises, pour éviter d’amplifier autre chose que les régions désirées (Henegariu et al., 1997). Par amorces spécifiques, on entend que les amorces ne doivent pas se lier ailleurs dans l’ADN. De plus, si les amorces sont spécifiques, la compétition présente dans une PCR multiplexe entre les amorces pour les réactifs est atténuée. Lors de la création des amorces, il est aussi important de s’assurer que les amorces ne sont pas complémentaires entre elles pour éviter la formation de structures nommées dimères. Cela a pour effet négatif de diminuer la quantité d’amorces disponibles pour l’amplification voulue, donc d’obtenir une quantité moindre d’amplicons à la fin de la réaction. Parmi les autres conditions qui doivent être respectées, il y a la température de fusion des amorces. Les amorces doivent avoir des températures de fusion
(Tm) semblables de 3-5°C pour que l’alignement des amorces avec l’ADN soit optimal à la température à laquelle le thermo-cycleur sera programmé. Cette température de fusion se situe habituellement aux environs de 55-60°C, mais plus la séquence de l’amorce contient de nucléotides G ou C, plus la Tm sera élevée (Biosoft, 2018).
Plusieurs paramètres se doivent d’être ajustés lors de la mise en place d’un test PCR multiplexe. Par exemple, puisque les amorces peuvent se faire compétition pour les autres réactifs de la solution, leur concentration doit être optimale pour qu’il n’y ait pas d’amorces plus amplifiées que d’autres. Pour contrôler ce paramètre, on met d’abord les amorces dans une même réaction et on ajuste ensuite les concentrations à la baisse pour les amorces menant à une amplification trop forte et à la hausse pour celles ayant une amplification trop faible. Les concentrations optimales sont obtenues lorsque chaque amorce est amplifiée avec une intensité égale (Sint et al., 2012). De plus, dépendamment de la quantité d’ADN mise dans la réaction, le nombre de cycle se doit d’être ajusté afin d’obtenir un bon rendement de produit PCR final. En effet, augmenter un peu le nombre de cycle peut permettre d’obtenir plus de produits suite à la réaction. Par contre, plus de 45 cycles peuvent causer l’apparition de bandes non spécifiques et diminuer l’efficacité de la PCR dû à l’accumulation de sous-produits de la réaction et la diminution des réactifs (ThermoFischer Scientific, 2019).
La PCR multiplexe comporte plusieurs avantages. Comme mentionné précédemment, le multiplexage permet de sauver du temps et de l’argent, puisqu’on utilise approximativement la même quantité de réactifs pour faire une seule réaction de PCR multiplexe que lorsqu’on fait une seule réaction PCR en uniplex, c’est-à-dire que les paires d’amorces sont séparées dans différents tubes de réaction. On détecte donc plusieurs produits avec la même quantité de réactifs que lorsqu’on en détecte un seul. De plus, il est souvent plus rapide de tester toutes les amorces dans une même réaction, plutôt que de les tester individuellement. Par la suite,
PCR pour l’identification des champignons
À l’origine, l’identification de champignons était réalisée grâce à la culture de ces derniers sur des milieux de culture sélectifs à partir de parties de plants infectés. Le mycélium et les structures de fructifications étaient observés au microscope pour être décrites. La principale difficulté rencontrée est la complexité de l’isolement en culture pure de l’agent pathogène exposé à la contamination. Pour contrer ces difficultés, la réaction PCR a été fort utile. En effet, cette méthode permet d’amplifier une région de l’ADN du champignon inconnu et d’ensuite faire séquencer cet amplicon. Les résultats du séquençage sont par la suite comparés aux bases de données pour identifier le champignon (Cullings, 1992).
Les premières identifications de champignons par PCR ont été réalisées dans les années 1990. Parmis les premiers champignons pathogènes des plantes, Phoma tracheiphila fut identifié par PCR par Rollo et al. (1990). D’autres champignons pathogènes ont été subséquemment identifiés grâce à la PCR (Färber et al., 1997, Geisen, 1998, Henson et French, 1993, Shapira et al., 1996).
Par la suite, de nouvelles amorces pour l’identification de champignons producteurs de trichothécènes tels que Fusarium, Myrothecium, Stachybotrys, Trichoderma, Trichothecium, Verticimonosporium, etc. ont été développées (Niessen, 2007). Il s’agit, pour la plupart, d’agents pathogènes pouvant infecter les plantes.
Chapitré 2 — Manuscrit
Discriminant haplotypes of avirulence genes of Phytophthora
sojae lead to a molecular assay to
predict phenotypes
Chloé Dussault-Benoit, Geneviève Arsenault-Labrecque, Humira Sonah#, François
Belzile, and Richard R. Bélanger†
Department of Phytology, Université Laval, Québec, QC, Canada 2019
Running head
Molecular assay to predict Avr alleles of P. sojae
Keywords
Soybean diseases, Phytophthora root rot (PRR), Plant pathogen, Avr genes, Effectors, Rps genes, Multiplex Polymerase Chain Reaction
Résumé
La méthode de lutte la plus efficace pour lutter contre Phytophthora sojae, agent pathogène
causant la pourriture phytophthoréenne du soya (Glycine max), est la lutte génétique. En se
basant sur cette relation de type gène pour gène, il a été possible de développer un test moléculaire permettant d’identifier l’allèle (virulent vs avirulent) des sept principaux gènes Avr retrouvés chez P. sojae en Amérique (Avr1a, Avr1b, Avr1c, Avr1d, Avr1k, Avr3a et Avr6). En se basant sur les analyses génomiques de 31 isolats ayant un pathotype connu, des amorces spécifiques à chaque gène Avr ont pu être créées à partir de marqueurs discriminant les différents haplotypes afin d’amplifier l’allèle avirulent de chacun des gènes. Le test PCR multiplex a ensuite été réalisé sur 15 isolats au pathotype inconnu, et les résultats ont ensuite été comparés à ceux obtenus lors du bioessais en bassins
permet ainsi d’identifier les gènes Avr fonctionnels chez les isolats testés, et servira d’outil de décision aux producteurs de soya dans la sélection de cultivars résistants aux pathotypes retrouvés dans leurs champs.
Summary
The soybean-Phytophthora sojae interaction operates on a gene-for-gene relationship, where the product of a resistance gene (Rps) in the host recognizes that of an avirulence gene (Avr) in the pathogen to generate an incompatible reaction. To exploit this form of resistance, one must match with precision the appropriate Rps gene with the corresponding Avr gene. Currently, this association is evaluated by phenotyping assays that are labor-intensive and often imprecise. To circumvent this limitation, we sought to develop a molecular assay that would reveal the avirulent allele of the seven main Avr genes (Avr1a, Avr1b, Avr1c, Avr1d, Avr1k, Avr3a, and Avr6) in order to diagnose with precision the pathotypes of P. sojae isolates. For this purpose, we analyzed the genomic regions of these Avr genes in 31 recently sequenced isolates with different virulence profiles and identified discriminant mutations between avirulent and virulent alleles. Specific primers were designed to generate amplicons of a distinct size, and PCR conditions were optimized in a final assay of two parallel runs. When tested on the 31 isolates of known virulence, the assay accurately revealed all avirulence alleles. The test was further assessed and compared to a phenotyping assay on 25 isolates of unknown virulence. The two assays matched in 97% (170/175) of the interactions studied. Interestingly, the sole cases of discrepancy were obtained with Avr3a, which suggests a possible imperfect interaction with Rps3a. This molecular assay offers a powerful and reliable tool to exploit and study with greater precision soybean resistance against P. sojae.
Introduction
Soybean (Glycine max (L.) Merr.) production has steadily increased all over the world and particularly in Canada over the last decade, and it is now the third most important crop in the country, covering more than 3 million hectares (Statistics Canada, 2018). This rapid expansion has led to increased disease pressure from a variety of pathogens. Among them, Phytophthora sojae Kaufm. & Gerd., the causal agent of Phytophthora root rot (PRR), is arguably the most economically important soybean pathogen in Canada (Xue et al., 2015).
filamentous eukaryotic organism related to brown algae, but it does not perform photosynthesis. Like other oomycetes, morphologically, it strongly resembles fungi (Tyler, 2007), but its hyphae, unlike those of fungi, are non-partitioned and consist of cellulose rather than chitin (Bartnicki-Garcia, 1968).
The pathogen can infect soybean at all growth stages, from seed to adult plants (Tyler, 2007), and can cause damage at any time in the season. Symptoms include seed rot, damping-off, browning and death of the roots and stem. Sometimes the foliage can turn brown and wither. Conditions favorable to the development of this disease are poor drainage, frequent in clay soils, which can easily be flooded for a longer period. Under favorable conditions, the oospores, the survival structure of P. sojae, will germinate into sporangia containing zoospores that can move easily in water with their two flagella. These motile spores are attracted to the root exudates of soybean by chemotaxis. Once they reach the roots, the zoospores are able to penetrate and invade the root cells with a structure called a haustorium. Following sexual reproduction in the root cortex, new oospores are formed and can survive in the dead plant or tissues for many years even under adverse conditions (Tyler, 2007).
Currently, the most effective method to curb the devasting impact of P. sojae is genetic resistance in the host. Complete (or vertical) resistance against P. sojae is conferred by single genes named Rps (Resistance to Phytophthora sojae). These resistance genes act by recognizing the products of avirulence genes (Avr gene) of the pathogen, in a traditional gene-for-gene relationship. The Avr genes of P. sojae encode effector proteins, most with
RxLR (Arg-X-Leu-Arg) motifs, and these effectors are recognized by proteins having
nucleotide-binding site and leucine-rich repeat (NLR receptors) motifs, encoded by the Rps genes (Tyler and Gijzen, 2014).
Rps gene can be overcome and cease to be effective when the pathogen loses the ability to produce the corresponding Avr protein, following deletion or altered expression of an Avr gene, or alters its structure through point mutations or indels in the gene, making it unrecognizable by the product of the Rps gene (Tyler and Gijzen, 2014). Currently, there are approximately 25 known Rps genes in soybean, of which six have been introgressed internationally into commercial soybean varieties (Rps1a, Rps1b, Rps1c, Rps1k, Rps3a, Rps6), concordant with Avr genes in P. sojae (Abeysekara et al., 2016, Sugimoto et al., 2012).
To properly exploit Rps genes against P. sojae, deployment of soybean cultivars with an appropriate combination of Rps genes, to match Avr genes present in field isolates is essential. It is thus imperative to identify the virulence profile or pathotypes of these isolates present in a given environment by precisely detecting the presence or absence of functional Avr genes. For this purpose, phenotyping assays with soybean differentials carrying a single Rps gene have typically been exploited. The hypocotyl assay has been historically the most commonly used because of its simple protocol (Dorrance et al., 2008, Kaufmann and Gerdemann, 1958). However, problems with false positives, intermediate responses and stability of Avr genes/expression over time have hampered its reliability. More recently, Lebreton et al. (2018) proposed a new assay to phenotype the isolates of P. sojae that relied on zoospore inoculation in hydroponic solutions to more closely reproduce the natural course of infection. While this bioassay appears to eliminate some of the shortcomings of the hypocotyl assay, it remains labor-intensive and requires several weeks to complete.
In a recent effort to associate phenotypes with genotypes, Arsenault-Labrecque et al. (2018) sequenced 31 P. sojae isolates collected from Canadian fields, and analyzed the sequences and variants of the seven most common Avr genes: 1a, 1b, 1c, 1d, 1k, 3a, and 6. They were able to define specific haplotypes for each Avr gene, and further show, through the use of the hydroponic assay, that each haplotype corresponded to a distinct virulence profile. This suggests that genomic signatures can be used as accurate predictors of phenotypes and could potentially be exploited as diagnostic tools to identify pathotypes in P. sojae isolates. On the basis of this hypothesis, the objectives of the present study were:
et al. (2018), that are specific and discriminant to detect the presence of the allele(s) conferring avirulence for each of the seven Avr genes; 2) to design primers from these sequences and to develop a molecular approach combining all the specific pairs of primers needed to amplify the seven avirulence alleles and 3) to test the efficiency of the molecular tool by comparing the results with those obtained with a phenotyping assay. Here we describe a novel and unique diagnostic tool that define with unprecedented precision the virulence profiles of P. sojae isolates. This tool can find direct applications in the study of functionality of Avr and Rps genes and in the development and selection of germplasm resistant to P. sojae.
Experimental Procedures
Phytophthora sojae isolates and DNA extraction
All P. sojae isolates used in this study, including 31 previously sequenced by Arsenault-Labrecque et al. (2018), were sampled across Ontario (Canada) and their virulence profile was determined by the hypocotyl assay as reported by Xue et al. (2015).
The isolates were first subcultured on V8 agar medium (20% clarified V8) covered with wax paper to facilitate harvest of hyphae and spores. After one week, cultures were scraped off the paper with a scalpel and placed in 1.5-ml tubes with screw caps (OMNI International inc., Kennesaw, Gerorgia, United States). The tubes were then kept in the freezer at -80°C for 2-3 hours, and lyophilized overnight. The lyophilized samples were crushed with an Omni Bead Ruptor 24 (OMNI International). Then, the DNA was extracted from the crushed samples using the E.Z.N.A Plant DNA kit (Norcross, Georgia, United-States) following the manufacturer’s protocol for dried samples with slight modifications.
Sequence variations and allele-specific primer design
For designing allele-specific primers, the discriminant variations in the sequences of the different Avr genes of 31 isolates were studied and identified based on the Phytophthora
for marker development were selected to design the primer pairs for the seven Avr genes under study (Avr1a, 1b, 1c, 1d, 1k, 3a and 6). In cases where deletions were present, at least one primer was positioned in the deletion such that the avirulent allele (i.e. without the deletion) could be amplified. If only SNPs differentiated the virulent and avirulent alleles, primers were designed in such a way that these variant positions were located at the 3’ extremity to maximize the specificity of amplification. Regions with two or more SNPs were preferentially selected to increase the allelic specificity. The primers were then synthesized by Thermo-Fisher Scientific (Waltham, Mass., USA). The details of the nine pairs of primers are presented in Table 1.
Validation of primer specificity
A simple PCR was first conducted with each of the primer pairs individually to ascertain their specificity. The PCR reaction was performed in a T Professional Thermocycler (Biometra, Dublin, Ireland) and was carried out in a reaction volume of 20 µl. Each primer was diluted at a concentration of 0.25 µM. The One Taq NEB (New England Biolabs,
(Ipswich, Massachusetts, United States)was used at 0.025 U/µl with 2 µl of DNA extracted
from P. sojae at a concentration of 10 ng/µl, 5X One Taq Standard reaction buffer (New
England Biolabs, Ipswich, Massachusetts, USA), 0.2 mM of dNTPs and 2.5% of DMSO
(Sigma, Saint-Louis, Missouri, United States). The PCR reaction conditions were as
follows: an initial denaturation at 94°C for 5 min, followed by 30 cycles of denaturation at 94°C for 30 sec, annealing at 60°C for 30 sec, elongation at 68°C for 1 min, and a final elongation at 68°C for 5 min. DNA fragment analysis was performed using a QIAxcel Advanced System on a DNA high resolution cartridge, based on method OH500 with alignment markers of 15 and 3000 bp according to the manufacturer’s instructions (Qiagen, Hilden, Germany). A PCR was performed on each of the 31 isolates of P. sojae with a known pathotype to validate that the presence of the expected amplicon was associated with an avirulent response.
Optimization of the multiplex PCR
Following optimization of primer concentration, annealing temperature and dNTP concentration, primers were mixed together in a single PCR reaction to check their compatibility in a multiplex PCR. It was found that the primers amplifying the Avr1c gene
were formed. Attempts to design alternative sets of primers were unsuccessful, so it was decided that the primers for Avr1c would be used in a separate assay in parallel with the multiplex assay. The multiplex PCR therefore contains the following eight primer sets: Avr1a-indel, Avr1a-snp1, Avr1a-snp2, Avr1b, Avr1d, Avr1k, Avr3a and Avr6.
The optimal number of cycles for the reaction was 40 cycles. Furthermore, a temperature gradient revealed that the temperature allowing obtaining the most intense and most distinct bands was 55°C for the multiplex PCR reaction and 60°C for the uniplex PCR. The dNTP concentration chosen was 0.25 mM. The final PCR products were analyzed with the QIAxcel Advanced System (Qiagen, Hilden, Germany).
The PCR reactions were carried out in a reaction volume of 20 µl. Following several tests of dilution of the primers, each primer was diluted at the optimal concentration detailed in Table 1. The One Taq (New England Biolabs, Ipswich, Massachusetts, États-Unis) was used at 0.025 U/µl with 2 µl of DNA at a concentration of 10 ng/µl, 5X One Taq Standard reaction buffer (New England Biolabs, Ipswich, Massachusetts, USA), 0.25 mM of dNTPs and 2.5% of DMSO (Sigma, Saint-Louis, Missouri, United States). The multiplex PCR conditions consisted in an initial denaturation at 94°C for 5 min, followed by 40 cycles of denaturation at 94°C for 30 sec, annealing at 55°C for 30 sec, elongation at 68°C for 1 min, and a final elongation at 68°C for 5 min. For the uniplex PCR reaction (Avr1c), the conditions were the same, except for annealing at 60°C.
Detection limits of the multiplex PCR
To determine the lowest concentration of DNA at which the multiplex and the uniplex PCR worked, dilutions from 0.01 pg to 20 ng were tested with the two PCR conditions described above. It was determined that the PCR multiplex could detect a DNA concentration of up to 0.2 ng, while the primers tested individually could detect a DNA concentration of 0.2 pg.
Lebreton et al. (2018). For the hydroponic assay, zoospores were inoculated into a hydroponic system containing a nutrient solution diluted in water. Seven differential soybean lines were grown in the hydroponic system with a susceptible control (cultivar Harosoy), and the virulence profile of the isolate tested was determined on the basis of which Rps genes resulted in immunity. Phenotypic responses (resistance or susceptibility) were recorded 14 days post inoculation. Phenotyping results were then compared to results obtained with the multiplex PCR assay.
Results
Gene-specific PCR-based markers for seven Avr genes in Phytophthora sojae
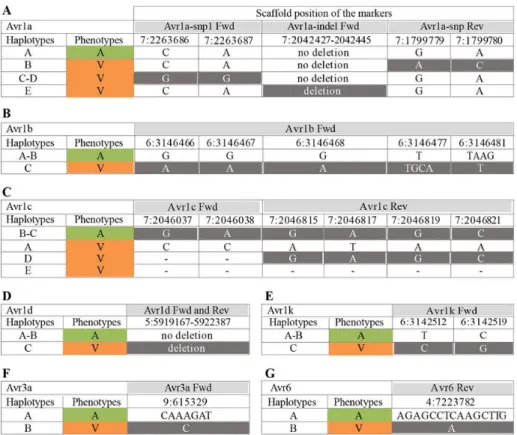
For all seven Avr genes under study, all primers were designed to amplify sequences associated with the avirulent allele of the genes (Fig. 1). In some cases, markers defined by Arsenault-Labrecque et al. (2018) were used directly, and in cases where markers were not discriminant enough for PCR (e.g. a single SNP), additional markers associated with the same alleles were used for the design of the primers. In other cases where the discriminant variants were located outside of the coding region, the primers were developed based on the specific haplotype linked to the avirulent allele. The positions of all the amplified regions are shown in Table 1.
Table 1. Primer sequences, with their genomic positions, amplicon size and concentrations, used for the detection of seven Avr genes in the Phytophthora sojae genome
Primer names Amplicons position*
Product
size Forward sequence
‡ Reverse sequence Concentrations F and
R primers (µM) Avr1a-indel Scaffold_7:2,042,431-2,042,664 234 GAAAGTGGACGGATATTTTCAAC CAAGGACGGACTGGTACAGA 0.100 Avr1a-snp1 Scaffold_7: 2,263,667-2,263,879 213 CTTAGTGTGCACCAACAGCCA ACCACACTTCACGGAGCATT 0.100 Avr1a-snp2 Scaffold_7: 1,799,519-1,799,796 278 GCTTTTCATCCAACGCTCAT AATGATTGGCGGCAGATC 0.150 Avr1b Scaffold_6:3,146,464-3,146,866 403 AAGGGGTACAGCCTGGATAAG CTTGCGCTGTGAAGTGTCAT 0.150 Avr1c Scaffold_7:2,046,020-2,046,821 802 CGGCAGAAGTTCTGGAAGA GCCTTCCTTTGTCAGATTCG 0.250 Avr1d Scaffold_5:5,919,385-5,919,881 497 CACGAGCAATGTCCTGTACG CGAGCGTCCGATTTATAACTGG 0.075 Avr1k Scaffold_6: 3,142,499-3,142,801 303 CTGTTCAGAAACTTCCGGTGC CATGAAAAAGTCGGGGTTTG 0.150 Avr3a Scaffold_9: 615,324-615,930 607 CTAGGCAAAGATGTCACCTG ATCATGGCAAGCACCAATCT 0.100 Avr6 Scaffold_4: 7,223,071-7,223,796 726 GTCGTGCTGCATACTCTTGG CAAGCTTGAGGCTCTGTGCT 0.100
For Avr1a, multiple variants were found on alleles conferring virulence on soybean lines carrying the Rps1a gene (Fig. 1A). One such variant was an 18-bp deletion conferring virulence to all isolates carrying it. For those virulent isolates lacking the deletion, they were distinguished from the avirulent ones on the basis of two adjacent SNPs, found in two separate regions (Avr1a-snp1 and Avr1a-snp2).
In the case of Avr1b, a combination of SNPs and indels, located within 15 bp of each other, was found to discriminate the avirulence allele (Fig. 1B). A forward primer was thus designed in that region to encompass all five variants.
The Avr1c avirulent allele could be discriminated from the virulent form on the basis of two SNPs situated at the 3' end of the forward primer, and four SNPs positioned at the 5' end of the reverse primer (Fig. 1C). This design allowed to target specifically the avirulent haplotypes against several other haplotypes linked to virulence.
In the case of P. sojae isolates carrying Avr1d, they were easily distinguished from those with pathotype 1d on the basis of a complete deletion of the gene (Fig. 1D). Primers were thus simply designed to amplify a region within the gene.
For Avr1k, two SNPs were selected within 7 bp of each other that discriminated the avirulent allele from the virulent one to design the primers (Fig. 1E).
Based on two distinct haplotypes, the avirulent allele of Avr3a presented an extra sequence of six nucleotides (Fig. 1F). This area was therefore selected to design discriminant primers.
Finally, a 15-bp deletion upstream of Avr6 was consistently observed in all virulent isolates (Fig. 1G). As it was consistently associated with a phenotype of virulence, it was used for primer design.
Figure 1 Discriminant haplotypes associated with distinct phenotypes in seven avirulence genes of Phytophthora sojae used to build discriminant primers. (A) Avr1a, (B) Avr1b, (C) Avr1c, (D) Avr1d, (E) Avr1k, (F) Avr3a, and (G) Avr6. A = avirulent and V = virulent. Haplotype letters refer to those previously identified by Arsenault-Labrecque et al. (2018). Scaffold positions refer to the position of the markers used as per the Phytophthora sojae reference genome v3.0.
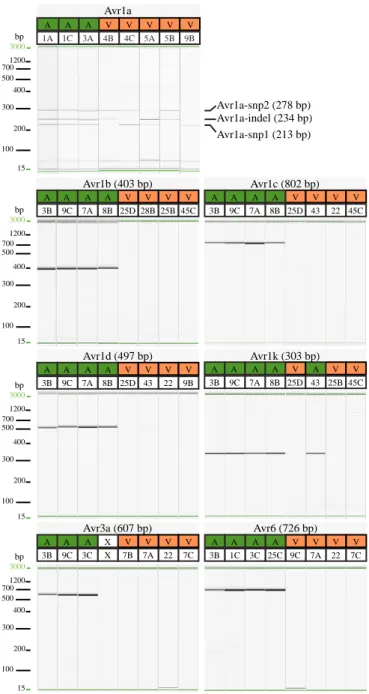
Uniplex PCR amplification and specificity
The results showed that successful amplification of the functional version of the Avr genes matched perfectly the expected phenotype for each of the seven Avr genes, thus confirming the specificity of the primers for the targeted region (Fig. 2). For six of the seven genes, a single set of primers was sufficient to discriminate the haplotypes leading to a virulent or
Figure 2 Gel images of uniplex PCR amplifications of discriminant regions associated with avirulence alleles for seven Avr genes in Phytophthora sojae. For each Avr gene, the expected phenotype (A = avirulent and V = virulent) against the corresponding Rps gene for each of the eight isolates tested is indicated at the top of the gel. The expected size of the amplicon is noted in parentheses next to each Avr gene, except for Avr1a. For Avr1a, three distinct regions, Avr1a-indel, Avr1a-snp1 and Avr1a-snp2, must amplify to detect a phenotype of avirulence.
FIGURE 2. CDB et. al.
A A A A V V V 3B 9C 7A 8B 25D 28B 25B 45C V Avr1b (403 bp) 3000 1200 700 500 400 300 200 100 15 bp A A A A V V V 3B 9C 7A 8B 25D 43 22 45C V Avr1c (802 bp) 3000 1200 700 500 400 300 200 100 15 bp A A A A V V V 3B 9C 7A 8B 25D 43 22 9B V Avr1d (497 bp) A A A A V A V 3B 9C 7A 8B 25D 43 25B 45C V Avr1k (303 bp) 3000 1200 700 500 400 300 200 100 15 bp A A A X V V V 3B 9C 3C X 7B 7A 22 7C V Avr3a (607 bp) A A A A V V V 3B 1C 3C 25C 9C 7A 22 7C V Avr6 (726 bp) 3000 1200 700 500 400 300 200 100 15 bp Avr1a-snp1 (213 bp) 1A 1C 3A 4B 4C 5A 5B 9B A A V V V V Avr1a-snp2 (278 bp) Avr1a-indel (234 bp) V Avr1a A
Multiplex PCR and specificity
Following optimization of the PCR conditions, the molecular assay was carried out in two parallel runs: one multiplex assay for the detection of Avr1a, Avr1b, Avr1d, Avr1k, Avr3a and Avr6, and one singleplex assay for Avr1c. The presence of a band, or three in case of Avr1a, of a specific size as described in Table 1 indicates that the tested isolate carries the avirulent allele associated with the amplicon of the Avr gene of that size. Conversely, the absence of an amplicon for a given gene indicates that the isolate carries the corresponding virulence allele. For instance, Fig. 3 presents results from the multiplex PCR assay on the 31 known isolates with their corresponding pathotype based on a phenotypic assay (Arsenault-Labrecque et al., 2018). Results show that the pathotype, as expressed by the absence of an amplicon for a given gene, is accurately predicted by the molecular assay. As an illustration, isolate 1A shows amplicons for Avr1a, 1b, 1k, 3a and 6 (Fig. 3A) and none for 1d and 1c (Fig. 3B), which translates into pathotype 1c and 1d.
Figure 3 Gel images of multiplex PCR amplifications of discriminant regions associated with avirulence alleles for seven Avr genes in Phytophthora sojae. (A) Results obtained with 31 isolates with a known pathotype, as indicated at the bottom of the gel, for Avr1a, 1b, 1d, 1k, 3a and 6. Expected size of the amplicon for each Avr gene is indicated on the right. (B) Complementary gel of PCR amplification of discriminant region associated with the avirulence allele for Avr1c (right) along with results obtained for the 31 isolates (A = avirulent and V = virulent) where A or V indicates presence or absence of the amplicon, respectively. For each isolate, the pathotype should correspond to the absence of an amplicon for each corresponding gene.
A A V 1A V A 3B 4B 5B 7B 8B 9B 25B 28A 43 45C V V V V V A A A A 1C V 3C 4C 5C 7C 8C 9C 25C 28B 45A V V V V A A A A A 3A 4A 5A 7A 8A 9A 22 25D 28C 45B V V V V V A A A A A B 15 bp 3000 1200 400 200 500 300 100 600 800 2000 Avr1c Actin 1c, 1 d 1c 1c 1a 1a 1a, 1 c 1a, 1 c 1a, 1 c 1a, 1 c, 6 1a, 1 c 1a, 6 1a, 3 a, 6 1a, 3 a, 6 1a, 3 a, 6 1a, 6 1a, 6 1a, 1 c, 1d 1a, 6 1a, 1 c, 1d Pathotype Pathotype bp 3000 1200 400 200 15 500 300 100 600 800 2000 Avr3a Avr1d Avr1b Avr1k Avr1a-snp2 Avr1a-indel Avr1a-snp1 Avr6 1A 1C 3A 3B 3C 4A 4B 4C 5A 5B 5C 7A 7B 7C 8A 8B 8C 9A 9B bp 3000 1200 400 200 15 500 300 100 600 800 2000 Avr3a Avr1d Avr1b Avr1k Avr1a-snp2 Avr1a-indel Avr1a-snp1 Avr6 25B 25C 22 25D 28A 28B 28C 43 45A 45B 45C 9C 1a, 6 1a, 1 c, 3 a, 6 1a, 1 c 1a 1a, 1 b, 1 k 1a 1a, 1 c, 1d 1a, 1 c, 6 1a, 1 b, 1 k, 6 1a, 1 b, 1 c 1d, 1 k 1a, 1 b, 1 c 1d, 1 k 1a, 1 b, 1 c 1d, 1 k
Genotyping and phenotyping of isolates with unknown pathotype
After validation of the multiplex PCR assay with the 31 known isolates, 25 uncharacterized isolates were randomly selected to confirm the effectiveness of the assay. Representative results obtained following the molecular and hydroponic assays are presented for two isolates (Fig. 4). As seen in Fig. 4A, the presence of amplicons for Avr1b, Avr1d and Avr1k on the gel is indicative that isolate 2012-82 should have pathotype 1a, 1c, 3a, 6. When compared with the bioassay (Fig. 4B), the phenotypes obtained clearly corroborated the molecular assay where a compatible interaction was observed between the isolate and differentials Rps1a, 1c, 3a and 6. In the other example with isolate 2012-156 (Fig. 4C), the molecular assay showed amplification for Avr1a (Avr1a-snp1, Avr1a-indel, Avr1a-snp2), 1b, 1k, 3a and 6, which leads to a diagnostic of pathotype 1c, 1d for that isolate. Interestingly, the phenotypic assay shown in Fig. 4D confirmed the compatible interaction with differentials Rps1c and 1d but also suggested one with Rps3a, in spite of the molecular assay clearly showing an amplicon for Avr3a. As a matter of fact, when results were combined for all 25 isolates and seven Avr genes (175 interactions), there was only a single and similar discrepancy when the molecular assay and the phenotypes did not match perfectly (Table 2). Indeed, in five cases, a compatible interaction was observed with Rps3a in the hydroponic assay, in spite of the presence of an amplicon for the avirulent allele of Avr3a. All other interactions generated a perfect match between the molecular and the phenotyping assay for a prediction accuracy of 97% (170/175).
Figure 4 Comparison of molecular and phenotyping assays to determine the pathotypes of Phytophthora sojae isolates. (A) Gel image of multiplex PCR amplifications of discriminant regions associated with avirulence alleles for seven Avr genes in P. sojae isolate 2012-82. Presence of amplicons for Avr1b, 1d and 1k predicts a pathotype 1a, 1c, 3a and 6. (B) Phenotyping results for isolate 2012-82 indicates a compatible interaction with Harosoy (no Rps), Rps1a, Rps1c, Rps3a and Rps6 and an incompatible interaction with Rps1b, Rps1d and Rps1k thereby assessing a
M u lt ip le x A vr 1c bp 3000 1200 500 400 300 100 15 600 800 2000 200 Avr1c Avr6 Avr3a Avr1d Avr1b Avr1k Avr1a-snp2 Avr1a-snp1 Avr1a-indel A B Avr1c Avr6 Avr3a Avr1d Avr1b Avr1k Avr1a-snp2 Avr1a-snp1 Avr1a-indel bp 3000 1200 500 400 300 200 100 15 600 800 2000 M u lt ip le x A vr 1c C D
pathotype 1a, 1c, 3a and 6, similar to the molecular assay. (C) Gel image of multiplex PCR amplifications of discriminant regions associated with avirulence alleles for seven Avr genes in P. sojae isolate 2012-156. Presence of amplicons for Avr1b, 1k, 3a, 6 and all three amplicons for Avr1a predicts a pathotype 1c and 1d. (D) Phenotyping results for isolate 2012-156 indicates a compatible interaction with Harosoy (no Rps), Rps1c, Rps1d and Rps3a and an incompatible interaction with Rps1a, Rps1b, Rps1k and Rps6 thereby assessing a pathotype 1c, 1d, and 3a, with 3a being the only interaction at odds with the molecular assay.
Table 2. Comparative results of predicted pathotypes between the molecular assay and the hydroponic assay for 25 isolates of Phytophthora sojae
Isolate Predicted pathotype
(Molecular assay) Observed pathotype § (Hydroponic assay) 2010-29 1a, 1c 1a, 1c, 3a 2010-32 1a, 1c 1a, 1c 2010-42 1a, 1c 1a, 1c 2010-44 1a, 1c 1a, 1c, 3a 2011-35 1a, 1c, 3a 1a, 1c, 3a 2011-40 1a, 1b, 1c, 1k 1a, 1b, 1c, 1k
2012-01 1a, 1c, 3a, 6 1a, 1c, 3a, 6
2012-120 1a, 1c, 6 1a, 1c, 6 2012-127 1a, 1c 1a, 1c 2012-136 1a, 1c, 6 1a, 1c, 6 2012-156 1c, 1d 1c, 1d, 3a 2012-40 1a, 1c, 6 1a, 1c, 6 2012-57 1a, 1c, 1d 1a, 1c, 1d 2012-76 1a, 1c 1a, 1c, 3a
2012-82 1a, 1c, 3a, 6 1a, 1c, 3a, 6
2016-20 1a, 1b, 1c, 1d, 1k 1a, 1b, 1c, 1k, 1d 2018-01 1a, 1c, 1d, 3a 1a, 1c, 1d, 3a 2018-02 1a, 1b, 1c, 1d, 1k 1a, 1b, 1c, 1d, 1k 2018-03 1a, 1b, 1c, 1d, 1k 1a, 1b, 1c, 1d, 1k, 3a 2018-04 1a,1b, 1c, 1d, 1k, 3a 1a, 1b, 1c, 1d, 1k, 3a 2018-05 1a, 1c, 1d, 3a 1a, 1c, 1d, 3a 2018-06 1a, 1b, 1c, 1d, 1k 1a, 1b, 1c, 1d, 1k