HAL Id: hal-02809405
https://hal.inrae.fr/hal-02809405
Submitted on 6 Jun 2020HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Impact du travail du sol sur l’abondance et la
biodiversité des communautés microbiennes
Xavière Brivet
To cite this version:
Xavière Brivet. Impact du travail du sol sur l’abondance et la biodiversité des communautés microbi-ennes. [Stage] Université de Bourgogne (UB), FRA. 2012, 25p. �hal-02809405�
UMR 1347 Agroécologie/AgroSup/INRA/uB Plateforme GenoSol 17 rue de Sully - BP 86510 21065 DIJON Cedex Master 1 Sciences de l’Environnement
Université de Bourgogne 4 boulevard Gabriel 21000 DIJON
RAPPORT DE STAGE
Impact du travail du sol sur l’abondance et
la biodiversité des communautés
microbiennes
Encadrants :
Amandine VIOLLET
Lionel RANJARD
Xavière BRIVET
Promotion 2011/2012
REMERCIEMENTS
Je voudrais tout d’abord remercier Philippe Lemanceau, directeur de l’UMR Agroécologie pour m’avoir permis d’effectuer ce stage. Un grand merci à Lionel Ranjard, responsable de la plateforme GenoSol, pour avoir accepté mon entrée au sein de l’équipe et pour m’avoir proposé un sujet aussi intéressant.
Mille mercis à Amandine Viollet, ma tutrice de stage, pour tout ce qu’elle a fait pour moi : pour sa précieuse aide dans la rédaction du rapport, pour avoir permis une intégration aussi facile au sein de l’équipe, pour m’avoir invité à une soirée bowling, pour sa patience face à une novice, pour tous ses conseils et tout ce qu’elle m’a appris, pour sa confiance et pour sa bonne humeur. Je remercie également Samuel Dequiedt pour son aide dans l’élaboration de ce rapport, Tiffanie Regnier et Mélanie Lelièvre pour leur accueil et leur sympathie et Pierre Plassart pour tous ses conseils aussi bien en anglais qu’en français. J’adresse tous mes remerciements à l’équipe de prélèvement qui a rendu possible la réalisation de ce stage.
Je pense aussi à l’ensemble de l’équipe et des gens que j’ai pu côtoyer : Julie, Vivi, Anne-Laure, PAM, Vince, Le geek, Bébé geek, Flo, Fafa, Fabiola, Yoyo…. pour leur accueil, leur gentillesse, qui ont facilité mon intégration sans laquelle le stage ne se serait pas aussi bien déroulé et pour avoir mangé mes gâteaux en me disant qu’ils étaient bon (ou en faisant semblant…). Une attention particulière à Emilie, pour nos discussions, son écoute attentive et pour nos futurs footing…
Je souhaite remercier les secrétaires, Catherine Morel et Fabienne Garcia, pour s’être occupée si gentiment des formalités administratives.
Et enfin, une pensée pour mes camarades de bureau qui ont partagé mes galères de stagiaire : Arnaud (et sa coupe "M"), Vivien et Elina.
LISTE DES ABRÉVIATIONS
ADN : Acide DésoxyriboNucléique
ARISA : Automated Ribosomal Intergenic Spacer Analysis BBT : Bleu de BromoPhénol
BET : Bromure d’EThidium
CIPAN : Culture Intermédiaire Piège à Nitrates EA : Environnement et Agronomie
EDTA : acide Ethylène Diamine Tétra Acétique
EFPA : Ecologie des Forêts, Prairies et milieux Aquatiques
EPST : Etablissement Public à caractère Scientifique et Technologique FAM : France Agri Mer
GIS GC-HP2E : Groupement d’Intérêt Scientifique Grande Culture à Hautes Performances Economique et Environnementale
INRA : Institut National de la Recherche Agronomique PCR : Polymerase Chain Reaction
PVPP : Poly Vinyl Poly Pyrrolidone SDS : Sodium Dodécyl Sulfate
SPE :Santé des Plantes et Environnement TBE : Tris, Borate, EDTA
UMR : Unité Mixte de Recherche UE : Unité Expérimentale
SOMMAIRE
1. Présentation des laboratoires d’accueil __________________________________________ 1 1.1 ARVALIS, Institut du Végétal ________________________________________________________ 1 1.2 Institut National de la Recherche Agronomique ________________________________________ 1 1.3 UMR 1347 Agroécologie ___________________________________________________________ 2 1.4 Plateforme GenoSol _______________________________________________________________ 3 2. Contexte scientifique _________________________________________________________ 3 3. Objectifs du travail de recherche ________________________________________________ 5 4. Matériels et méthodes ________________________________________________________ 6 4.1 Site expérimental et échantillonnage _________________________________________________ 6 4.2 Tamisage et stockage des échantillons ________________________________________________ 9 4.3 Détermination du taux d’humidité __________________________________________________ 10 4.4 Mesures de pH __________________________________________________________________ 11 4.5 Lyophilisation ___________________________________________________________________ 11 5. Extraction d’ADN ___________________________________________________________ 11 5.1 Obtention de la banque des lysats __________________________________________________ 12 5.2 Obtention de la banque des ADNS bruts (non purifiés) __________________________________ 14 5.3 Quantification des ADN bruts ______________________________________________________ 15 6. Statistiques ________________________________________________________________ 16 7. Premiers résultats et Discussion _______________________________________________ 17 7.1 Mesures de pH __________________________________________________________________ 17 7.2 Mesure d’humidité _______________________________________________________________ 17 7.3 Quantification des ADNs __________________________________________________________ 18 8. Conclusion et perspectives de travail ___________________________________________ 20 BIBLIOGRAPHIE ________________________________________________________________ 22 ANNEXES _____________________________________________________________________ 24INDEX DES FIGURES
Figure 1 - Vue aérienne de l’essai Environnement ... 6
Figure 2 - Exemple d’une parcelle de l’essai Environnement ... 7
Figure 3 - Description des différents traitements choisis ... 7
Figure 4 - Disposition des parcelles des traitements choisis au sein de l’essai Environnement . ... 7
Figure 5 - Carottier ... 8
Figure 6 - Points de prélèvements ... 8
Figure 7 - Résumé des différentes étapes de préparation des échantillons ... 9
Figure 8 - Présentation des différentes étapes du tamisage des échantillons ... 9
Figure 9 -Congélateurs -40°C de la plateforme GenoSol permettant de conserver les échantillons de sol ... 10
Figure 10 - Lyophilisation des échantillons de sol ... 11
Figure 11 - Préparation des tubes pour la lyse mécanique ... 12
Figure 12 - Appareil permettant de filtrer le tampon d’extraction ... 13
Figure 13 - Agitateur FastPrep-24 ... 13
Figure 14 - Tubes après l’agitation au FastPrep-24 ... 14
Figure 15 - Gel d’électrophorèse avant dépôt ... 15
Figure 16 - Gel d’électrophorèse après migration ... 16
Figure 17 - Enceinte à ultraviolets . ... 16
Figure 18 - Moyenne des pH par traitement ... 17
Figure 19 - Moyenne des taux d’humidité en % . ... 18
Figure 20 - Exemple d’un gel d’électrophorèse obtenu après migration des ADN ... 18
Figure 21 - Moyenne de la biomasse moléculaire en μg d’ADN/g de sol sec ... 19
INDEX DES TABLEAUX
Tableau 1 - Présentation des pôles et plateformes de l’UMR Agroécologie ... 21. Présentation des laboratoires d’accueil
Le travail effectué au cours de ce stage rentre dans le cadre d’une collaboration entre ARVALIS, Institut du Végétal et la plateforme GenoSol de l’Institut National de la Recherche Agronomique de Dijon. Ce projet scientifique est financé par France Agri Mer (FAM) et le Groupement d’Intérêt Scientifique Grande Culture à Hautes Performances Economique et Environnementale (GIS GC-HP2E).
1.1 ARVALIS, Institut du Végétal
ARVALIS est un organisme de recherche appliquée, produisant des références technico-économiques et agronomiques directement applicables dans les systèmes de production. Cet institut compte 35 sites sur l’ensemble du territoire national. Ces derniers sont répartis en stations de recherche, sites expérimentaux, fermes d’application et divers laboratoires.
Le champ d’activité de l’institut concerne 80% de la superficie agricole française et couvre les techniques agricoles de production, de récolte, de stockage et de conservation. L’institut a pour objectifs d’augmenter la compétitivité des exploitations, d’ouvrir des débouchés, d’améliorer la rentabilité et de satisfaire au mieux les consommateurs et les citoyens.
1.2 Institut National de la Recherche Agronomique
L’Institut National de la Recherche Agronomique (INRA) est un organisme public français de recherches finalisées en agronomie. Depuis 1984, il porte le statut d’Etablissement Public à caractère Scientifique et Technologique (EPST). Aujourd’hui, cet institut est placé sous la double tutelle du ministère de l’enseignement supérieur et de la recherche et du ministère de l’agriculture et de la pêche. Deuxième institut de recherche agronomique dans le monde, premier en Europe, il est réparti en 21 centres régionaux. L’INRA mène des recherches à l’échelle locale et planétaire. Elles ont pour but de préserver l’environnement pour une agriculture compétitive et durable, respectueuse des territoires et des ressources naturelles. Pour cela, il met en œuvre un réseau de partenariat tout en veillant à préserver le bien public et partager ses connaissances avec un large public.
L’INRA est présent en Bourgogne depuis 1946. Le centre de Dijon, qui gère 40 millions d’euros, comporte 4 thématiques de recherche : (i) aliment, sensorialité et comportement
alimentaire, (ii) agroécologie, (iii) territoire et développement et (iv), écologie des systèmes aquatiques et microbiens. Le centre comprend environ 600 personnes, dont la moitié de chercheurs, doctorants et post-doctorants. Il est composé de 4 UMR (Unité Mixte de Recherche), 2 UR (Unité de Recherche) et 1 UE (Unité Expérimentale) :
- UMR Agroécologie,
- UMR Centre des Sciences, du Gout et de l’Alimentation,
- UMR Centre d'Economie et Sociologie Appliquées à l'Agriculture et aux Espaces Ruraux, - UMR Centre Alpin de Recherche sur les Réseaux Trophiques des Ecosystèmes Limniques, - UR Laboratoire de recherche sur les innovations socio-techniques et organisationnelles en
agriculture,
- UR Technologie et Analyses laitières, - UE Domaine expérimental.
Les unités sont réparties sur 5 sites, principalement en Bourgogne mais aussi en Franche-Comté et Rhône-Alpes. On compte 12000 m² de laboratoires, 2000 m² de serres et chambres climatisées et un domaine expérimentale de 120 hectares.
1.3 UMR 1347 Agroécologie
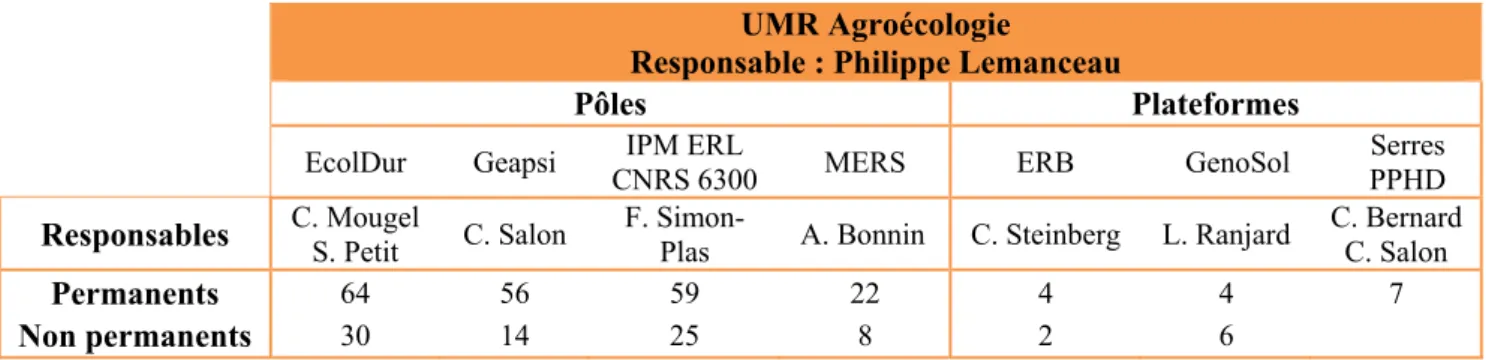
L’UMR Agroécologie, dirigée par Philippe Lemanceau, a été récemment mise en place en Janvier 2012 dans le but de regrouper plusieurs unités. Multipartenaires (AgroSup, INRA, Université de Bourgogne), elle comprend environ 250 personnes organisées autour de 4 pôles et 3 plateformes (Tableau 1).
Tableau 1 – Présentation des pôles et plateformes de l’UMR Agroécologie. UMR Agroécologie
Responsable : Philippe Lemanceau
Pôles Plateformes
EcolDur Geapsi CNRS 6300 IPM ERL MERS ERB GenoSol PPHD Serres
Responsables C. Mougel S. Petit C. Salon F. Simon-Plas A. Bonnin C. Steinberg L. Ranjard C. Bernard C. Salon
Permanents 64 56 59 22 4 4 7
Non permanents 30 14 25 8 2 6
Son objectif est d’enrichir le domaine agronomique par de nouvelles connaissances des processus écologiques. Un des grand axe de recherche de cette UMR est l’étude des interactions et régulations entre organismes à différents niveaux d’intégration (communautés, populations,
organismes, cellules et molécules) et à différentes échelles spatio-temporelles (parcelles, paysages, territoire, cycles de cultures, rotations…).
1.4 Plateforme GenoSol
La plateforme GenoSol a été créée en 2008 par l’INRA avec le soutien des départements scientifiques : Environnement et Agronomie (EA), Santé des Plantes et Environnement (SPE) et Ecologie des Forêts, Prairies et milieux Aquatiques (EFPA). GenoSol est un centre de ressources biologiques unique en France et en Europe, dédié à la conservation et à l’analyse des ressources génétiques des communautés microbiennes du sol.
La plateforme a pour objectif de centraliser les ressources génétiques microbiennes des sols, de les mettre à disposition de la communauté scientifique ainsi que d’optimiser des technologies de pointe pour en assurer la caractérisation des populations microbiennes. Dans un contexte de développement durable, cette infrastructure est essentielle en tant que conservatoire du patrimoine génétique des sols et plateforme technologique.
L’objectif est de fournir une structure logistique assurant l’acquisition, la conservation, la caractérisation et la mise à disposition des ADN de sols (Acides DésoxyriboNucléiques) issus d’échantillonnages de grande envergure (réseau de surveillance, sites expérimentaux, zones ateliers… ; Annexe 1 et 2). Cette plateforme a pour mission d’établir un référentiel d’interprétation des caractérisations moléculaires microbiennes (abondance, densités, diversités taxonomiques et fonctionnelles…) sur les sols ainsi que la définition et la connaissance de la gamme de variation de ces indicateurs dans le temps et dans l’espace. L’objectif est de conférer un caractère plus générique aux études portant sur l’évaluation des ressources génétiques des sols et d’améliorer la prise en compte de la composante microbienne dans la gestion durable des sols.
2. Contexte scientifique
Le sol est la couche superficielle de la croûte terrestre. Formé d'une fraction minérale et de matière organique (humus), il se forme par des processus très lents. Les sols acquièrent progressivement des structures qui leur sont propres une fois soumis à l'action des agents physiques et chimiques (vent, précipitations, variations de température…) et biologiques (végétations, macro et microorganismes du sol…).
Les microorganismes représentent la majorité des organismes vivants du sol. Il a été estimé qu’un gramme de sol contenait de 1010 à 1011 bactéries (Horner-Devine et al., 2003), de 6000 à 50000 espèces bactériennes (Curtis et al., 2002) et jusqu’à 200 mètres d’hyphes fongiques (Leake et
al., 2004). Les microorganismes sont les organismes du sol les plus diversifiés taxonomiquement et
fonctionnellement (Torvisk et al., 2002) et constituent une part importante de la diversité génétique de la planète. Le sol représente la 3ème frontière biotique avec une biodiversité correspondant à 25% de la biodiversité totale. Néanmoins, la totalité des espèces est loin d’être connue. En effet, 90 % des espèces sont encore ignorées (Wall et André, 2002). De plus, il existe une relation positive entre biodiversité et productivité ainsi qu’entre biodiversité et stabilité montré par le concept d’assurance écologique pour les écosystèmes (Loreau et al., 2000).
Les microorganismes telluriques jouent un rôle fondamental dans la structure du sol (Rilli et Mummey, 2006) mais aussi dans son fonctionnement global. En effet, la plupart des bactéries ont un rôle important dans les divers cycles géochimiques comme celui de l’azote (Kowalchuk et Stephen, 2001) ou du carbone (Högberg et al., 2001) et en particulier dans les processus d’oxydo-réduction (Clark, 1989). La biomasse microbienne est essentielle dans de nombreux processus telle que l’humification direct (par transformation de la biomasse morte) et indirect (par l’action enzymatique qu’elle exerce au cours des différentes étapes de l’humification). Les bactéries hétérotrophes et saprophytes par exemple, décomposent les celluloses et les sucres, constituant ainsi des sources d’énergie. Les bactéries autotrophes oxydent certains composés et assimilent le carbone du CO2. Les bactéries protéolytiques, quant à elles, hydrolysent les protéines et libèrent des acides aminés. Les actinomycètes et les champignons ont aussi un rôle non négligeable dans la décomposition des litières et donc dans les processus d’humification.
Enfin, les microorganismes du sol peuvent être impliqués dans diverses interactions avec les plantes. Ces interactions peuvent avoir des effets pathogènes mais aussi bénéfiques pour leurs hôtes végétaux. Les microorganismes interviennent notamment dans l’acquisition des éléments nutritifs par les plantes (Smith et Read, 2008 ; Sprent, 2001), améliorant ainsi leur croissance. De plus, certains microorganismes, tels que les champignons mycorhizogènes et les bactéries fixatrices d’azote, sont capables de mettre en place des interactions symbiotiques avec les plantes qui ont pour conséquences bénéfiques d’améliorer leur croissance et leur santé.
Au vu de la grande diversité des microorganismes et leur rôle essentiel dans de nombreux processus biochimiques et les interactions avec les plantes, il est donc important d’étudier l’impact
de l’environnement et des activités anthropiques sur ces populations. En effet, l’introduction des intrants de synthèse, les changements de pratiques agriculturales, les modifications climatiques, la pollution atmosphérique sont des pressions environnementales pouvant avoir un fort impact sur les communautés microbiennes du sol. De ce fait, ces dernières sont susceptibles d’intégrer ces stress affectant le sol. Elles apparaissent ainsi comme des bio-indicateurs précoces de l’évolution des sols (Cortet et al., 1999 ; Ranjard et al., 2010).
Actuellement, peu d’études génériques ont été réalisées pour développer ces bio-indicateurs, permettant de diagnostiquer l’état biologique des sols. L’opérationnalité passe par la connaissance des gammes de variations naturelles de ces bio-indicateurs et la définition de l’impact des activités anthropiques. L’application d’outils de caractérisation standardisés en routine sur les sites expérimentaux permet l’amélioration des connaissances ainsi que de la robustesse de ces indicateurs précoces de perturbations.
3. Objectifs du travail de recherche
L’objectif du projet scientifique est d’étudier les effets éventuels de différents systèmes de
culture sur les communautés microbiennes de la couche superficielle du sol (0-20 cm). Ainsi, ce projet propose de caractériser l’abondance, la structure et la diversité des communautés microbiennes des sols agricoles par le biais de calages méthodologiques et d’effets cumulatifs du régime du travail du sol. Le but est d’étudier l’impact de la modification de l’itinéraire cultural sur les composantes microbiennes. L’étude en cinétique sur une année (à cheval sur 2 cycles de culture) va permettre une meilleure connaissance de la variation temporelle de ces composantes et donc d’augmenter la robustesse des conclusions apportées. Ce projet permettra aussi d’étudier les capacités de résilience et d’analyser la stabilité annuelle biologique des sols. Cette étude est réalisée à partir de divers échantillons de sol prélevés dans les parcelles de l’essai Environnement du site ARVALIS de Boigneville.
L’objectif de ce stage, s’inscrivant dans le cadre de la première année du master "Science de
l’Environnement", est de compléter les résultats obtenus par la caractérisation plus fine de l’abondance microbienne. Il consiste à réaliser un échantillonnage stratifié qui sera mis en place en complément de l’échantillonnage effectué le 21 mai 2012. La stratification porte sur 2 horizons différents : 0-10 cm et 10-y cm (y étant la profondeur du labour initial) et sera appliquée sur toutes
les parcelles étudiées. L’objectif est de voir comment sont distribués les microorganismes dans la couche superficielle du sol.
4. Matériels et méthodes
4.1 Site expérimental et échantillonnage
Le site expérimental d’ARVALIS se situe à Boigneville, en Ile de France dans le département de l’Essonne (91).Le site expérimental d’ARVALIS est caractérisé, d’une manière générale, par un sol limoneux-argileux et brun calcique saturé.
L’essai Environnement (Figure 1) a débuté en 1991. Cet essai de longue durée a pour objectif initial la diminution des pertes en nitrates par lessivage. La gestion des résidus dans le cas du labour (déchaumage ou mulch) et des CIPAN (Culture Intermédiaire Piège à Nitrates ; crucifères ou légumineuses) permet de contrôler ces pertes en nitrates.
Figure 1 - Vue aérienne de l’essai Environnement.
L’essai Environnement de longue durée (21 ans) est divisé en plusieurs parcelles. Chaque parcelle a une longueur de 48 mètres et une largeur de 9 mètres (Figure 2).
Site ARVALIS
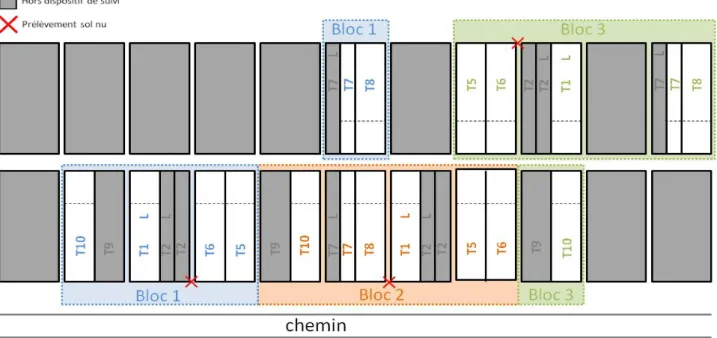
L’ensemble des parcelles correspond aux 10 traitements différents résultant du couplage de plusieurs modalités (CIPAN, travail du sol…). Parmi ces divers traitements, 6 ont été retenus pour ce stage : T1, T5, T6, T7, T8 et T10 (Figures 3 et 4). Pour chaque traitement, 3 répétitions de terrain (ou blocs) ont été mises en place.
Figure 2 – Exemple d’une parcelle de l’essai
Environnement.
Les zones entre les parcelles ont été maintenues en sol nu depuis le début du dispositif. Trois zones de prélèvements ont été choisies sur ce sol nu au sein de chaque bloc pour servir d’échantillons témoins (Figure 4).
Figure 3 – Description des différents traitements choisis.
Figure 4 - Disposition des parcelles des traitements choisis au sein de l’essai Environnement.
SN T1
Labour crucifère T5
CIPAN
crucifère puis légumineuse T6
SN T7
SD crucifère T8
CIPAN
L’échantillonnage a été réalisé le lundi 21 mai 2012 à l’aide d’un carottier (Figure 5) permettant de prélever 2 horizons : 0-10 cm et 10-y cm (y étant la profondeur de l’ancien labour différente selon les parcelles : 27,5 ou 28,5 cm). Au total, 42 échantillons ont été prélevés : (6 traitements + 1 sol nu) x 3 blocs x 2 horizons.
Figure 5 – Carottier.
Pour chaque échantillon correspondant à une parcelle, un pool de 5 prélèvements a été réalisé afin d’être représentatif de la surface d’échantillonnage. Ces prélèvements ont été effectués en W d’après la norme ISO 10381-1:2002(E), à 2 m de chaque bord et en évitant la zone où ont été placées des bougies poreuses (Figure 6). Il est nécessaire d’utiliser du matériel propre et lavé à l’eau au préalable. Entre chaque traitement, la terre restant sur le carottier doit être enlevée au maximum afin d’éviter la contamination du traitement suivant. Pour chaque échantillon de sol nu, un pool de 5 prélèvements a également été effectué. Ces prélèvements ont été réalisés dans un cercle de 2 m de diamètre (Figure 6).
Figure 6 - Points de prélèvements. A : parcelle traitée. B : sol nu.
Sur le terrain, les prélèvements sont conservés dans des sachets à zip de type sac de congélation. Une fois prélevés, les échantillons sont placés le plus rapidement possible dans une glacière avec des blocs froids afin de les maintenir dans des conditions de conservation adaptées. En attendant leur préparation, tous les échantillons sont ensuite placés à 4°C dès leur arrivée au laboratoire (Figure 7).
Figure 7 – Résumé des différentes étapes de préparation des échantillons.
4.2 Tamisage et stockage des échantillons
Le tamisage des échantillons est effectué le plus tôt possible après leur prélèvement afin d’éviter la dérive des communautés microbiennes et d’avoir un échantillon bien représentatif des conditions de prélèvement. Le tamisage permet une meilleure accessibilité des communautés microbiennes en cassant les éventuelles structures compactes et garantit la robustesse des analyses. L’ensemble des différentes étapes de préparation est présenté dans la figure 8. Pour chaque échantillon, il est nécessaire d’utiliser du matériel propre et de porter des gants neufs.
Figure 8 – Présentation des différentes étapes du tamisage des échantillons.
Dans un premier temps, la totalité de l’échantillon est transvasée dans un bac où les gros
agrégats de sol sont cassés à la main (étapes 1 et 2). Le sol est alors mélangé afin d’obtenir une répartition homogène. Pour les échantillons de l’horizon 10-y cm, le sol est tout d’abord quarté (séparé en 4 parties équivalentes) afin d’obtenir un sous-échantillon représentatif en quantité
1 2 3
4 5 6
suffisante pour la suite des analyses (étape 3). La totalité de ce sous-échantillon est tamisée à 4 mm qui correspond à un bon compromis entre faisabilité et destruction des structures les plus grosses (étape 4). En ce qui concerne les échantillons de l’horizon 0-10 cm, la quantité de sol prélevé étant faible, la totalité du prélèvement est tamisée.
Dans un deuxième temps, l’échantillon tamisé a été réparti de façon homogène sur un papier
kraft. Un pilulier de 50 mL en polypropylène est ensuite rempli en prélevant le sol sur l’intégralité des diagonales afin d’éviter de sélectionner une seule catégorie de particules de sol (étapes 5 et 6).
La quantité de sol introduite dans les piluliers est déterminée (étape 7). Afin de stabiliser la vie biologique, les piluliers étiquetés sont stockés à -40°C dans les congélateurs de la plateforme GenoSol (Figure 9).
Figure 9 – Congélateurs -40°C de la plateforme GenoSol permettant de conserver les échantillons de sol.
Le sol tamisé restant est alors replacé dans son sachet d’origine ouvert afin qu’il sèche à température ambiante en vue d’analyses physico-chimique ultérieures. Cette manipulation est réalisée sur l’ensemble des échantillons et de manière rigoureuse dans un souci d’homogénéité.
4.3 Détermination du taux d’humidité
La détermination du taux d’humidité a pour but de quantifier la teneur en eau contenu dans le sol au moment du prélèvement, permettant de donner une indication sur son état hydrique. Ce paramètre sera par la suite interprété en parallèle à la caractérisation de la biomasse microbienne.
Cette étape est réalisée une fois les gros agrégats cassés (Figure 8 : étapes 1 et 2). Pour chaque échantillon, 3 répétitions techniques sont réalisées. Dix grammes de sol sont introduits dans une boite en aluminium (M1 = masse du sol humide). Les boites sont ensuite placées dans une étuve à 105°C pendant 48 h. La masse de sol sec est ensuite déterminée (M2 = masse du sol sec) afin de mesurer la perte en eau. Enfin, le taux d’humidité est calculé en pourcentage d’après la formule suivante : ((M1 - M2) / M2 ) x 100.
4.4 Mesures de pH
Les mesures de pH sont réalisées à partir des échantillons de sol tamisés séchés à l’air libre. La procédure appliquée suit la norme ISO 10390.
Dans une bouteille propre de 100 mL, 10 mL de sol de chaque échantillon sont introduits et 50 mL d’eau distillée sont ajoutés avec une éprouvette propre. La suspension ainsi obtenue est agitée pendant 1 h à environ 20-25°C et 150-200 rpm. La mesure du pH est réalisée à l’aide d’un pHmètre sous agitation avec un barreau aimanté.
4.5 Lyophilisation
La lyophilisation permet de retirer l’eau des échantillons du sol sans passer par une décongélation ni un séchage classique conservant ainsi au mieux les caractéristiques des communautés microbiennes. La durée de la lyophilisation dépend de la quantité d’eau à extraire, de la température des échantillons, de la surface d’échange ainsi que des caractéristiques du sol.
Figure 10 – Lyophilisation des échantillons de sol.
Les piluliers contenant les échantillons congelés sont introduits dans le lyophilisateur, bouchons dévissés, pendant 48 h.
5. Extraction d’ADN
L’abondance des communautés microbiennes telluriques est mesurée grâce à des outils de biologie moléculaire développés, optimisés et standardisés au sein de la plateforme GenoSol. L’objectif est d’extraire l’ADN génomique total à partir de 2 g de sol sec. L’extraction aboutit à l’obtention d’ADN le moins dégradé possible respectant ainsi les standards définis par GenoSol. De
la qualité des ADN obtenus dépend l’application mais aussi la représentativité et la robustesse des caractérisations moléculaires des communautés pratiquées par la suite (PCR, qPCR, séquençage…).
D’une manière générale, l’extraction a lieu dans des conditions de propretés strictes en prenant soin de n’utiliser que du matériel stérile. L’eau ultra-pure ainsi que les diverses solutions utilisées seront également stérilisées par filtration ou autoclavage quand cela est possible. Deux étapes sont nécessaires pour extraire l’ADN : i) broyage et lyse permettant l’obtention d’une suspension de sol ou lysat et ii), déproténéisation et précipitation pour obtenir un ADN brut.
5.1 Obtention de la banque des lysats
L’extraction consiste en une lyse mécanique et chimique des échantillons de sol. La lyse mécanique permet, par l’action de billes en agitation, la fragmentation des particules de sol et la séparation des cellules biologiques accrochées à celles-ci. La lyse chimique est caractérisée par une dénaturation des protéines et des lipides par l’action à haute température d’un détergent, le SDS (Sodium Dodécyl Sulfate), permettant la lyse des membranes cellulaires.
Dans un tube de 15 mL sont introduits : 4 g de billes de 0,1 mm en silice, 5g de billes de 1,4 mm en céramique et 8 billes de 4 mm en verre (Figure 11). Un tube par échantillon est ainsi préparé et 2 g de sol sec sont introduits dans chacun des tubes. L’ensemble des pesées est réalisée à l’aide d’une balance de précision.
Figure 11 – Préparation des tubes pour la lyse mécanique.
Le tampon d’extraction est préparé et filtré à l’aide d’une membrane de 0,2 µm afin d’éliminer les contaminations microbiennes éventuelles (Tableau 2 et Figure 12).
Tableau 2 – Composition du tampon d’extraction pour un volume final de 10 mL.
Solution Volume Rôle
Tris-HCl 1M pH8 1 mL Tamponner la solution
EDTA 0,5 M pH8 2 mL Chélater la majorité des cations bivalents (Mg2+), cofacteur de nombreuses DNAses NaCl 1 M 1 mL Limiter les dénaturations partielles de l'ADN et libérer les interactions ADN-protéines. SDS 20% 1 mL Faciliter la lyse des membranes et la dénaturation des protéines
H20 ultra-pure 5 mL
Figure 12 – Appareil permettant de filtrer le tampon d’extraction.
Huit millilitres de tampon d’extraction filtré sont ajoutés dans chacun des tubes contenant les billes et le sol. Immédiatement après, les tubes sont vortexés afin d’homogénéiser la suspension et d’éviter la création d’un bouchon de sol. Les tubes sont ensuite disposés dans le FastPrep-24 (agitation en forme de 8) pendant 90 s à une vitesse de 4 m.s-1 (Figures 13 et 14).
Après l’agitation, les tubes sont vortexés puis incubés au bain-marie à 70°C pendant 15 min. Cette dernière étape est réalisée deux fois. Les tubes sont alors centrifugés à 7000 g (ou rcf) pendant 5 min à 18°C. La centrifugation sépare les éléments grossiers (particules de sol notamment) du surnageant (lysat) contenant des débris cellulaires, les protéines et l’ADN métagénomique de l’ensemble des microorganismes. Le lysat obtenu est alors récupéré : 1 µL de est stocké à 4°C dans l’attente de la 2ème étape qui doit avoir lieu dans les 24 à 48 heures et 4,5 mL de lysat sont conservés à plus long terme à -20°C .
5.2 Obtention de la banque des ADNS bruts (non purifiés)
L'ADN est séparé des protéines dénaturées par une déprotéinisation consistant à précipiter les protéines en présence d'une forte concentration en sel. Cette méthode présente deux avantages : les interactions ADN-protéines sont bien détruites et les produits ne sont pas toxiques (contrairement aux solvants organiques, phénol et chloroforme). Toute cette étape est réalisée au maximum dans la glace pour limiter au maximum la dégradation de l’ADN.
Cent microlitres d’acétate de potassium 3 M pH 5,5 sont ajoutés à 1 mL de lysat. Le mélange est homogénéisé par retournement puis incubé 10 min dans la glace. Cette étape permet la précipitation des protéines, le froid favorisant la formation de cristaux qui vont piéger les protéines. L’ensemble est centrifugé à 14000 g (ou rcf) pendant 5 min à 4 °C et le surnageant est délicatement récupéré à la pipette. Cette étape de centrifugation à haute vitesse permet d’obtenir un surnageant clair contenant l’ADN débarrassé des protéines en suspension.
L’ADN est ensuite précipité par ajout de 900 µL d’isopropanol à -20°C. Les échantillons sont placés pendant 30 min au minimum à -20 °C pour accélérer le processus. Une nouvelle centrifugation à 13000 rpm pendant 30 min à 4°C est réalisée. Le surnageant obtenu est éliminé par retournement des tubes et le culot contenant l’ADN est lavé avec 400 µL d’éthanol 70° à -20°C. Les tubes sont centrifugés à 13000 rpm pendant 5 min à 4°C. Enfin, l’alcool est éliminé à la pipette puis par évaporation dans une étuve à 60°C pendant quelques minutes.
Le culot d’ADN est resuspendu dans 200 µL d’H2O ultra-pure. L’ADN brut ainsi obtenu est conservé à -20°C pour assurer sa conservation en vue des analyses ultérieures. Cet ADN contient encore des éléments coextraits (sucres, macromolécules phénoliques, acides humiques…).
5.3 Quantification des ADN bruts
La quantification des ADN bruts correspond à la mesure de la biomasse moléculaire microbienne. Celle-ci correspond à un bon indicateur de l’abondance en microorganismes, sensible aux modifications et perturbations du milieu (Dequiedt et al., 2011). Une bonne biomasse est un garant d’un bon fonctionnement du sol.
Les ADN sont quantifiés par migration électrophorétique. La technique d’électrophorèse est dite séparative, elle est utilisée dans un but analytique. Le principe est de soumettre un mélange de molécules à un champ électrique ce qui entraine la migration des molécules chargées. Les acides nucléiques, chargés négativement, migrent vers la cathode c'est-à-dire le pôle positif. Les molécules d’ADN migrent sur le gel à une vitesse proportionnelle à leur taille en paires de bases et sont alors séparées des molécules plus grosses (débris cellulaires) et des plus petites (acides humiques).
Les ADN bruts sont dilués au 5ème dans de l’eau ultra-pure et 10 µL d’ADN dilué sont mélangés avec 5 µL de BBP 0,5% (Bleu de BromoPhénol). Le BBP est utilisé pour rendre le front de migration visible et alourdir l’ADN ce qui facilite le dépôt. Les échantillons sont déposés dans des puits de 1,5 mm d’épaisseur d’un gel d’agarose à 1% préparé dans du TBE 1X (TBE en entier ; Figure 15). La concentration en ADN est estimée en comparaison avec une gamme d'ADN de thymus de veau de concentration connue : 250 ; 125 ; 62,5 ; 31,25 et 15,62 ng d’ADN dans 10 µL. Comme pour les échantillons, 10 µL de chacun des points de la gamme sont mélangés avec 5 µL de BBP 0,5% puis déposés dans les puits du gel.
La migration est réalisée à 100 V pendant 1h30 environ. Après migration (Figure 16), le gel est révélé dans un bain de BET (Bromure d’EThidium) pendant 20 min puis lavé dans un bain d’eau distillée pendant 10 min. Le gel est placé sous les ultraviolets dans une enceinte fermée et une
image est prise à l’aide d’un appareil photo particulier (Figure 17). Les bandes d’acides nucléiques marquée au BET se trouvant dans le gel fluorescent et sont alors visibles. L’image du gel ainsi obtenue est analysée à l’aide d’un logiciel : Image Quant TL version 7.0.
Le BET est à manipuler avec une très grande précaution. En effet, le BET est un agent intercalant cancérigène qui supprime ou inhibe la synthèse d’ARN et de protéine dans de nombreuses cellules.
6. Statistiques
Les tests statistiques présents dans ce rapport, sont réalisés avec Excelstat. L’utilisation de tests statistiques va nous permettre de tester des hypothèses, de relever des différences ou des égalités entre des groupes de données relevées sur le terrain.
Le test de KrusKal-Wallis 5% a été choisi pour réaliser toutes les analyses statistiques de ce rapport de stage. Il s’agit d’un test non paramétrique, utilisé lorsque les distributions ne suivent pas une loi Normale et/ou lorsque qu’il n’y a pas homoscedasticité. Il permet la comparaison de plusieurs échantillons indépendants par la somme des rangs. L’hypothèse nulle de ce test correspond au fait que les médianes des deux distributions à comparer sont égales (les données des échantillons sont réparties de façon aléatoire induisant que les échantillons soient identiques). L’hypothèse alternative correspond quant à elle au fait que les médianes sont différentes.
7. Premiers résultats et Discussion
7.1 Mesures de pH
Le pH varie entre 5,4 et 7,4 (Figures 18). Le sol semble être légèrement plus acide en surface qu’en profondeur. Néanmoins il n’y a aucune différence significative de pH entre les deux horizons. De même, il n’y a aucune différence significative de pH selon les traitements. Les valeurs de pH ne semblent donc pas être influencées par les traitements. Ainsi, le labour, le semi-direct, la présence ou non de CIPAN n’ont pas de réelle influence sur le pH du sol.
Figure 18 - Moyenne des pH par traitement. Les lettres minuscules correspondent aux groupes homogènes.
7.2 Mesure d’humidité
Les taux d’humidité sont compris entre 18,69 % et 22,33 % pour le sol de l’horizon 0-10 cm et entre 15,94 % et 18,57 % pour les sols de l’horizon 10-y cm (Figure 19). Globalement, il y a très peu de variations significatives du taux d’humidité du sol en fonction des traitements et ce, pour les sols des deux horizons. Cependant, le taux d’humidité du sol nu apparaît être légèrement plus faible que les traitements sous couvert végétal. Cela pourrait s’expliquer par le fait que la plante, via son système racinaire, retiendrait d’avantage l’eau pendant son infiltration ce qui conduirait à un plus fort taux d’humidité.
0 1 2 3 4 5 6 7 8 SN T1 T5 T6 T7 T8 T10 pH Traitements 0‐10 cm 10‐y cm ab ab ab ab b ab a ab ab b abab ab ab
Figure 19 - Moyenne des taux d’humidité en %. Les lettres minuscules correspondent aux groupes homogènes.
Les résultats montrent néanmoins, que l’humidité du sol en surface est significativement plus élevée que l’humidité du sol plus en profondeur. Il faudra vérifier par la suite que ce résultat n’influence pas la biomasse microbienne plus que les traitements en eux-mêmes.
7.3 Quantification des ADNs
Figure 20 – Exemple d’un gel d’électrophorèse obtenu après migration des ADN.
0 5 10 15 20 25 30 SN T1 T5 T6 T7 T8 T10 Tau x d 'h u m id it é (% ) ‐ Ho ri zo n 0 ‐10 cm 0 5 10 15 20 25 30 SN T1 T5 T6 T7 T8 T10 Tau x d'hum id it é (% ) ‐ Ho ri zo n 0 ‐y cm a a ab bc bc c bc a b c ab ab ab ab
Echantillons d’ADN à analyser Gamme
d’ADN de thymus
Marqueur de taille
Le gel ci-dessus présenté en figure 20, montre que de l’ADN a été correctement extrait et que celui-ci semble peu dégradé. La biomasse moléculaire est comprise entre 13,73 et 46,48 µg d’ADN/g de sol sec pour les sols de l’horizon 0-10 cm et entre 21,39 et 31,28 µg d’ADN/g de sol sec pour ceux de l’horizon 10-y cm (Figure 21).
Figure 21 – Moyenne de la biomasse moléculaire en μg d’ADN/g de sol sec. Les lettres minuscules représentent les groupes homogènes.
D’une manière générale, en ce qui concerne le sol nu et les traitements labour (T1, T5 et T6), la biomasse microbienne en surface semble être plus faible qu’en profondeur. Ce schéma s’inverse pour les traitements semis-direct (T7, T8 et T10), pour lesquels la biomasse moléculaire semble être plus élevée en surface. Plusieurs remarques peuvent être effectuées. D’une part, le fait que le sol nu et le labour aient le même effet sur la répartition de la biomasse moléculaire n’est pas surprenant sachant que le sol nu est maintenu ainsi par travail mécanique du sol. D’autre part, l’inversement de situation entre labour et semis direct montre que le travail du sol a un effet important sur la répartition de la biomasse moléculaire dans le sol et ce, même si peu de différences significatives ne sont mises en évidence par les analyses statistiques.
De plus, ces résultats montrent que l’effet des CIPAN est dépendant du travail du sol. En effet, il ne semble avoir un effet bénéfique sur la quantité de biomasse que ce soit pour l’horizon 0-10 cm et 0-10-y cm uniquement pour les traitements en semis-direct. Le travail du sol par le labour pourrait donc annuler les effets induits par les cultures intermédiaires.
Malgré les variations du taux d’humidité du sol, des modifications sont visibles. Il semble donc que la biomasse microbienne soit influencée de manière minime par ce paramètre. Nous avons
0 10 20 30 40 50 60 SN T1 T5 T6 T7 T8 T10 μg d' A D N br ut / g de s ol s ec Traitements 0‐10 cm 10‐y cm ab bc bc c bc bc a ab ab bc ab ac ab ac
vu que les variations observées n’étaient pas nettement significatives. Cependant, il est important de signaler que les prélèvements ont été réalisés sur site et que contrairement à un essai en laboratoire moins proche de la réalité, il y a de nombreux paramètres extérieurs plus ou moins contrôlés (conditions météorologiques, apports d’engrais, enrichissement en azote…).
8. Conclusion et perspectives
Les résultats ont montré qu’il n’y avait pas de différence significative de pH entre les deux horizons et entre les différents traitements. Les mesures d’humidité du sol en surface ont révélé qu’elles étaient significativement plus élevées que l’humidité du sol en profondeur. De plus, la quantification de la biomasse microbienne a indiqué que cette dernière semble être plus faible en surface qu’en profondeur pour le sol nu et les traitements labour (T1, T5 et T6). Cependant, elle semble plus élevée en surface pour les traitements semis-direct (T7, T8 et T10). Nous avons ainsi pu constater que le travail du sol a un effet important sur la répartition de la biomasse moléculaire et ce, même si peu de différences significatives sont mises en évidence par les analyses statistiques. Les mesures sur les parcelles avec CIPAN ont montré un effet travail du sol. Ainsi le labour pourrait réduire les différences de biomasse microbienne.
Le stage au sein de l’INRA se terminant le 3 août, plusieurs manipulations et analyses complémentaires sont prévues afin de répondre à l’objectif de départ, c’est-à-dire la caractérisation de l’abondance et de la diversité des communautés microbiennes des sols agricoles. Pour ce faire, nous avons besoin de mesures permettant d’identifier et d’analyser les communautés bactériennes et fongiques.
L’ADN brut doit en premier lieu être purifié. La purification se fait par deux méthodes successives et différentes. La première étape est un tamis moléculaire réalisé sur colonne de PVPP (Poly Vinyl Poly Pyrrolidone). La seconde étape est une colonne d’exclusion fournie dans un kit spécifique. Les éléments polluants tels que les protéines, sucres, macromolécules phénoliques, acides humiques seront ainsi éliminés. Par la suite, l’abondance des bactéries et des champignons microscopiques sera estimée par la quantification des séquences taxonomiques de ces organismes sur l’ADN de sol par l’intermédiaire de la PCR quantitative. L’objectif de ce mode opératoire est de dénombrer le nombre de copies d’ADN ribosomal (16S pour les bactéries et 18S pour les champignons) au sein d’un échantillon d’ADN. La structure génétique des communautés bactériennes et fongiques sera ensuite caractérisée par une technique de génotypage ARISA
(Automated Ribosomal Intergenic Spacer Analysis) directement à partir de l’ADN purifié du sol. Cette technique exploite le polymorphisme de taille de l’IGS (InterGenic Spacer) entre les gènes codant pour l’ARNr 16S et 23S pour les bactéries et l’ARNr 18S et 28S pour les champignons.Elle permet d’établir des similitudes ou différences de la structure des communautés des différentes modalités étudiées.
Ce stage m’a permis d’entrevoir le monde de la recherche et le travail en laboratoire que je ne connaissais pas. Mes connaissances générales ont été fortement enrichies tant sur le plan théorique que pratique car j’ai pu pratiquer plusieurs techniques de biologie moléculaire. L’appréhension de l’agroécologie par le biais de l’écologie microbienne m’a beaucoup intéressé. Cette expérience m’a permis de mieux préciser mon projet professionnel et a confirmé de poursuivre dans le domaine de l’écologie microbienne.
BIBLIOGRAPHIE
• Cortet, C., Gomot-De Vauflery, A., Poinsot-Balaguera, N., Gomot, L., Texier, C. et Cluzeau, D. (1999). The use of invertebrate soil fauna in monitoring pollutant effects.
European Journal of Soil Biology, 35 : 115-134
• Curtis, T.P., Sloan, W.T. et Scannell, J.W. (2002). Estimating prokaryotic diversity and its limits. Proceedings of the National Academy of Sciences of the USA, 99 : 10494-10499
• Dequiedt, S., Thioulouse J., Jolivet, C., Saby, N.P.A., Lelievre, M., Maron, P.A., Martin, M.P., Chemidlin-Prévost-Bouré, N., Toutain, B., Arrouays, D., Lemanceau, P. et Ranjard, L. (2009). Biogeographical patterns of soil bacterial communities. Environmental
Microbiology Reports, 1 : 251-255
• Dequiedt, S., Saby, N.P.A., Lelievre, M., Jolivet, C., Thioulouse, J., Toutain, B., Arrouays, D., Bispo, A., Lemanceau, P. et Ranjard, L. (2011). Biogeographical patterns of soil
molecular microbial biomass as influenced by soil characteristics and management. Global
Ecology and Biogeography, 20 : 641-52
• Duchaufour, P. (2001). Introduction à la science du sol. Sol, Végétation, Environnement, 6ème édition, Dunod, Paris, France
• Högberg, P., Nordgren, A., Buchmann, N., Taylor, A.F.S., Ekblad, A., Hogberb, M.N., Nyberg, G., Ottosson-Löfvenius, M. et Read, D.J. (2001). Large-scale forest girdling shows that current photosynthesis drives soil respiration. Nature, 411 : 789-792
• Horner-Devine, M.C., Leibold, M.A., Smith, V.H et Bohannan, B.J.M. (2003). Bacterial diversity patterns along a gradient of primary productivity. Ecology Letters, 6 : 613-622
• Kowalchuck, G.A. et Stephen, J.R. (2001). Ammonia-oxidizing bacteria : a model for molecular microbial ecology. Annual Review of Microbiology, 55 : 485-529
• Leake, J.R., Johnson, D., Donnelly, D.P., Muckle, G.E., Boody, L. et Read, D.J. (2004). Networks of power and influence : the role of mycorrhizal mycelium in controlling plant communities and agroecosystem functioning. Canadian Journal of Botany, 82 : 1016-1045
• Loreau, M. (2000). Biodiversity and ecosystem functioning: recent theorical advances.
Oikos, 91 : 3-17
• Ranjard, L., Dequiedt, S., Jolivet, C., Saby, N.P.A., Thioulouse, J.. Harmand, J., Loisel, P., Rapaport, A. , Fall, S., Simonet, P., Joffre, R., Chemidlin-Prevost-Bouré., N., Maron, P.A., Mougel, C., Martin, M.P., Toutain, B., Arrouays, D. et Lemanceau, P. (2010). Biogeography of soil microbial communities : a review and a description of the ongoing French national initiative. Agronomy for Sustainable Development, 30 : 359-365
• Rillig, M.C. et Mummey, D.L. (2006). Mycorrhizas and soil structure. New Phytologist, 171 : 41-53
• Smith, S.E. et Read, D.J. (2008). Mycorrhizal symbiosis. 3ème edition, Academic Press, New-York, Etats-Unis
• Sprent, J.I. (2001). Nodulation in Legumes. Royal Botanic Gardens, Kew, Royaume-Uni
• Torsvik, V. et Øvreås., L. (2002). Microbial diversity and function in soil: from genes to ecosystems. Current Opinion in Microbiology, 5 : 240-245
ANNEXES
Annexe 1 - Articulation de l’activité de la plateforme GenoSol.
Annexe 2 - Organigramme de la plateforme GenoSol.
Résumé
Actuellement, la qualité des sols est caractérisée principalement par des données physiques et chimiques.
Cependant, les microorganismes représentent la majorité des organismes vivants du sol. Ils ont un rôle très important dans son fonctionnement et notamment dans les cycles biogéochimiques. Les communautés microbiennes apparaissent comme des bio-indicateurs précoces de l’évolution des sols. Peu d’études génériques ont été réalisées pour développer ces bio-indicateurs permettant de diagnostiquer l’état biologique du sol et directement opérables par les utilisateurs de ces sols.
L’objectif du stage est d’étudier la distribution des communautés microbiennes dans le sol et d’évaluer l’impact de différents régimes du travail du sol sur ces dernières.
Des prélèvements ont été réalisés sur un essai du site d’ARVALIS situé à Boigneville (91), selon un protocole d’échantillonnage stratifié. Ils ont été effectués sur 2 horizons différents : 0-10 et 10-y cm (y étant la profondeur de l’ancien labour). Deux modalités de travail du sol différentes sont ainsi suivies : le labour et le semi-direct. En parallèle des prélèvements de sol nu ont été effectués pour être utilisés en tant que témoins.
Après avoir été tamisés, lyophilisés et conservés à -40°C, des mesures de pH et d’humidité ont été effectuées sur ces échantillons.
Par la suite, une estimation de l’abondance microbienne a été réalisée sur les différents horizons par des analyses de biologie moléculaire. Pour ce faire, une extraction de l’ADN génomique total à partir de 2 g de sol sec et une électrophorèse ont abouti à la quantification de la biomasse moléculaire en microgrammes par gramme de sol.
Les résultats ont montré que la biomasse moléculaire est sous l’influence de l’horizon de prélèvement et du travail du sol.
Mots clés
Sol, Communautés microbiennes, Travail du sol, Biomasse moléculaire, Horizons, Stratégie d’échantillonnage