© Benoit Bérubé, 2019
Impact à long terme de la conservation des résidus de
culture et des effluents d'élevage sur les communautés
bactériennes et fongiques du sol selon une approche
métagénomique
Mémoire
Benoit Bérubé
Maîtrise en biologie végétale - avec mémoire
Maître ès sciences (M. Sc.)
Impact à long terme de la conservation des résidus
de culture et des effluents d’élevage sur les
communautés bactériennes et fongiques du sol
selon une approche métagénomique
Mémoire
Benoit Bérubé
Sous la direction de :
Anne Vanasse, directrice de recherche
Chantal Hamel, codirectrice de recherche
iii
Résumé
Le microbiome du sol contribue à plusieurs rôles écologiques favorables à une agriculture durable. L’étude métagénomique des communautés du sol permet une nouvelle compréhension de l’impact des pratiques culturales sur l’écologie microbienne du sol. L’objectif du projet était de vérifier les impacts du travail de sol (labour ou travail réduit), de la fertilisation (fumier de poulet, lisier de bovin laitier, lisier de porc, engrais minéraux NPK ou PK) et de la gestion des résidus de culture (retournés ou exportés) après sept et huit années sur les communautés bactériennes et fongiques dans deux sols contrastés (loam sableux [LS] et argile limoneuse [AL]). La conservation des résidus a augmenté les diversités bactériennes dans le LS chaque année et fongiques dans chaque type de sol et année. Cette pratique a influencé les Proteobacteria, Acidobacteria, Actinobacteria,
Planctomycetes, Verrucomicrobia, Chloroflexi, Bacteroidetes, Gemmatimonadetes, Cyanobacteria et Nitrospirae, davantage en 2016 qu’en 2015 et différemment selon le
type de sol. L’application de lisier de porc a diminué la diversité fongique de chaque type de sol et année tout en favorisant l’abondance relative des Pyronemataceae, en comparaison à tous les autres traitements de fertilisation. Ces résultats ont été corrélés à des teneurs élevées en Cu des sols. La diversité des champignons mycorhiziens arbusculaires n’a pas été clairement affectée par les pratiques culturales. Les Glomus,
Paraglomus et Claroideoglomus, les trois genres mycorhiziens les plus abondants, ont
été affectés par le travail du sol dans le LS, par la fertilisation dans l’AL alors que la gestion des résidus a eu des effets dans l’AL en 2015 et dans le LS en 2016. En conclusion, les communautés bactérienne et fongique ont été influencées par les résidus pour chaque type de sol et année. La fertilisation, l’application de lisier de porc corrélé aux teneurs de Cu du sol, a influencé les communautés fongiques.
v
Abstract
Soil microbiome is involved in many ecological services contributing to sustainable agriculture. Metagenomic techniques now allow a whole new level of comprehension of soil microbial communities. The aim of our research project was to define the impacts of tillage (plowing or reduced tillage), fertilization (poultry manure, dairy cattle slurry, pig slurry, NPK and PK mineral fertilizers) and residues management (returned or exported) on bacterial and fungal soil communities on two soils with contrasting texture (sandy loam [LS] and silty clay [AL]) after seven and eight years. Residues conservation increased bacterial diversity in the sandy loam each year and fungal diversity in each soil type and year. Residues conservation also impacted Proteobacteria, Acidobacteria,
Actinobacteria, Planctomycetes, Verrucomicrobia, Chloroflexi, Bacteroidetes, Gemmatimonadetes, Cyanobacteria and Nitrospirae, more in 2016 than 2015, and
differently according to soil types. Pig slurry application reduced fungal diversity. This type of fertilizer was furthermore linked to higher relative abundance of Pyronemataceae compared to other fertilizers for each soil type and year. These effects were correlated to high copper concentration in soil. Arbuscular mycorrhizal fungi diversity was not clearly impacted by the treatments. Glomus, Paraglomus and Claroideoglomus, the three more abundant mycorrhizal genera, were affected by soil tillage in LS each year, by fertilization in AL each year and by residues management in AL for 2015 and in LS for 2016. In
conclusion, bacterial and fungal communities were influenced the most by residues conservation. Fertilization via pig slurry applications was correlated with high soil copper concentration and impacted the most fungal community.
vii
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des tableaux ... ix
Liste des figures ... xi
Remerciements ... xiii
Avant-propos ... xv
Introduction générale ... 1
0 Revue de littérature ... 3
0.1 Le sol, une matrice bien vivante ... 3
0.1.1 Dynamique des bactéries ... 3
0.1.2 Dynamique des champignons ... 5
0.1.3 Dynamique des champignons mycorhiziens arbusculaires ... 6
0.1.4 Variabilité temporelle du microbiome ... 7
0.1.5 Variabilité spatiale du microbiome... 8
0.2 Étude du microbiome du sol ... 9
0.2.1 Techniques d’identification microbiologique ... 9
0.2.2 Techniques d’analyse écologique ... 11
0.3 Facteurs influençant le microbiome du sol ... 12
0.3.1 Caractéristiques édaphiques ... 12
0.3.2 Impacts des pratiques culturales ... 15
1 Objectifs et hypothèses ... 31
2 Impacts à long terme de la conservation des résidus et des effluents d’élevage sur les communautés bactériennes et fongiques selon une approche métagénomique ... 32
Résumé ... 32
Long term impact of residues conservation and farmyard manures application on bacterial and fungal soil communities using a metagenomic approach ... 35
Abstract ... 35
Introduction ... 37
Matériel et méthodes ... 40
viii
Échantillonnage de sol ... 42
Extraction et amplification ... 42
Traitement bioinformatique ... 44
Identification des OTUs... 44
Analyses statistiques ... 45 Résultats ... 46 Communautés bactériennes ... 46 Communautés fongiques ... 48 Communautés mycorhiziennes ... 50 Discussion ... 52
Impact des traitements sur les communautés bactériennes ... 52
Impacts des traitements sur les communautés fongiques ... 57
Impact des traitements sur les communautés mycorhiziennes ... 59
Conclusion ... 62 Remerciements ... 63 Références ... 64 Tableaux et figures ... 69 3 Discussion générale ... 83 Communautés bactériennes ... 83 Communautés fongiques ... 85 Communautés mycorhiziennes ... 87 Conclusion générale ... 89 Bibliographie ... 91 Annexes complémentaires ... 105
ix
Liste des tableaux
Tableau 1. Rapport C/N et teneurs moyennes en N des effluents d’élevage à l’étude.. 19
Tableau 2. Caractérisation des effluents d’élevage appliqués sur le loam sableux (LS) et
l’argile limoneuse (AL) pour la phase blé en 2016. ... 69
Tableau 3. Effets significatifs des traitements sur l’indice de diversité de Shannon
(ANOVA) et la structure des communautés (perMANOVA) bactérienne, fongique et de champignons mycorhiziens arbusculaires (CMA) dans le loam sableux (LS) et l’argile limoneuse (AL) des phases soya en 2015 et blé en 2016. ... 70
Tableau 4. Effets significatifs des traitements sur la structure des communautés des
champignons mycorhiziens arbusculaires (CMA) associées aux phases soya en 2015 et blé en 2016, dans le loam sableux (LS) et l’argile limoneuse (AL) selon une perMANOVA menée sur les profils de genres. ... 71
Tableau 5. Moyennes (± erreur-type) des teneurs en éléments minéraux (mg/kg) du loam
sableux (LS) et l’argile limoneuse (AL), tels qu’extraits par Mehlich-3 et en fonction des traitements, dans les phases soya en 2015 et blé en 2016. ... 105
xi
Liste des figures
Figure 1. Impacts de la gestion des résidus de culture, retournés (R) versus exportés (E),
sur les indices de diversité de Shannon bactériens (A) et fongiques (B) dans le loam sableux (LS) et l’argile limoneuse (AL) des phases soya en 2015 et blé en 2016. Des lettres différentes entre les barres (n = 30) témoignent d’effets significatifs selon un test de Tukey à un seuil alpha de 5 %. Les barres d’erreur représentent l’erreur-type. ... 72
Figure 2. Impact de la fertilisation, soit avec du fumier de poulet (FP), du lisier de bovin
laitier (LB), du lisier de porc (LP), de l’engrais minéral complet (NPK) ou de l’engrais minéral sans azote (PK), seule ou en interactions, sur les indices moyens de diversité de Shannon bactériens dans le loam sableux (A) et l’argile limoneuse (B) des phases soya en 2015 et blé en 2016. La fertilisation et le travail de sol (Travail), soit un labour (L) ou un travail réduit (Tr), sont en interaction. Des lettres différentes entre les barres (seule, n = 12; interactions, n = 6) témoignent d’effets significatifs selon un test de Tukey à un seuil alpha de 5 %. Les barres d’erreur représentent l’erreur-type. ... 73
Figure 3. Abondances relatives des principaux phyla bactériens (A), familles fongiques
(B) et genres mycorhiziens (C) composant les communautés du loam sableux (LS) et de l’argile limoneuse (AL) des phases soya en 2015 et blé en 2016. La catégorie « Indéterminés » regroupe les OTUs qui n’ont pas pu être identifiées au niveau taxonomique spécifié. ... 74
Figure 4. Effets significatifs des traitements, soit le travail de sol (T), la fertilisation (F)
et la gestion des résidus de culture (G), sur l’abondance relative des taxons dominants et sur la structure (calculée par perMANOVA) des communautés, par phyla bactériens (A) et familles fongiques (B) dans le loam sableux (LS) et l’argile limoneuse (AL) des phases soya en 2015 et blé en 2016... 75
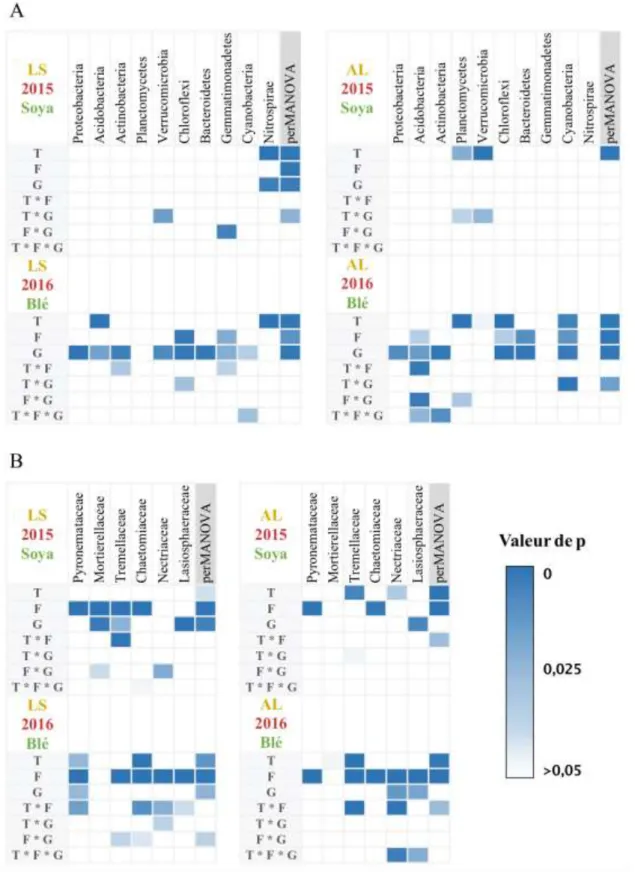
Figure 5. Impacts de la gestion des résidus de culture, retournés (R) versus exportés (E),
sur les abondances relatives des principaux phyla bactériens (n = 30 pour chaque barre) composant les communautés du loam sableux [LS] (A) et de l’argile limoneuse [AL] (B) des phases soya en 2015 et blé en 2016. La catégorie « Indéterminés » regroupe les OTUs qui n’ont pas pu être identifiées au niveau taxonomique spécifié. ... 76
Figure 6. Impact de la fertilisation, soit du fumier de poulet (FP), du lisier de bovin laitier
(LB), du lisier de porc (LP), de l’engrais minéral complet (NPK) ou de l’engrais minéral sans azote (PK), seule ou en interactions, sur les indices moyens de diversité de Shannon fongiques dans le loam sableux (A) et l’argile limoneuse (B) des phases soya en 2015 et blé en 2016. La fertilisation et le travail de sol (Travail), soit un labour (L) ou un travail réduit (Tr), ainsi que la fertilisation et la gestion des résidus (Résidus), retournés (R) ou exportés (E) des parcelles, sont en interaction. Des lettres différentes entre les barres
xii
(seule, n = 12; interactions, n = 6) témoignent d’effets significatifs selon un test de Tukey à un seuil alpha de 5 %. Les barres d’erreur représentent l’erreur-type. ... 77
Figure 7. Impact de la fertilisation, soit du fumier de poulet (FP), du lisier de bovin laitier
(LB), du lisier de porc (LP), de l’engrais minéral complet (NPK) ou de l’engrais minéral sans azote (PK) sur les abondances relatives de la famille fongique des Pyronemataceae dans le loam sableux (A) et l’argile limoneuse (B) des phases soya en 2015 et blé en 2016. Des lettres différentes entre les barres (n = 12) témoignent d’effets significatifs selon un test de Tukey à un seuil alpha de 5 %. Les barres d’erreur représentent l’erreur-type. . 78
Figure 8. Graphique de régression, présentant les tendances des corrélations de Pearson,
entre les indices de diversité de Shannon des champignons en fonction des teneurs en cuivre du sol des parcelles (mg/kg) pour le loam sableux (LS) et l’argile limoneuse (AL) des phases soya en 2015 et blé en 2016. Les lignes représentent les équations de régression et les zones ombragées les erreurs-types associées pour chaque combinaison sol et année (n = 60) ... 79
Figure 9. Graphique de régression, présentant les tendances des corrélations de Pearson,
entre les abondances relatives des Pyronemataceae en fonction des teneurs en cuivre du sol des parcelles (mg/kg) dans le loam sableux (LS) et l’argile limoneuse (AL) des phases soya en 2015 et blé en 2016. Les lignes représentent les équations de régression et les zones ombragées les erreurs-types associées pour chaque combinaison sol et année (n = 60). ... 80
Figure 10. Analyses en composantes principales (PCA) illustrant les effets significatifs
des traitements, soit du travail de sol (T), de la fertilisation (F) et de la gestion des résidus de culture (G), sur les genres mycorhiziens (CMA) pour le loam sableux (LS) et l’argile limoneuse (AL) des phases soya en 2015 et blé en 2016. Impacts des traitements dans le LS (A – B – C) et dans l’AL (D – E – F). Le T est un labour (L) ou un travail réduit (Tr). La F comprend l’application de fumier de poulet (FP), lisier de bovins laitiers (LB), lisier de porc (LP), d’engrais minéral complet (NPK) et d’engrais minéral sans azote (PK). La G consiste en la conservation des résidus de culture dans la parcelle (R) ou leurs exportations (E). Les huit genres mycorhiziens sont : Glomus (Glom.), Paraglomus (Para.), Claroideoglomus (Clar.), Diversispora (Dive.), Archaespora (Arch.),
Acaulospora (Acau.), Scutellospora (Scut.) et Ambispora (Ambi.). Les points noirs
représentant les CMA indiquent la présence d’un seul genre, tandis que les points blancs indiquent plusieurs CMA confondues en un seul point. Les noms des CMA représentés par ces points blancs, pour les PCA D – E – F, sont écrits en diagonale inférieure gauche du point blanc sur chacune des sous-figures. Chaque point représentant les traitements est une moyenne des répétitions des 20 combinaisons de traitements possibles comprenant les trois facteurs à l’étude. ... 81
xiii
Remerciements
Je tiens d’abord à remercier ma directrice Anne Vanasse. Sa passion de l’agronomie, son dévouement envers ses élèves et son sens de la pédagogie ont été pour moi des éléments très inspirants pour en venir à continuer aux études graduées. J’ai eu la chance de bénéficier aussi bien de la grande expérience terrain d’Anne que de ses commentaires justes pour affiner l’analyse de mes résultats et la qualité de ma rédaction. Merci de m’avoir supporté dans ce projet ambitieux, d’avoir tenté l’expérience métagénomique à mes côtés et pour tes retours rigoureusement rapides malgré tes horaires bien chargés.
Un grand merci à Chantal Hamel pour sa codirection. Tu m’as assurément bien guidé pour en venir à apprivoiser les nombreux concepts écologiques et subtilités de la microbiologie des sols auxquels j’étais totalement étranger au départ. Merci pour ta grande disponibilité, ton ouverture face à toutes mes ambitions et mes nombreuses interrogations. Ta franchise indéfectible et ta rigueur bien critique sont pour moi des qualités qui m’ont permises de me dépasser.
Un merci particulier à Martin Chantigny pour sa contribution depuis des années à la coordination et à la gestion du site expérimental. Chaque échange et discussion en sa compagnie à propos de l’expérience ont été enrichissants.
Merci à mon équipe de recherche rapprochée d’Agriculture et agroalimentaire Canada, notamment pour nos nombreux échanges permettant de démystifier les subtilités innombrables de la bioinformatique. Réjean Desgagnés, nos escapades sur le terrain et ton excellent sens de l’humour ont toujours été appréciés. David Gagné, merci de m’avoir toujours accueilli avec bonne humeur à ton bureau, malgré les bons défis techniques pour lesquels je te sollicitais. Mario Laterrière, merci de m’avoir permis de rester en bons termes aussi bien avec mes analyses que mes statistiques grâce à ton aide et tes questionnements bienveillants. Sans votre support, je serais certainement encore à coder sous Linux, USearch et R.
Merci à Johanne Tremblay, Gabriel Lévesque et Annie Robichaud pour votre contribution à mon projet, votre temps pour m’avoir aidé à démystifier l’historique de l’expérience et votre soutien précieux lorsque je venais visiter vos laboratoires. Merci à Isabelle Royer
xiv
et Catherine Pinsonneault pour votre accueil chaleureux au laboratoire. J’ai apprécié tout mon temps passé en votre compagnie, les discussions passionnantes sur la caractérisation des sols (oui, oui), le monde de la recherche et la vie en général en fait. Merci à Marie-Ève Giroux pour son support dans mes premiers temps en recherche, aussi bien lorsque j’étais auxiliaire de recherche qu’étudiant gradué. Merci à Francis Gagnon et toute l’équipe de la ferme expérimentale de Saint-Augustin-de-Desmaures pour leur travail remarquable, toujours disponible malgré les imprévus et intempéries. Un merci non négligeable à tous les autres employés ainsi qu’étudiants d’été d’Agriculture et agroalimentaire Canada et de l’Université Laval qui ont participé de près et de loin à mon projet.
Non les moindres, un gros merci à mes amis et collègues étudiants gradués qui m’ont accompagné dans toutes les péripéties de ma maîtrise. Cindy Denoncourt, notre duo de site expérimental a certainement été le meilleur, depuis le baccalauréat, en tant qu’acolytes de travaux aux champs, partenaires de danse, de Normandin et d’aventures à travers le Québec! Hugo Alix, tu as été tout un partenaire de bureau formidable, un bourreau de travail et de bon temps. Stéphanie Patenaude, Laura Thériault, Catherine Couture, Andréeanne Belley, Jean-Pascal Matteau, Andréanne Lavoie, vous savez que j’ai toujours apprécié nos moments de travail, de discussion et de détente ensemble. Virginie Caron, Véronique Villeneuve, Naomie Bouchard-Laroche, Marie-Ange Therrien et Mélanie Dionne, merci de rester des amies proches malgré la distance. Amélie Gervais et Olivier Samson-Robert, votre présence a été inestimable dans les derniers miles. Les Bonenfants, merci de m’avoir permis de conjuguer un stage fantastique avec la fin de ma maîtrise à temps partiel.
Enfin, merci à ma famille pour leur support inconditionnel et leur compréhension. Merci d’être des modèles d’humanité, d’entrepreneuriat, de résilience et de fougue. Merci d’être toujours présent même à l’autre bout du monde. Merci à mes nièces, Jeanne et Charlotte, et mon filleul, Léon, de me rappeler les bonnes priorités malgré les échéanciers.
xv
Avant-propos
Ce mémoire comprend six chapitres. Le premier est une introduction générale qui permet de contextualiser le cadre de recherche. Le second chapitre constitue une revue de littérature sur le microbiome du sol, les techniques de séquençage, bioinformatiques et statistiques, et les impacts actuellement connus des pratiques culturales sur celui-ci. Le troisième chapitre énonce les objectifs et hypothèses de la recherche. Le quatrième chapitre est présenté sous forme d’article scientifique. Il présente les résultats obtenus après sept et huit ans de traitement sur deux sols aux textures contrastées quant à l’impact du travail réduit de sol, de la fertilisation à base d’effluents d’élevage et de la conservation des résidus de culture sur les communautés bactériennes, fongiques et des champignons mycorhiziens arbusculaires. Ce travail est en préparation et ne sera pas soumis pour publication sous cette forme, mais intégré avec d’autres résultats de recherche comprenant plus d’années d’analyse et séparé par groupe taxonomique. Benoit Bérubé est l’auteur principal, ayant assuré la rédaction et participé à toutes les étapes de l’étude; Chantal Hamel et Anne Vanasse en sont les coauteures, ayant contribué à la rédaction de l’article, tout comme à la codirection du projet de recherche; enfin, Martin Chantigny en est coauteur, ayant été responsable de la gestion du projet de recherche et de réviser l’article. Le cinquième chapitre effectue un retour sur les objectifs de l’étude en regard des résultats obtenus, soulignant les implications de la recherche dans son contexte agricole. Le sixième et dernier chapitre rappelle les conclusions majeures de l’étude.
Les résultats de l’étude ont été présentés jusqu’à ce jour aux occasions suivantes : en 2017, au 31e congrès annuel de l’Association québécoise des spécialistes en science du sol (AQSSS); au Colloque Mycorhize 2017 du Centre d’étude de la forêt (CEF) de l’Université Laval; en 2018, au déjeuner-conférence de l’Ordre des agronomes du Québec (OAQ) – Section Capitale nationale.
Le projet s’est concrétisé grâce au soutien financier d’Agriculture agroalimentaire Canada ainsi que de l’Université Laval au courant des années pour le maintien des parcelles à long terme.
1
Introduction générale
L’importance d’avoir des sols en santé, notamment actifs biologiquement, est une préoccupation du XXIe siècle. Cette dernière succède aux excès découlant de la modernisation rapide de l’agriculture durant la deuxième moitié du XXe siècle, dite « Révolution verte ». L’utilisation plus massive d’intrants de synthèse, l’augmentation drastique des rendements des cultures et la mécanisation à large échelle des travaux agricoles ne sont que quelques exemples des changements opérés à cette époque. Cela a littéralement transformé l’histoire de l’humanité, d’une agriculture de subsistance à la production de masse par peu d’individus (FAO et ITPS, 2015). Néanmoins, le recul actuel permet de déterminer certaines limites de cette révolution, notamment en regard de la pression exercée sur les sols cultivés (Evenson et Gollin, 2003). L’acidification, la compaction, l’érosion et la diminution de l’activité biologique des sols ne sont que quelques conséquences de cette pression sur les terres agricoles. Cette pression est maintenant reconnue comme un problème de taille à maîtriser pour assurer la sécurité alimentaire mondiale (Turbé et al., 2010b). Le développement durable désormais recherché passe par le respect des ressources-sols limitées ainsi que fragiles à l’échelle humaine. Des pratiques culturales plus responsables et respectueuses de l’environnement sont alors nécessaires (Doran, 2002). Pour en arriver à cette agriculture priorisant la conservation des sols, il importe de bien comprendre le fonctionnement du microbiome impliqué. Sa composition jusqu’aux interactions complexes avec son milieu, en passant par les fonctions des communautés microbiennes, sont des éléments pouvant favoriser la croissance des plantes (Doran et Zeiss, 2000).
Ce mémoire représente une pierre à l’édifice des connaissances de la santé des sols en grandes cultures. Il se concentre sur les impacts du travail de sol, de la fertilisation à base d’effluents d’élevage et de la conservation des résidus de culture sur les communautés bactériennes et fongiques du sol. Une attention particulière est aussi portée au phylum des champignons mycorhiziens à arbuscules (CMA) puisqu’ils constituent des alliés naturels de taille pour favoriser des pratiques culturales durables dans de nombreuses productions végétales.
3
0 Revue de littérature
0.1 Le sol, une matrice bien vivante
Dans un seul gramme de sol, plus d’un milliard de microorganismes peuvent être présents (Voroney et Heck, 2015), mais seulement entre un et dix pourcents de ces espèces sont connues et décrites selon les connaissances actuelles (Thies, 2015). Cela s’explique partiellement par l’impossibilité d’en cultiver une majorité en laboratoire. La difficulté d’utiliser des méthodes non destructrices présente aussi un défi pour étudier les sols en conditions réelles (Thies, 2015). La vie dans les sols constitue un réseau trophique complexe qui inclue aussi bien des mammifères, vers de terre, arthropodes, nématodes que des protozoaires (Paul, 2015). De surcroît, les bactéries et les champignons colonisant les sols sont des groupes d’intérêts en agriculture puisqu’ils participent au développement de la structure ainsi que de la fertilité des sols (Morris et Blackwood, 2015; Voroney et Heck, 2015). Leurs activités métaboliques peuvent aussi modifier l’acidité des sols et ils sont responsables du relâchement d’enzymes extracellulaires modifiant les propriétés du sol (Plante et al., 2015). D’un point de vue phytosanitaire, autant les communautés bactériennes que fongiques peuvent induire chez les plantes des résistances aux stress abiotiques et biotiques ou constituer une menace pour les cultures en tant qu’agent pathogène (Balestrini et al., 2015).
0.1.1 Dynamique des bactéries
L’étude des bactéries en sol agricole se concentre souvent autour d’organismes connus ayant des effets bénéfiques ou pathogènes sur des cultures données. En ce sens, les symbioses entre les bactéries du genre Rhizobium et les légumineuses permettant de fixer l’azote atmosphérique sont bien étudiées (Long, 1996). Pareillement, les cycles de vie des bactéries responsables de nécroses sont mieux compris (Agrios, 2005b). Néanmoins, ces microorganismes cultivables en laboratoire et aux fonctions connues ne représentent pas la majorité du microbiome bactérien présent dans les sols. Comme dévoilé par les approches d’identification moléculaires, c’est une diversité de bactéries qui est difficile à étudier en conditions contrôlées et aux fonctions bien souvent méconnues qui compose ce microbiome (Cardenas et Tiedje, 2008; Kandeler, 2015).
4
L’étude du microbiome bactérien doit s’effectuer en tenant compte de certaines limites des connaissances actuelles. Historiquement, la phylogénie bactérienne est basée sur celle de plantes, c’est-à-dire un système de classification où les organismes sont sexués. Au contraire, les bactéries sont des organismes procaryotes, conséquemment asexués, ce qui ne rend pas aussi évident le concept de filiation et la linéarité de leur carte évolutionnelle (Corel et al., 2016; Killham et Prosser, 2015; Yarza et al., 2014). Les modifications génétiques chez les bactéries surviennent par mutation du génome, mais aussi par l’acquisition de gènes entres organismes. Une certaine variabilité génotypique, dont l’origine n’est pas forcément claire, existe à l’intérieur des individus d’une même espèce. Cela se traduit par des fonctions présentes chez plusieurs organismes non reliés directement par la phylogénie (Killham et Prosser, 2015; Klenk et Göker, 2010). Il est essentiel de reconnaître les capacités organisationnelles des bactéries. Bien qu’étant des organismes unicellulaires, les bactéries ne privilégient pas une vie sous forme libre, dite planctonique, dans l’environnement. Elles optent plus souvent pour la création de biofilms. Les biofilms sont constitués de communautés de bactéries, de la même espèce ou non. Ces espèces interagissent de façon symbiotique pour mieux survivre aux contraintes de leur environnement et se reproduire. Le biofilm se matérialise par une structure composée d’exsudats bactériens, principalement de polysaccharides. Il se fixe à un milieu solide, vivant ou non. Il fonctionne comme une entité unique et développe des fonctions propres à cette organisation selon les conditions environnementales (Flemming et Wingender, 2010; Prosser, 2011). En tenant compte des bases de la phylogénie bactérienne, de la fluidité génétique dont peuvent faire preuve les bactéries et de leur préférence à croître sous forme de biofilms, il est évident que l’étude du microbiome bactérien et des fonctions reliées à ce dernier doivent se baser sur des analyses liant les organismes aux caractéristiques du milieu d’origine. La prise en compte des interactions possibles des organismes avec le reste du microbiome présent est aussi à considérer (Killham et Prosser, 2015)
Les rôles écologiques sont influencés par la façon dont les bactéries acquièrent leur énergie et le carbone. Elles peuvent respectivement être phototrophe ou chimiotrophe, capter le carbone via du dioxyde de carbone ou des composés organiques. Les besoins en oxygène, les différentes formes de matières inorganiques et organiques utilisables par les bactéries modulent leurs actions. La compréhension de l’action catalytique de leurs enzymes permet également de mieux comprendre l’écologie microbienne des sols. La
5
cinétique de croissance d’un type de bactérie dépendra de sa compétitivité à croître plus efficacement comparativement aux autres organismes selon un substrat donné à une concentration donnée. Ces quelques facteurs discriminants mettent en évidence la grande variabilité physiologique présente dans la diversité bactérienne. Il faut garder à l’esprit l’adaptabilité dont peuvent faire preuve certains organismes : capacité d’utiliser de nouveaux substrats de croissance en cas de carence du substrat principal ou encore capacité à fonctionner sous métabolisme anaérobique facultatif par exemple (Killham et Prosser, 2015; Prescott et al., 2010a).
De cette diversité taxonomique et fonctionnelle découlent plusieurs rôles écologiques centraux pour la croissance des cultures en contexte agricole. La biomasse bactérienne est partie prenante des cycles du carbone, du phosphore et de l’azote, tous des éléments essentiels pour les plantes (Prescott et al., 2010b). Les procaryotes, archées incluses, constituent eux-mêmes un réservoir de C organique aussi important que la biomasse végétale. Les bactéries et archées constituent également un réservoir 10 fois plus important d’azote comparativement aux végétaux. L’activité métabolique et la quantité présente de ces microorganismes influencent conséquemment la fertilité du sol et la gestion de l’eau (Killham et Prosser, 2015). Les ressources utilisées pour le métabolisme des bactéries peuvent même parfois entrer en compétition avec les besoins nutritifs des plantes. Il sera alors question d’immobilisation des ressources par les microorganismes au détriment des besoins des plantes (Stevenson, 1986).
0.1.2 Dynamique des champignons
Les champignons représentent un règne d’organismes eucaryotes qui prolifèrent dans les sols en relation étroite avec la décomposition et la stabilisation de la matière organique (Lee et Sinsabaugh, 2015). Ils s’alimentent grâce à une vaste gamme d’enzymes. Cela leur permet de dégrader la matière organique directement dans l’environnement, à l’extérieur de l’organisme, puis de l’absorber par transport direct et osmose. Cette manière de fonctionner est partie prenante du recyclage des éléments nutritifs composant la matière organique et de la capacité de souches pathogènes fongiques à s’attaquer aux cultures (Caldwell, 2005). Les communautés fongiques ne présentent pas une aussi grande diversité de fonctions que les bactéries et sont essentiellement hétérotrophes (Prescott et al., 2010c). Leurs réseaux d’hyphes sont en mesure d’explorer de plus grandes
6
distances. Cela permet aux champignons d’êtres plus résilients à des conditions extrêmes comparativement aux bactéries: absence d’eau, discontinuité de la présence d’oxygène dans le profil de sol et température extrêmes. Par cette faculté de se propager plus largement et densément dans les sols, les champignons agissent littéralement comme un réseau de transport, assurant la distribution des ressources, parfois même entre certains organismes extérieurs (Lee et Sinsabaugh, 2015). Les hyphes fongiques sont communément une surface propice pour le développement de biofilms symbiotiques qui peuvent également inclure une association végétale tripartite (de Menezes et al., 2017).
L’étude de la diversité fongique comporte, tout comme celle des bactéries, ses subtilités. La classification taxonomique des champignons, de façon analogue à celle des bactéries, porte à confusion. Elle a été initialement basée sur le règne végétal et la description des caractères morphologiques. Cela a mené à un dédoublement des dénominations pour un organisme unique dans le cas des champignons, selon qu’il ait été étudié sous leur forme sexuée, téléomorphe, ou asexuée, anamorphe (Hibbett et Taylor, 2013). Les champignons sont en mesure de moduler leur reproduction selon les conditions du milieu. Notamment, la production de spores leur permet de survivre à des conditions adverses telles des variations extrêmes de température et la sécheresse (Prescott et al., 2010c).
Les champignons jouent un rôle complémentaire aux bactéries pour assurer la fertilité des sols et dominent dans des conditions différentes. En milieu plus acide, pauvre en nutriments, mais riche en litières avec des ratios carbone sur azote (C/N) élevés, une majorité de champignons croît plus facilement (Fierer et al., 2009). Au contraire, les bactéries se propagent globalement mieux au détriment des champignons dans un environnement riche en azote et phosphore facilement accessibles, en conditions salines ou plus alcalines et en absence d’oxygène (Joergensen et Wichern, 2008).
0.1.3 Dynamique des champignons mycorhiziens arbusculaires
Les champignons mycorhiziens à arbuscules (CMA), avec plus de 300 espèces validement décrites à ce jour, composent le phylum des Glomeromycota (amf-phylogeny.com, 2019). Ils sont en mesure d’établir des symbioses avec plus de 250 000 espèces végétales. Cela présente un intérêt en agriculture puisque les plantes mycorhiziennes incluent la majorité des espèces végétales cultivées commercialement
7
(Fortin et al., 2015d). La famille des crucifères, entre autres le canola et les choux, ou celle des chénopodiacées, dont les betteraves et les épinards, n’en font pas partie. Chaque CMA possède une spécificité d’association. Il peut s’associer à un nombre variable de plantes et être plus ou moins efficace dans les échanges symbiotiques selon les cultures impliquées dans la symbiose (Fortin et al., 2015c). Cette symbiose entre les CMA et les plantes permet à la culture d’accéder plus facilement aux nutriments (Neumann et George, 2010), spécialement ceux peu mobiles dans les sols comme le phosphore (Harrison et al., 2010), à l’eau (Ellouze et al., 2014), ainsi que d’être plus résistante aux stress abiotiques (Ruiz-Lozano et Aroca, 2010) et biotiques (Pozo et al., 2010). Les CMA reçoivent du carbone sous forme de sucres de la part des végétaux qui synthétisent ces derniers par photosynthèse (Balestrini et al., 2015). Les CMA peuvent agir comme des parasites en s’appropriant les sucres des plantes à leurs détriments en condition abondante d’N et de P accessibles directement par les végétaux (Antunes et al., 2012). Pour un mètrecarré de sol exploré par des racines, il est possible pour une plante d’en explorer jusqu’à 10 m2 en s’associant aux hyphes fongiques. Ces hyphes sont plus fins et réactifs aux échanges avec la solution du sol que les systèmes racinaires seuls. La plante peut bénéficier directement de ces avantages compétitifs qu’ont les CMA d’être en mesure d’explorer plus efficacement les sols (Fortin et al., 2015d). En contexte agricole, les Glomeraceae, incluant le genre Glomus, composent la majorité des CMA retrouvés (Borriello et al., 2012).
0.1.4 Variabilité temporelle du microbiome
Le microbiome des sols, aussi bien bactérien que fongique, varie à travers le temps et cela par rapport à deux échelles d’intérêt. Il change au courant d’une saison de culture, mais également en termes d’années consécutives (Tardy et al., 2015). Ces deux référentiels prennent en compte les fluctuations dues aux mêmes facteurs : température, humidité, oxygénation, interactions du microbiome avec les plantes selon l’espèce végétale et le stade physiologique, ainsi que les pratiques culturales effectuées (Donn et al., 2015; Morris et Blackwood, 2015). Bien que des variations saisonnières puissent avoir des impacts à chaque année sur la diversité du microbiome de façon temporaire, cela n’implique pas nécessairement un changement majeur dans la composition des communautés microbiologiques sur le long terme (Gonzalez et al., 2012). Les variations temporelles du microbiome à la suite de plusieurs années consécutives de perturbation
8
peuvent être cumulatives. Elles sont davantage d’intérêt dans une perspective de développement durable. Ainsi, les choix de pratiques culturales d’un producteur influencent la composition du microbiome du sol à long terme. Il est possible de favoriser la biodiversité des sols et même d’en venir à moduler les communautés composant le microbiome des sols agricoles. Ce concept est latent aux pratiques biologiques, écologiques et raisonnées en agriculture au Québec (Canada, 2015; CARTV, 2011). Hormis de savoir qu’une activité microbienne élevée favorise généralement la biodiversité et garantit le bon fonctionnement du sol, peu est connu sur les facteurs favorisant les communautés bénéfiques ou délétères pour les cultures (Turbé et al., 2010a).
0.1.5 Variabilité spatiale du microbiome
La présence de variabilité spatiale lors d’échantillonnages de sols est un phénomène bien connu, autant lorsqu’il est question d’évaluer les propriétés physiques ou chimiques des sols (Frey, 2015). Cette variabilité est également présente lorsqu’il est question d’évaluer la diversité microbienne du sol (Gonzalez et al., 2012). À l’intérieur d’un même champ, plusieurs points de mesures sont nécessaires pour assurer la représentativité de l’échantillon (CEAEQ, 2010). Il faut bien définir quelle profondeur du profil sera étudiée. L’activité biologique varie dans les sols selon la profondeur du profil. L’intensité de l’activité biologique des sols est plus élevée en surface, spécialement dans les quinze premiers centimètres de sol. Ceux-ci représentent la zone cultivable la plus fréquemment travaillée en agriculture et là où l’activité racinaire est la plus importante (Frey, 2015). La variabilité spatiale tient compte de microsites dans la structure des sols. C’est-à-dire qu’il est possible de retrouver des milieux avec des conditions très contrastantes les uns à côté des autres (Chenu et al., 2015; Kravchenko et al., 2014). Par exemple, certains microsites anaérobiques existent dans les premiers centimètres de sol, malgré la présence plus abondante d’oxygène environnante. Ces microsites permettent la prolifération d’espèces microbiennes aux premiers abords incompatibles avec les conditions générales du milieu.
La variabilité spatiale dans un champ peut être liée au développement racinaire. La rhizosphère, les quelques millimètres de sols entourant les racines, est un milieu riche d’échanges entre microorganismes et plantes qui est alimenté par les exsudats racinaires (George et al., 2011; Nagahashi et al., 2010). Ce microbiome de la rhizosphère est
9
différent de celui en plein sol où les conditions environnementales ne sont pas les mêmes (Donn et al., 2015). Les parties aériennes de la plante, la phyllosphère, constituent un autre environnement au microbiome distinct (Turner et al., 2013). De façon plus large, un organisme et l’ensemble des microorganismes le colonisant constituent une entité biologique unique, qualifiée d’holobionte. Le microbiome du sol, celui en plein sol, est la résultante des holobiontes qui l’ont influencé. Plus encore, les microorganismes composant ce microbiome peuvent eux-mêmes influencer par la suite les holobiontes qu’ils côtoient. Concrètement, le microbiome du sol à long terme a donc une influence directe sur la croissance des plantes (Vandenkoornhuyse et al., 2015).
0.2 Étude du microbiome du sol
0.2.1 Techniques d’identification microbiologique
L’étude de la microbiologie du sol a débuté avec l’observation des phénotypes des microorganismes. Ce processus a été intimement lié à l’identification et à la classification taxonomique des espèces (Killham et Prosser, 2015; Lee et Sinsabaugh, 2015). Dans ce processus d’identification, dès les années 1960, le concept d’unité taxonomique opérationnelle (OTU) a été proposé. Il regroupe les organismes de manière quantitative quant à la similarité de leurs traits. Ce concept flexible quant au niveau taxonomique qu’il est possible d’utiliser permet le regroupement d’organismes sous une étiquette unique pour en venir à une classification de type phylogénique (Sneath et Sokal, 1973). Il s’agit d’une des premières tentatives répertoriées d’un réseau d’agrégation (cluster), concept qui est toujours aujourd’hui à la base de la microbiologie du sol et intégré dans les algorithmes de création d’OTUs (Edgar, 2010).
Bien qu’au départ, les traits observables aient été utilisés, l’avènement du séquençage d’ADN par la méthode techniquement robuste de Sanger et la découverte de l’amplification par PCR ont drastiquement modifié l’étude des microorganismes (Shendure et Ji, 2008). Les études phylogéniques ont pu être basées directement sur les séquences nucléiques des organismes et non pas seulement sur le phénotype. Un nouveau cap a été franchi au tournant des années 2000 lorsque les techniques de séquençage de nouvelle génération (NGS) sont devenues plus fonctionnelles et accessibles à la communauté scientifique (Cardenas et Tiedje, 2008; Shendure et Ji, 2008). Cela a permis de séquencer plus rapidement de plus petites séquences à un bon niveau de précision.
10
Cette qualité satisfaisante n’atteint pas la robustesse des résultats auparavant obtenus avec la méthode de Sanger (Mardis, 2008). Depuis, les appareils, techniques et méthodes de NGS ne cessent de gagner en efficacité, particulièrement par la diminution du coût par séquençage (Caporaso et al., 2012; Vincent et al., 2017).
Les techniques de NGS sont incontestablement plus prolifiques au niveau du nombre de séquences produites, mais cet avantage constitue également un défi de taille : la gestion efficace des gros jeux de données produits. Le développement des NGS s’est fait de concert avec le développement de la bioinformatique. Celle-ci permet de traiter efficacement les millions de séquences nucléiques produites par les techniques de NGS (Vincent et al., 2017). Les traitements bioinformatiques nécessitent plusieurs étapes d’assemblage et d’alignement pour en venir à extraire des OTUs de qualité (Edgar et Flyvbjerg, 2015; Vincent et al., 2017).
Initialement, avec l’utilisation du séquençage robuste de Sanger, le seuil d’identité permis entre les séquences pour créer les OTUs a été fixé à 97 %. Ce seuil a été conservé jusqu’à présent, permettant de balancer les écarts de précision dans le séquençage. C’est donc dire que pour que deux séquences nucléiques soient agrégées au même OTU les séquences ne peuvent pas différer de plus de trois paires de bases sur 100. Ce seuil considère la longueur totale des séquences étudiées. Ces deux séquences comptent alors comme deux représentantes d’un même taxon virtuel. Ce critère d’identité pour la création des OTUs a été remis en question dernièrement puisqu’il ne permet pas d’identifier notamment des séquences nucléiques véritables pour l’identification biologique (Callahan et al., 2017). Les séquences nucléiques représentant les OTUs à 97% sont créés par le traitement bioinformatique et n’ont pas nécessairement de séquences équivalentes dans les données brutes amplifiées à partir de l’ADN extrait du sol. Ce problème limite la comparaison des OTUs obtenues entre les études. Le concept de « Variantes de séquences d’amplicons » (ASVs) a été mis de l’avant pour y pallier, également dénommé dans la littérature scientifique sous le vocable « Zero-OTUs » (ZOTUs). Les ASVs constituent des séquences biologiques identiques à 100 %. Chaque réseau d’agrégation regroupe pour chaque ASVs des séquences où toutes les bases nucléiques sont identiques pour chaque séquence la composant (Edgar, 2017). Les ASVs décrivent plus fidèlement la variabilité génotypique présente en comparaison des OTUs puisque le critère d’agrégation est plus sévère. Ce niveau de précision supérieur présente un intérêt pour mieux discerner les
11
fonctions reliées aux génotypes des microorganismes (Callahan et al., 2017; Edgar, 2017). L’approche des ASVs conduit en pratique à un plus grand nombre d’ASVs que d’OTUs générés pour un même jeu de données. Cela représente un frein à l’utilisation répandue des ASVs comme variable descriptive de la diversité microbiologique des sols de par la difficulté de gestion bioinformatique. Cette contrainte technique a orienté l’utilisation bien établie des OTUs dans le présent projet.
0.2.2 Techniques d’analyse écologique
Une fois les OTUs obtenues, la diversité spécifique peut être calculée par l’utilisation de différents indices. L’abondance est la mesure qui témoigne de la quantité, du nombre d’individus composant la communauté (McCune et al., 2002c). L’utilisation de NGS telle que la technologie MiSeq ne permet d’obtenir que des valeurs relatives d’abondance et non absolues. Avec cette technologie, l’amplification est menée à une profondeur donnée, soit le nombre de répétitions désiré par échantillon (Shendure et Ji, 2008). Cela permet de conserver les proportions respectives des microorganismes, d’amplifier uniformément les échantillons. Un portrait global des communautés est accessible pour l’analyse, mais les espèces rares ne sont alors pas les mieux détectées. Il s’agit d’une limitation technique qui complexifie les comparaisons entre des échantillons amplifiés à des profondeurs variables et lors de différents essais (Caporaso et al., 2012). Les abondances obtenues par la technologie MiSeq peuvent seulement être traitées de façon relative entre les traitements, non pas pour définir des seuils d’abondance absolus de microorganismes présents ou non (McCune et al., 2002a).
La richesse se définit comme un indicateur du nombre d’espèces différentes présentes, mais ne tient pas compte de l’abondance de chacune (McCune et al., 2002c). La diversité constitue une valeur unique calculée conjointement avec l’abondance et la richesse des OTUs. Une valeur basse de la diversité témoigne d’un milieu dominé par quelques taxons bien abondants. Au contraire, une valeur élevée sera le signe d’un milieu plus riche en OTUs avec des abondances plus modérées (McCune et al., 2002c). La diversité peut être calculée de différentes manières, notamment par les indices de Chao, Shannon et Simpson. Ces indicateurs témoignent du comportement de différents taxons (McCune et al., 2002c). La richesse d’une communauté témoigne mieux des taxons plus rares, tout comme l’indice de Chao, la diversité de Simpson des taxons plus abondants et la diversité
12
de Shannon serait un indicateur plus balancé des communautés, témoignant aussi bien du comportement des taxons rares que dominants du milieu (McCune et al., 2002c; Morris et al., 2014). L’étude des relations entre les OTUs est possible en tenant compte de l’ensemble des covariances entre chaque échantillon. Des matrices de distances sont utilisées pour permettre d’étudier simultanément les variations multivariées entre chaque OTUs à travers tous les traitements, soit la structure des communautés (McCune et al., 2002c). L’étude multivariée des OTUs est complétée par l’utilisation de techniques d’ordination, bien qu’elles ne soient pas à proprement parler des outils statistiques. L’ordination permet de lier les variations des OTUs aux variations présentes dans les traitements appliqués ou aux gradients des caractéristiques environnementales du milieu (McCune et al., 2002b).
L’étude des fonctions des communautés microbiennes des sols agricoles est l’objectif pratique visé dans une perspective agronomique. Une OTU peut agir en tant qu’agent saprophyte, endophyte, pathogène ou réaliser la dégradation d’un composé donné (Anderson et Habiger, 2012; Bainard et al., 2017; Bergkemper et al., 2016). Historiquement, déterminer les fonctions d’un organisme passait par sa culture en laboratoire. Cela représente un frein pour les organismes qu’il n’est pas possible de cultiver en laboratoire (Kandeler, 2015). En parallèle de l’identification par NGS, il est possible d’inférer les fonctions d’une OTU par homologie de la séquence obtenue avec les banques de données phylogénétiques déjà existantes en ligne. Des index de fonctions existent en ce sens (Aßhauer et al., 2015; Nguyen et al., 2016). Il faut être conscient que la précision taxonomique de l’identification pourra jouer sur les résultats. Des interprétations basées sur des OTUs identifiées au phylum pourraient créer une perte d’information par rapport à une identification des OTUs au niveau de l’ordre ou du genre (Degrune et al., 2015). Autrement, la métagénomique permet d’amplifier des gènes spécifiques ou des protéines d’un environnement pour les étudier, sans passer nécessairement par l’identification des organismes les produisant (Fraser et al., 2015).
0.3 Facteurs influençant le microbiome du sol
0.3.1 Caractéristiques édaphiques
Pour bien comprendre les variations dans la composition et la diversité du microbiome résultant des pratiques culturales, il est essentiel d’exposer les mécanismes affectant
13
l’accès aux éléments nutritifs pour les microorganismes. Ces mécanismes physiques et chimiques affectent les fractions solide, liquide et gazeuse dans les sols. La fraction solide se divise en une portion inorganique et une autre organique, chacune aux comportements bien distincts. Pour être absorbés par les microorganismes, les éléments nutritifs doivent se retrouver disponibles dans la solution du sol. Celle-ci agit comme un milieu essentiel pour la propagation de la vie grâce à la grande réactivité de l’eau (Morris et Blackwood, 2015).
Les éléments nutritifs sont partie intégrante de la composition des minéraux primaires et secondaires. Les minéraux primaires sont issus des processus de formations géologiques sous l’action de la chaleur et de la pression terrestre. Les minéraux secondaires découlent d’une altération subséquente en surface ou peuvent être néoformés à partir d’éléments auparavant en solution dans le sol (Shen et al., 2011). Une fraction minérale plus dégradée, plus petite que 2 mm, est décrite comme faisant partie de la texture du sol. Il est alors question des proportions en sable, limon et argile qui permettent de décrire la taille des particules. La texture est une caractéristique importante pour décrire un sol agricole. La taille des particules, leur capacité d’échange ionique et leur surface spécifique influencent l’adsorption ainsi que la désorption des éléments nutritifs tout comme de l’eau. La texture du sol influence la perte d’éléments nutritifs par ruissellement, lessivage et érosion (Angers et al., 2010). Combiné à l’action des microorganismes, les particules de sols peuvent se structurer différemment, avoir des organisations tridimensionnelles plus propices à la croissance des cultures, à la solubilisation de la fraction inorganique, à la minéralisation de la fraction organique, et à la circulation de l’air ainsi que de l’eau dans le profil de sol (Varoney et Heck, 2015).
La fraction organique, définie communément sous le vocable matière organique du sol comprend l’ensemble du carbone organique présent dans les organismes vivants aussi bien que morts, en décomposition et leurs produits métaboliques. Le taux de matière organique d’un sol agricole est souvent considéré comme un indicateur de fertilité (Angers et al., 2010). Le calcul de la biomasse microbienne est utilisé pour estimer la quantité globale des microorganismes actifs dans les sols via le CO2 dégagé par la
respiration cellulaire (Moorberg et Crouse, 2017). Bien qu’étant une donnée quantitative, les fluctuations de la respiration de la biomasse microbienne ne constituent qu’un
14
indicateur qualitatif de l’activité microbienne du sol. Elles ne témoignent pas directement de la composition du microbiome (Bissett et al., 2013).
Le microbiome du sol joue un rôle important dans les mécanismes de désintégration, de solubilisation, de minéralisation et d’immobilisation. La désintégration réfère à la dégradation physique et chimique des roches. La désintégration physique survient par l’action de la température, de la pression et des cycles gels-dégels. La désintégration peut survenir au niveau chimique, par l’action des acides organiques résultant de l’activité métabolique du microbiome (Duarte et al., 2017). La solubilisation est le processus permettant aux éléments nutritifs intégrés dans les minéraux ou adsorbés sur les particules minérales d’être désorbés. Cela se produit par l’action des acides organiques sous leurs formes ioniques dans la solution du sol (Jones et Oburger, 2011; Masood et Bano, 2016). Chaque acide organique a un pouvoir désintégrateur ainsi que solubilisant différent selon les particules minérales initiales et peut avoir un champ d’action très spécifique ou général par rapport à ce substrat (Sharma et al., 2013). La minéralisation concerne l’action d’enzymes spécifiques produites par les microorganismes. Ces enzymes permettent de dégrader la matière organique pour qu’elle soit accessible dans la solution du sol. Il existe toute une gamme d’enzyme plus ou moins spécifique selon le pouvoir récalcitrant à la dégradation de la matière organique (Geisseler et al., 2010; Mohammadi, 2012). L’immobilisation survient dans les sols lorsque la balance entre les éléments nutritifs n’est plus équilibrée. Les microorganismes accaparent alors certains éléments nutritifs du sol pour leur propre métabolisme au détriment des cultures (Robertson et Groffman, 2015; Stevenson, 1986). Le phénomène d’immobilisation est préoccupant en agriculture lors d’apports majeurs de carbone, dans le cas de résidus de culture retournés ou de fertilisation à base d’effluents d’élevage solides. Cela peut notamment limiter l’azote accessible pour les plantes (Chabot et al., 2010).
L’acidité de la solution du sol est une caractéristique édaphique à considérer dans l’étude des communautés microbiologiques. Elle affecte la disponibilité des éléments nutritifs, l’activité enzymatique et la croissance microbienne. Chaque élément nutritif a un pH optimal, souvent autour de la neutralité, qui lui permet d’être disponible dans la solution du sol pour les plantes et les microorganismes. L’adsorption, autant sur les particules minérales qu’organiques, présente un frein à leur disponibilité (Hébert et al., 2010). Plusieurs études ont démontré que le pH du sol est un facteur permettant de décrire les
15
fluctuations de la diversité bactérienne dans les sols. Son impact sur les phyla
Acidobacteria, Actinobacteria et Bacteroidetes a été démontré, mais il n’est pas bien
corrélé aux variations des communautés fongiques. La croissance des microorganismes peut être influencée, aussi bien positivement que négativement selon l’espèce, par l’acidité. Les conditions optimales fluctuent selon l’espèce, de même que l’adaptabilité de chaque espèce aux contraintes de l’environnement (Frey, 2015; Voroney et Heck, 2015). À long terme, la production d’acides organiques et d’enzymes par les microorganismes acidifie les sols. L’utilisation répétée d’engrais, surtout les engrais azotés minéraux, contribue également à cette acidification (Hébert et al., 2010; Hedley et Bolan, 2003).
0.3.2 Impacts des pratiques culturales
Les compositions bactérienne et fongique des sols varient selon les changements dans les caractéristiques de sols découlant des pratiques culturales. La composition des communautés bactériennes varie selon les systèmes à l’étude. Certains phyla dominants constituent ensemble plus de 50 % de l’abondance relative à travers les études consultées:
Acidobacteria, Actinobacteria, Bacteroidetes, Chloroflexi, Firmicutes et/ou
Proteobacteria (Chen et al., 2016; Degrune et al., 2015; Hartmann et al., 2015; Lienhard
et al., 2014; Navarro-Noya et al., 2013; Sun et al., 2015; Wang et al., 2017). Les
Ascomycota et les Basidiomycota, deux phyla majoritairement saprophytes, composent
généralement plus de 70 % de l’abondance relative fongique d’un sol agricole (Chen et al., 2016; Degrune et al., 2015; Hartmann et al., 2015; Lienhard et al., 2014; Panelli et al., 2017; Sun et al., 2016; Wang et al., 2017). De 30 à 60 % des champignons du sol peuvent être des espèces pathogènes selon la rotation de cultures (Bainard et al., 2017). Plusieurs genres composent les communautés de champignons mycorhiziens arbusculaires en contexte agricole : Glomus, Ambispora, Acaulospora, Archeospora, Claroideoglomus,
Diversispora, Funneliformis, Gigaspora, Paraglomus, Rhizophagus, Scutellospora et Septoglomus. Les genres dominants et les proportions de chaque taxon varient selon les
études (Alguacil et al., 2014; Manoharan et al., 2017; Qin et al., 2015).
0.3.2.1 Espèces végétales et rotation des cultures
Le microbiome du sol est influencé principalement par le microbiome de l’espèce végétale en culture et secondairement par les autres végétaux présents dans le champ (Bainard et al., 2017). L’interface sol-racine, la rhizosphère, est un milieu où l’activité
16
microbienne est intense étant donné la quantité importante d’exsudats racinaires qu’une plante peut produire et qui sert de source de nutriments pour les microorganismes (Bakker et al., 2015; Donn et al., 2015). La plasticité des racines influence le comportement du microbiome de la rhizosphère (Donn et al., 2015). La nature et la quantité des exsudats racinaires produits diffèrent selon les espèces végétales, favorisant ainsi des espèces microbiennes particulières (Musilova et al., 2016; Nguyen, 2003; Pausch et Kuzyakov, 2018). La variabilité génétique des plantes (ex : différents cultivars) modifie la conformation des racines (Corneo et al., 2016) et les communautés microbiennes associées aux racines (Bazghaleh et al., 2015). Cette variabilité peut favoriser différents microorganismes qui influencent ensuite l’implantation des cultures subséquentes (Ellouze et al., 2013; Estaún et al., 2010).
Le choix de la succession des cultures présente des intérêts agronomiques et écologiques par cette influence des espèces végétales sur le microbiome du sol. Des impacts positifs majeurs sont envisageables pour le producteur qui porte une attention particulière au choix de la rotation des cultures : meilleur contrôle des maladies et des mauvaises herbes, structure de sol améliorée ainsi que meilleure utilisation des nutriments tout comme de l’eau (Reeves, 2017). L’implantation successive de cultures de la même famille augmente la proportion de champignons pathogènes pour cette culture dans les sols (Bainard et al., 2017). Des rotations équilibrées vont mener à des inocula plus faibles de pathogènes (Dias et al., 2015). Bien que les cultures annuelles sont moins propices à la prolifération des mycorhizes que les cultures pérennes, des rotations comprenant des plantes pouvant établir une symbiose avec les CMA favorisent l’établissement des symbioses pour les années subséquentes (Fortin et al., 2015b).
0.3.2.2 Travail de sol
Le travail de sol agit de deux manières distinctes dans les sols : (1) il cause d’abord une perturbation mécanique dans la structure existante du sol et (2) il crée une incorporation des résidus ainsi qu’un brassage des particules dans le profil (Smith et al., 2015). Des particules de sol auparavant stabilisées se retrouvent alors sujettes à la solubilisation et à la minéralisation (Chávez-Romero et al., 2016). Le travail réduit stabilise la matière organique totale dans les sols et la rend moins accessible à une dégradation rapide par les microorganismes (Gupta et Germida, 2015). L’intensité du travail de sol s’évalue sur un
17
gradient allant d’une forte perturbation de sol, le labour, à une plus limitée, le travail réduit, ou presqu’inexistante en semis direct.
Un travail intensif du sol, en l’occurrence le passage annuel d’une charrue à disques combiné à une herse à disques pendant 14 ans a diminué la biomasse microbienne des sols argileux acides tropicaux, des oxysols, comparativement au semis direct (Hungria et al., 2009). La biomasse microbienne n’a pas été réduite après 20 ans pour des loams argileux sableux légèrement acides travaillés avec une herse à disques déportés par rapport à un semis direct. Dans cette étude, aucun effet négatif du travail de sol plus agressif sur les traits fonctionnels des sols n’a été enregistré (Bissett et al., 2013). En abondance absolue, après 31 ans de monoculture de coton sur un loam sableux, les bactéries tendent à être plus abondantes que les champignons dans les sols agricoles, même en condition de travail réduit (Mbuthia et al., 2015). Cela s’explique notamment par la physiologie de ces deux groupes : les champignons ont un avantage compétitif sur les bactéries lorsque des formes de carbone plus récalcitrantes sont à dégrader (Lee et Sinsabaugh, 2015).
La richesse bactérienne a été plus élevée que celle fongique pour un labour effectué sur un loam sableux comparativement à un travail réduit, alors qu’elle a été plus faible en travail réduit (Degrune et al., 2015). La dégradation physique des agrégats causée par le labour rend disponible rapidement les nutriments pour les bactéries, ce qui augmente leur diversité (Acosta-Martínez et al., 2008). Cette même perturbation par labour dans un oxysol argileux a nui à la prolifération des champignons en détruisant physiquement les réseaux d’hyphes, diminuant ainsi la diversité fongique (Lienhard et al., 2014). Les CMA sont reconnus pour être favorisés sous travail réduit en opposition au travail conventionnel. Ils se propagent préférentiellement par leurs réseaux d’hyphes qui se retrouvent moins perturbés en travail réduit (Brito et al., 2011). La diversité mycorhizienne est négativement reliée à l’intensité du travail de sol (Oehl et al., 2010).
La composition bactérienne fluctue selon un gradient d’intensité du travail de sol (Acosta-Martínez et al., 2008). De nombreux groupes taxonomiques présentent une préférence pour les sols perturbés, soit pour le labour, ou le travail réduit. Toutefois, les phyla bactériens Acidobacteria, Actinobacteria et Proteobacteria ont été favorisés en pâturage comparativement au labour. Cela a été respectivement attribué à l’utilisation de substrats
18
organiques complexes, à une sensibilité aux perturbations mécaniques et aux bactéries copiothrophes (Lienhard et al., 2014). Les Acidobacteria sont reconnus pour bien proliférer en milieu peu perturbé par leurs capacités oligotrophes (Ortiz-Cornejo et al., 2017). Au contraire, les Firmicutes ont été favorisés en travail conventionnel par rapport aux pâturages, ce qui a été lié à leur capacité à produire des endospores, structure de reproduction permettant de survivre aux conditions adverses (Lienhard et al., 2014). L’analyse de taxons inférieurs a permis de détecter des bactéries favorisées en travail conventionnel (Degrune et al., 2015): (1) des méthanotrophes [Methylocystaceae] (Conrad, 1996), (2) des bactéries en mesure de dégrader des résidus d’herbicides [Sphingomonas] (Sørensen et al., 2001), (3) ou capables de dégrader des composés organiques complexes [Saprospiraceae] (McIlroy et Nielsen, 2014), (4) des bactéries de la famille diversifiée des Oxalobacteraceae (Baldani et al., 2014) et (5) des bactéries ayant la capacité de dégrader la chitine [Chitinophaga] (Sangkhobol et Skerman, 1981).
Le travail de sol a un effet sur la composition des communautés fongiques du sol. Les champignons favorisés par le travail conventionnel s’adaptent à la minéralisation rapide de la matière organique fraîche. Ceux dominants en travail réduit exploitent préférentiellement les sources de matière organique récalcitrantes (Sharma-Poudyal et al., 2017). Les abondances relatives des phyla fongiques ont augmentées sous travail conventionnel pour les Ascomycota et les Chytridiomycota tandis que les Basidiomycota ont été favorisés sous pâturage ou en travail réduit (Degrune et al., 2015; Lienhard et al., 2014). Ces préférences sont attribuées dans la littérature aux aptitudes saprophytiques des
Ascomycota (Wang et al., 2016b), à la capacité de certaines espèces de Chytridiomycota
de bien survivre dans des environnements plus sujets à la sécheresse (Gleason et al., 2004) et aux aptitudes des Basidiomycota à dégrader la lignine ainsi que la cellulose en conditions anaérobiques (Boer et al., 2005). L’abondance des communautés mycorhiziennes est affectée négativement par un travail de sol intensif. Le genre Glomus demeure tout de même relativement dominant dans les sols arables (Avio et al., 2013). Le travail de sol induit des compositions de communautés mycorhiziennes bien distinctes entre les traitements (Alguacil et al., 2014; de Pontes et al., 2017; Mbuthia et al., 2015; Säle et al., 2015). Il ne s’agit pas d’un facteur déterminant modulant la croissance des CMA dans tous les cas (Bowles et al., 2017; Schalamuk et al., 2006). Quelques espèces de la famille des Glomeraceae sont dominantes dans les sols agricoles, soit les genres
19
de sélection favorisant les CMA pouvant survivre au travail de sol plus intensif (Oehl et al., 2003), particulièrement dans le cas des Glomus (Qin et al., 2015). Peu d’informations est disponible quant à l’impact du travail de sol sur des genres mycorhiziens particuliers. Les Claroideoglomus et Funneliformis sont favorisés par le labour du fait de leur résilience aux perturbations mécaniques (Alguacil et al., 2014). Les Ambispora,
Diversispora, Gigaspora, Rhizophagus et Septoglomus sont favorisés dans des milieux
moins perturbés (Alguacil et al., 2014; Manoharan et al., 2017; Soka et Ritchie, 2018).
0.3.2.3 Fertilisation des cultures
Les stratégies de fertilisation des producteurs sont déterminées par les besoins des cultures, les caractéristiques édaphiques et la disponibilité des fertilisants. La composition de l’engrais, les techniques d’épandage, la température, la disponibilité en eau et le stade physiologique de la culture sont tous des facteurs qui moduleront l’accessibilité des éléments nutritifs à la suite de l’application. Les engrais de synthèse sont pratiques au niveau de la manutention et permettent une bonne précision quant à la formulation d’engrais équilibrée ne contenant que les éléments minéraux désirés. La production de ce type d’engrais a néanmoins un impact environnemental : l’utilisation d’énergie fossile lors de leur fabrication ainsi que la création de coproduits dommageables pour l’environnement (Gagné et Parent, 2010). Le contenu en éléments nutritifs des effluents d’élevage est plus variable et fluctue selon les régies des élevages. Ces effluents fournissent des nutriments sous formes inorganiques et organiques. Cette fraction organique des effluents d’élevage se minéralise graduellement par l’action des microorganismes au courant de la saison de culture (CRAAQ, 2013). Le tableau 1 montre les différences dans la composition en C et N selon les effluents d’élevage nous intéressant dans la présente étude (CRAAQ, 2013).
Tableau 1. Rapport C/N et teneurs moyennes en N des effluents d’élevage à l’étude.
Effluents d’élevage C/N Ntotal (kg/t) N-NH4 (kg/t) N-NH4 / Ntotal (%) Lisier - porcs 3,4 3,5 2,4 69
Lisier – bovins laitiers 10 2,6 1,4 54
Fumier - poulets 11 27,5 10,4 38
L’utilisation de fertilisants stimule globalement l’activité microbienne, accélère la minéralisation et le recyclage des éléments nutritifs par le biais des cycles







![Figure 5. Impacts de la gestion des résidus de culture, retournés (R) versus exportés (E), sur les abondances relatives des principaux phyla bactériens (n = 30 pour chaque barre) composant les communautés du loam sableux [LS] (A) et de l’argile limoneuse](https://thumb-eu.123doks.com/thumbv2/123doknet/2916963.76138/84.892.152.757.157.958/retournés-exportés-abondances-principaux-bactériens-composant-communautés-limoneuse.webp)
