© Charles-Antoine Boyer, 2020
Recherche de nouvelles capsides du virus
adéno-associé porcin, et suivi de leur efficacité de
transduction
Mémoire
Charles-Antoine Boyer
Maîtrise en microbiologie-immunologie - avec mémoire
Maître ès sciences (M. Sc.)
Recherche de nouvelles capsides du virus adéno-associé
porcin, et suivi de leur efficacité de transduction
Mémoire
Par
Charles-Antoine Boyer
Sous la direction de :
Gary Kobinger, directeur de recherche Chung Kei ‘Gary’ wong, codirecteur de recherche
ii
Résumé du mémoire
En thérapie génique, les virus adéno-associés (AAV) ont prouvé maintes fois qu’ils étaient capables de transférer un gène désiré dans différents types de tissus cibles. La plupart des humains possèdent déjà une pré-immunité existante contre les sérotypes humains d’AAV, ce qui se caractérise par une réponse robuste des anticorps contre la capside virale contenant le transgène avant qu’il n’atteigne sa cible thérapeutique. Dû à ce phénomène, les nouveaux AAV avec une faible séroprévalence sont hautement attractifs comme véhicules de transfert de gènes. Des études ont démontré que les AAV découverts, provenant du porc comme AAVpo1 ou AAVpo2, ne sont pas reconnus par la pré-immunité humaine. Ce mémoire décrit des objectifs primaires de recherche comme l’isolation de nouveaux sérotypes d’AAV provenant des fèces de porc, la production des nouveaux vecteurs viraux et la caractérisation de leur capacité de transduction in vitro. Les résultats ont démontré que les nouveaux vecteurs AAV porcins sont capables de transduire une bonne variété de cellules et de tissus. Aussi, des évidences d’échanges de génome du gène CAP entre 2 AAV ont été trouvées durant le processus de la recherche. Même si les nouveaux vecteurs AAV ont réussi à transduire des lignées cellulaires avec succès, des études plus poussées sur la bio distribution du vecteur chez les souris seront nécessaires pour vérifier la fiabilité de ces nouveaux vecteurs.
iii
Table des matières
RESUME DU MEMOIRE ... II TABLE DES MATIERES ... III LISTES DES TABLES ... V LISTE DE FIGURES ... VI LISTE DES ABREVIATIONS ... VII REMERCIEMENTS ... IX
INTRODUCTION ... 1
1-GENERALITES : ... 1
1.1- Les caractéristiques des virus adéno-associés ... 1
1.2-Historique... 1
2-TAXONOMIE DES PARVOVIRUS ET L’ORGANISATION GENOMIQUE DES DIFFERENTS SOUS-GROUPES DE PARVOVIRIDAE. ... 2
3-SPECTRE D’HOTE ET IMMUNITE PREEXISTANTE ... 5
3.1- Tropisme ... 5
3.2- Ligands et Récepteurs ... 6
3.3- Immunité préexistante ... 7
4-CARACTERISTIQUES DU VIRION ... 8
4.1- Les propriétés du virion ... 8
4.2- Propriétés biochimiques ... 8
4.3- Propriétés antigéniques ... 8
5-BIOLOGIE MOLECULAIRE DES AAV ... 9
5.1- Nature du génome ... 9
5.2- Transcription des gènes du virus associé ... 9
5.3- Transcription et traduction des gènes des AAV ... 10
5.4- Réplication des AAV ... 12
6-PRODUITS DU GENOME ... 13
6.1- Protéines structurales ... 13
6.2- Protéines non structurales ... 13
6.3 Protéines auxiliaires ... 14
7-MORPHOLOGIE ET STRUCTURE... 15
7.1- En général ... 15
7.2 Les boucles chez les AAV ... 16
7.3 Les interactions des sous-unités. ... 16
8-TRAFIC INTRACELLULAIRE DES AAV ET LEUR ENTREE DANS LE NOYAU. ... 18
9-LES DIFFERENTES METHODES UTILISEES POUR ISOLER LES AAV JUSQU’A AUJOURD’HUI... 19
9.1- Les rongeurs ... 19 9.2- Le Porc ... 19 9.3- Les Reptiles ... 19 9.4- Les oiseaux ... 20 9.5- Les Chèvres ... 20 9.6- Le bœuf ... 20
9.7- Les primates non humains ... 21
9.8- Les humains ... 21
10-RAISONS POUR RECHERCHER DES AAV DANS LES TISSUS DE PORCS. ... 23
10.1- Similarité entre l’humain et le porc ... 23
iv
CHAPITRE 1 : OBJECTIFS ET HYPOTHESES ... 25
CHAPITRE 2 : MATERIEL ET METHODES ... 27
1-ISOLATION DE L’ADN GENOMIQUE DES FECES DE PORC, DETECTION DES AAV ET AMPLIFICATION DU GENE CAP ... 27
2-TRAITEMENT DE L’ADN GENOMIQUE A LA DNASE DEPENDANT DE L’ATP SURE POUR LES PLASMIDES ... 31
3-CLONAGE DU GENE CAP DES NOUVEAUX AAV ET INSERTION DU GENE CAP DANS LE PLASMIDE DJ ... 32
4-PRODUCTION DE CAPSIDES D’AAV AVEC UN TRANSGENE ET PURIFICATION ... 36
4.1- Les plasmides impliqués dans la production des AAV recombinés. ... 36
4.2- Culture cellulaire et transfection tri-plasmides. ... 37
4.3- Purification des particules d’AAV ... 39
4.4- Titration des particules d’AAV avec la PCR en temps réel ... 39
5-ÉVALUATION DE L’EFFICACITE DE LA TRANSDUCTION DES NOUVEAUX AAV IN VITRO. ... 43
CHAPITRE 3 : RESULTATS ... 44
1-IDENTIFICATIONS DE NOUVELLES SEQUENCES D’AAV VENANT DE FECES DE PORC VIA PCR ... 44
2-EFFICACITE DU TRAITEMENT PRE-PCR DE L’ADN GENOMIQUE A LA DNASE DEPENDANT DE L’ATP SURE POUR LES PLASMIDES. ... 51
3-VERIFICATION DE LA FONCTIONNALITE DES NOUVELLES CAPSIDES DES AAV. ... 52
CHAPITRE 4 : DISCUSSION ... 54
1-L’EFFICACITE DE DNASE DEPENDANT DE L’ATP SURE POUR LES PLASMIDES POUR AMELIORER LA DETECTION DES AAV. . 54
2-ISOLATION ET PRODUCTION DE NOUVEAUX SEROTYPES D’AAV PORCINS. ... 55
3-RECOMBINAISON DU GENOME VIRAL DES AAVPOP. ... 58
CONCLUSION. ... 62
v
Listes des tables
Table 3.1 : Le cycle PCR pour l’isolation du gène CAP d’AAV
Table 3.2- Mélange d’une réaction de RT-PCR pour la titration d’AAV recombinés.
Table 6.1- Pourcentage d’identité basé sur l’homologie de séquences des différents sérotypes d’AAV.
vi
Liste de figures
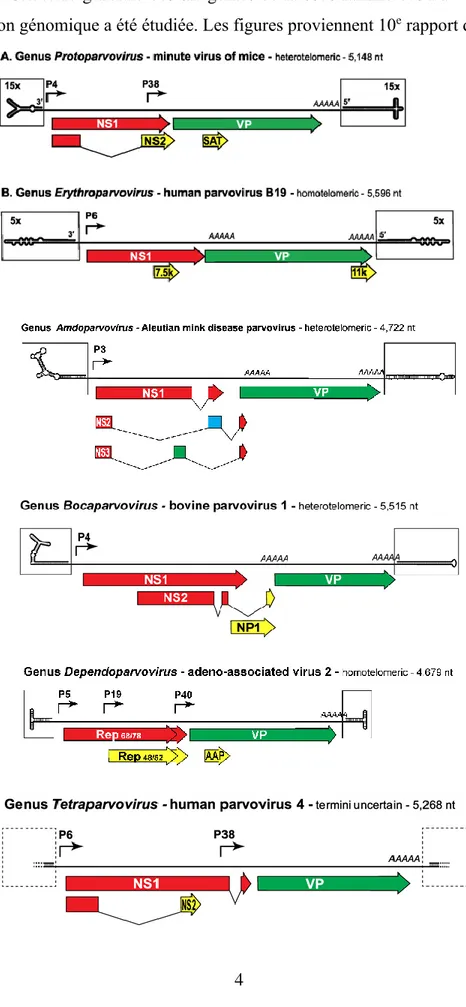
Figure 1.2 : Structure générale des six genres de la sous-famille des Parvovirinae dont l’organisation génomique a été étudiée.
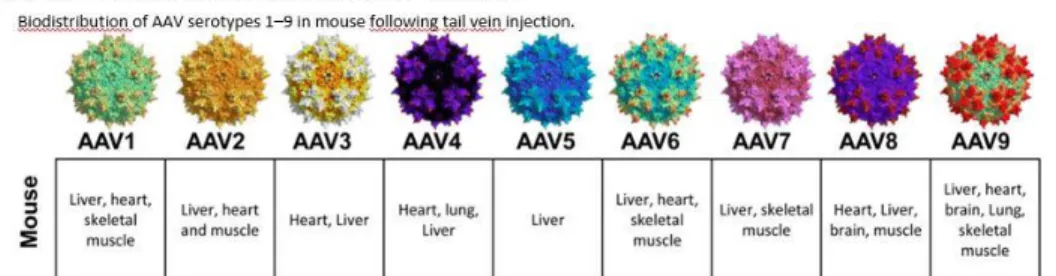
Figure 3 : Tropisme des différents AAV par rapport aux différents tissus de souris
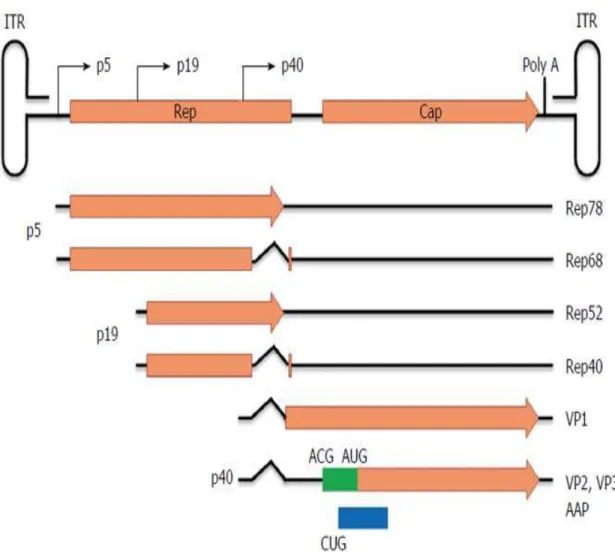
Figure 5 : Représentation schématique du génome d’AAV.
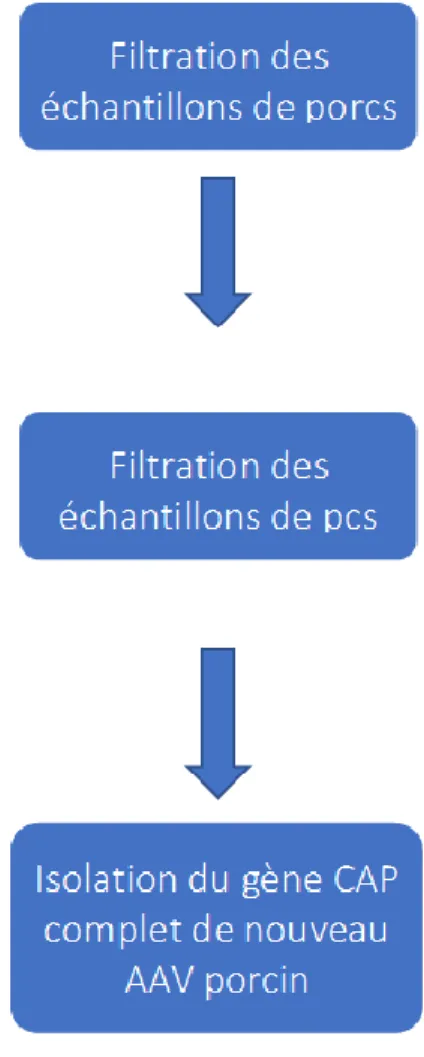
Figure 2.1 : Survol de l’isolation de nouveaux AAV porcins
Figure 2.2 : clonage de plasmide avec gène CAP par transformation chimique de E.coli competent
Figure 2.3 : Production de vecteurs d’AAV recombinés
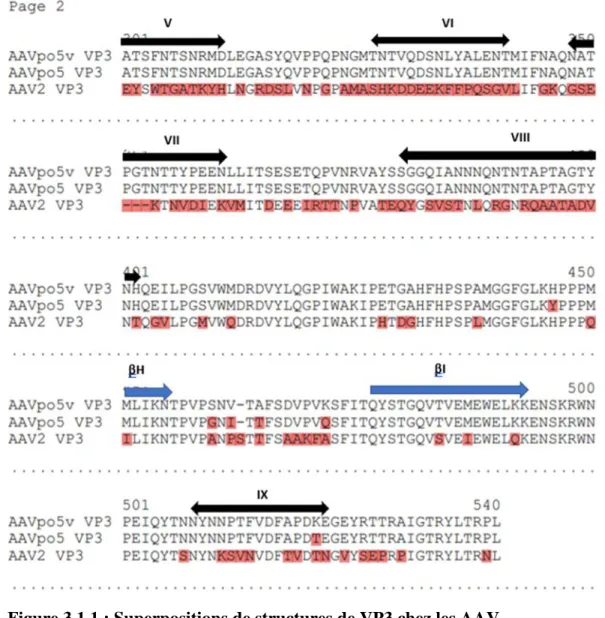
Figure 3.1.1 : Superpositions de structures de VP3 d’AAV
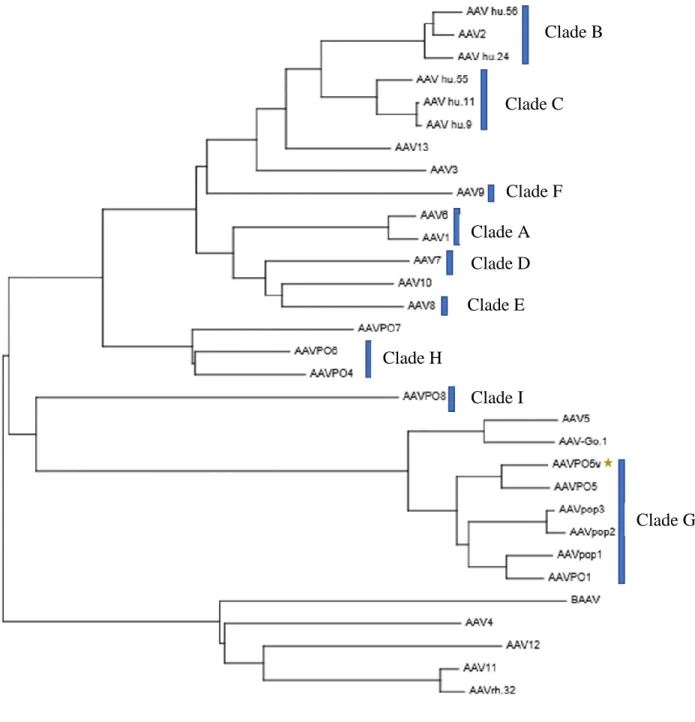
Figure 3.1.2 : Arbre phylogénétique pour la comparaison des nouveaux AAV porcins
Figure 3.1.2 : Arbre phylogénétique pour la comparaison des AAVPOPv porcins
Figure 3.1.3 : Alignements généraux des différents AAVPOP
Figure 3.2 : PCR sur l’ADN génomique traité à la Dnase dépendant de l’ATP sûre pour les plasmides.
vii
Liste des abréviations
AAV : virus adéno-associés.ORF : cadre de lecture ouvert.
ITR : séquences terminales inversées.
ICTV : Comité international sur la taxonomie des virus.
HSPG : protéoglycanes à héparane sulfate
FGFR1 :récepteur de facteur de croissance de fibroblaste 1
LamR : Récepteur laminine
Nabs : anticorps neutralisants
HSV : virus de l’herpès simplex
AAP : Protéine activatrice d’assemblement RFM : monomère à forme réplicative
RFD : dimère à forme réplicative
PLA2 : domaine phospholipase A2
NLS : signaux de localisation nucléique
NPC : complexe de pore nucléique
MVM : virus minute de la souris
Bp : paire de base
TAIL : thermal-asymmetric interlaced
LB :bouillon de lysogénie
CHUL : centre de recherche du CHU de Québec-Université Laval
HB : hotte biologique.
viii FBS : sérum de veau fœtal
CalPhos : phosphate de calcium
GC : nombre de copies du génome
RT-PCR : PCR en temps réel
BMS : Mic qPCR cycler
PBS : phosphate tamponné salin
X-gal: 5-bromo-4-chloro- 3-indolyl b-D-galactoside
ADD: Dnase dépendant de l’ATP sûre pour les plasmides
rvI: région variable I
rvII: région variable II
rvIII: région variable III
MDPV: parvovirus du canard de Moscovite
GPV: parvovirus de l’oie WT : état naturel
rAAV : AAV recombinants
TLR2 : récepteurs de type Toll 2
TLR9 : récepteurs de type Toll 9
IgG : immunoglobuline G
IgG1 : immunoglobuline G1
IgG2 : immunoglobuline G2
ix
Remerciements
D’abord et avant tout, je tiens à exprimer ma plus grande gratitude à mon directeur de maîtrise, le Dr Gary P. Kobinger. Je le remercie pour son support, sa direction, ses pépites de sagesse et surtout de m’avoir pris comme étudiant alors qu’il avait plus de personnel que nécessaire. Je le remercie également pour ses encouragements, son ouverture d’esprit ainsi que pour la liberté d’expérimentation qu’il m’a donnée. Dr Kobinger, j’ai beaucoup apprécié mon temps au sein de votre équipe et je vais toujours chérir cette expérience. Je voudrais aussi remercier tout le personnel du département d’infectiologie du centre hospitalier de l’université Laval (CHUL) pour leur enseignement et pour les indications. Je veux également exprimer ma reconnaissance à tous les membres du laboratoire du Dr Gary Kobinger pour leur support et l’enseignement que j’ai reçu durant mes études. Ces moments passés avec vous resteront inoubliables.
Je voudrais remercier tout spécialement mon mentor, Claude Lamarre, pour tous les précieux conseils et son support indéfectible lors mes recherches sur les AAV. Grâce à vous, j’ai beaucoup appris et le temps que vous avez investi dans ma formation me touche énormément. J’emporterai avec moi tout le savoir que vous m’avez transmis, peu importe où ma carrière me mènera. Je voudrais aussi remercier Matthew Wade, Chanel Dupont et Jannie Pedersen pour leur aide, leurs conseils, ainsi que pour les connaissances qu’ils m’ont transmises durant mon projet.
Finalement, je voudrais aussi remercier de tout mon cœur Luc-Alexandre, mon frère, ma sœur Julie-Anne, pour ses conseils en grammaire et surtout ma mère, Carole Martin ainsi que mon père, Martin Boyer, pour leur support. Un jour, des spécialistes vous ont dit que votre fils atteint de dysphasie sévère peinerait à terminer son secondaire. Vous avez toujours cru en moi et m’avez appris très jeune à y croire à mon tour. Merci pour votre amour et pour ce précieux cadeau.
1
Introduction
1-Généralités :
1.1- Les caractéristiques des virus adéno-associés
Les virus adéno-associés (AAV) sont des virus de la famille des Parvoviridae et du genre des Dependoparvovirus. Ce sont de très petites particules icosaédriques T=1 non enveloppées et ce sont aussi des Dependovirus avec un génome simple brin de 4.7 kb de longueur dont la polarité peut être négative ou positive. La caractéristique principale de ce genre est la nécessité d’un virus auxiliaire comme l’adénovirus pour se répliquer complètement. Le virus en lui-même ne cause aucune maladie chez l’homme et n’amplifie
ou n’ajoute même pas les symptômes venant des virus auxiliaires1. Aussi, la transmission
du virus est soupçonnée d’avoir lieu par voie gastro-intestinale et sexuelle29.
1.2-Historique
Le premier rapport des AAV remonte à 1965 par Robert W. Atchison, Bruce C. Casto et William McD. Hammon. Ces chercheurs, en observant des préparations d’adénovirus simiens de type 15 au microscope électronique, aperçurent de petites particules, ressemblant à des virus, en grande quantité parmi les adénovirus. Ils ont rapidement démontré que ces nouveaux virus ne se faisaient pas neutraliser par les sérums anti-adénovirus et les anticorps contre ce nouveau virus ne fonctionnent pas contre l’anti-adénovirus. De plus, il était nécessaire que les virus soient cultivés avec l’adénovirus pour se
répliquer80. Aujourd’hui, les AAV sont l’un des virus les plus étudiés de la famille des
2
2-Taxonomie des parvovirus et l’organisation génomique des
différents sous-groupes de Parvoviridae.
Parmi tous les virus pouvant infecter les cellules eucaryotes, les parvovirus figurent parmi les plus petits et les plus simples. La famille des Parvoviridae se divise en deux sous-familles : les Densovirinae, qui infectent les arthropodes et les Parvovirinae, qui infectent les vertébrés. Selon le 10e rapport du Comité international sur la taxonomie des virus (ICTV), la sous-famille des Parvovirinae est composée de huit genres : les Amdoparvovirus, Aveparvovirus, Bocaparvovirus, Copiparvovirus, Dependoparvovirus, Erythroparvovirus, Protoparvovirus et Tetraparvovirus. Pour être considéré comme un genre à part entière dans la sous-famille des Parvovirinae, un groupe de virus doit être monophylétique et le gène codant pour les protéines d’initiation de la réplication (NS1 couplé à NS2 ou Rep68 couplé à Rep78) doit montrer 30% ou moins de similitudes aux séquences d’acides aminés des espèces de virus d’un même genre. En cas de similarité de
séquence supérieure à 30%, les virus sont considérés comme du même genre.55
Grâce aux nombreuses décennies de séquençage, il est maintenant connu que les parvovirus possèdent une très grande diversité de séquences, mais aussi des motifs hautement conservés situés sur les gènes NS1 ou REP des huit genres, ce qui indique qu’ils
proviendraient d’un ancêtre commun57. Chaque parvovirus possède deux principaux cadres
de lecture ouverts (ORF), l’un pour les protéines non structurelles et l’autre pour les protéines structurelles. Certains genres ont des ORF supplémentaires codant pour une protéine additionnelle. Les parvovirus possèdent des séquences palindromiques imparfaites à leurs extrémités qui peuvent être homotélomériques ou hétérotélomériques. Les séquences homotélomériques ont pour la plupart des séquences terminales inversées (ITR) suivant une structure en tête d’épingle. Cette structure permet la réplication de l’ADN viral en fournissant un segment double brin nécessaire pour démarrer la réplication56.
Les Aveparvovirus et Copiparvovirus n’ont, à ce jour, aucune information vérifiée sur leur organisation génomique, au-delà de l’alignement avec des séquences de parvovirus dont l’organisation génomique est plus largement étudiée. En ce moment, deux nouveaux genres
3
sont proposés dans la sous-famille des Parvovirinae : Marinoparvovirus et Chapparvovirus78 et 79.
Par besoin de simplicité, seul le genre englobant les AAV sera discuté : les Dependovirus. Les Dependovirus ont un génome linéaire de 4,7 kilobases (kb) simple brin qui est encapsidé avec une quantité égale des deux polarités. La structure du génome est caractérisée par deux ITR aux extrémités de l’ADN viral flanquées de deux ORF codant pour les protéines de la réplication virale (REP) et la capside (CAP). Le génome possède trois promoteurs de transcription et un seul site de polyadénylation près de la fin de l’extrémité 3’. Les ITR ont la forme d’une tête d’épingle en « T » où les 125 premiers nucléotides forment un palindrome pour faciliter l’appariement des bases58. Le nom « Dependovirus » a été donné dû au fait qu’ils nécessitent un stimulus venant le plus souvent d’un virus auxiliaire et parfois d’un stress cellulaire103. Le sérotype le plus étudié
4
Figure 1.2 : Structure générale des six genres de la sous-famille des Parvovirinae dont
5
3- Spectre d’hôte et immunité préexistante
3.1- TropismeLes AAV possèdent un très large tropisme envers les différentes cellules du corps, dont celles du foie, du cœur, des muscles squelettiques, des yeux, des cellules nerveuses, du pancréas, des poumons, des cellules immunitaires et bien d’autres encore. Le tropisme est déterminé par le sérotype de l’AAV, par exemple AAV1 peut infecter les cellules du cœur,
mais pas celles des yeux comme AAV212. Qui plus est, il existe une grande quantité de
sérotypes des AAV pouvant infecter différentes espèces d’animaux, en commençant par l’homme, les primates non humains, les porcs, les moutons, différentes espèces d’oiseaux, les bovins, les chèvres, les espèces murines, les serpents et les lézards13-28.
6 3.2- Ligands et Récepteurs
Le virus doit commencer son cycle de réplication en se liant (en utilisant VP3, une protéine de la capside, dans le cas d’AAV2) à une lectine à la surface de la cellule, un récepteur pour les AAV (AAVR). Chez AAV2, ses VP3 se lient avec les récepteurs glycosaminoglycanes à la surface de la cellule hôte. Les AAV, pour entrer dans le trafic intracellulaire, ont besoin d’utiliser un co-récepteur. Les protéoglycanes à héparane sulfate (HSPG) sont des récepteurs primaires importants pour l’entrée et la transduction de la cellule. Des interactions secondaires avec le récepteur sont nécessaires pour l’attachement et l’entrée dans le trafic intracellulaire de la cellule hôte et donc la présence de l’un des récepteurs suivants est requise : Récepteur des facteurs de croissance des fibroblastes 1 (FGFR1), intégrines αVβ5/α5β1 et facteur de croissance des hépatocytes. Le large tropisme d’AAV2 est expliqué par la présence de glycosaminoglycanes à la surface de la plupart des
cellules adhérentes45, 46 et 47. Les AAV avec la plus basse homologie avec le VP3 d’AAV2
sont AAV4 et AAV5 qui, par conséquent, utilisent d’autres récepteurs que HSPG, qui sont respectivement l’O-acide sialique lié en α 2,3 et le N-acide sialique lié en α 2,3. Les récepteurs primaires utilisés par les autres AAV sont l’acide sialique lié en α2,3 et α2,6 pour AAV1, HSPG pour AAV3, des récepteurs d’acide sialique pour AAV4 et
5,N-galactose pour AAV9 ainsi que le récepteur laminine (LamR) pour AAV8 et AAV945, 46,
47, 50, 51, 52, 53 et 54.
La plupart des AAV utilisent comme récepteurs des lectines. Les lectines sont des protéines de liaison aux glucides servant d’outils aux cellules pour reconnaître la nature et la diversité des différentes structures glycosylées à la surface des cellules. Les lectines, qui sont utilisées par tous les organismes vivants et dont celles des plantes sont les plus étudiées, montrent une haute affinité avec les sucres48. Les interactions de la capside avec les lectines
sont relativement faibles, ce qui permet de les délier facilement et d’assurer l’affinité des lectines par rapport à la capside virale. Chaque lectine a au moins deux sites de liaison pour les molécules glycosylées.49
7 3.3- Immunité préexistante
Le virus AAV est très peu immunogène, mais à cause de l’exposition constante au virus, une grande majorité de la population possède une immunité préexistante. Lors d’une étude, 18% des tissus humains prélevés, venant d’individus différents, contenaient des séquences d’AAV. La prévalence de séquences des AAV endogènes dans une large gamme de tissus
humains suggère que l’infection naturelle est très commune pour ce groupe de virus29. À
force d’exposition, les AAV peuvent finir par être détectés par un récepteur de type Toll 2 (TLR2) ou un récepteur de type Toll 9 (TLR9) ou encore après la transduction. Les peptides de la capside vont être ubiquitinés et dégradés par les protéasomes avant d’être présentés par le complexe majeur d’histocompatibilité (CMH) de classe I, ce qui aura pour résultat de détruire les cellules transduites par l’intermédiaire des lymphocytes cytotoxiques T30. Le système immunitaire inné va rapidement mettre en branle l’immunité acquise afin de produire des anticorps contre le virus. Une étude démontre que les populations venant des États-Unis et de l’Europe de l’Est possédaient des anticorps neutralisants (Nabs) chez 54% des individus pour AAV2, 28% pour AAV5 et 38% pour AAV8. Les anticorps ne sont pas spécifiques à un sérotype d’AAV non plus, car seulement 33 % de ceux qui avaient des Nabs visaient spécifiquement un sérotype : 29% visaient uniquement les AAV2, 19%
visaient à la fois AAV2 et AAV8, 3% AAV2 et AAV5 et 47% visaient les trois AAV31. La
réponse humorale contre les AAV à l’état naturel (WT) mène à la production de toutes les sous-catégories d’anticorps d’immunoglobuline G (IgG) où l’immunoglobuline G1 (IgG1) est prédominante. La réponse peut varier selon le sérotype. Par exemple si le niveau d’immunoglobuline G2 (IgG2) est plus faible pour les réponses humorales anti-AAV spécifiques, elle sera plus forte contre AAV844.
8
4- Caractéristiques du virion
4.1- Les propriétés du virion
L’une des familles qui compte en son sein les plus petits virus sont les parvovirus. Les virions des AAV ont l’apparence d’une particule parasphérique sans enveloppe et mesurent 26 nm.
Les virus adéno-associés sont constitués essentiellement d’ADN et de protéines dans des proportions de ¼ et ¾ de leur masse85. La capside est composée de trois polypeptides
principaux : VP1 (87 kDa), VP2 (73 kDa) et VP3 (61 kDa)90.
La première structure de capside des AAV qui a été observée est celle du sérotype 2 en 2002 à l’aide de la cristallographie aux rayons X avec une résolution de 3-Å. La capside est composée de 60 sous-unités monomériques où chaque sous-unité est faite avec une protéine VP1, une VP2 et huit VP3. Les sous-unités sont disposées en pentamère aux 12 sommets de l’icosaèdre86 et 87.
4.2- Propriétés biochimiques
Les AAV et parvovirus sont très stables en général. Les virions de parvovirus tolèrent l’exposition à un pH entre 3 et 9 et ils ne sont pas sensibles à des solvants organiques comme le chloroforme ou la Dnase, car la capside ne possède pas de lipides ou d’hydrates de carbone. Par contre, les virions sont sensibles au formol, à la bêta-propriolactone, à l’hydroxylamine et aux agents oxydants. Les virions des AAV peuvent résister à une température de 70 degrés Celsius durant 15 minutes.81, 82 et 83.
4.3- Propriétés antigéniques
Les sérums d’anticorps contre un sérotype d’AAV peuvent fonctionner contre d’autres sérotypes d’AAV. Par exemple, les Nabs contre AAV2 ont tendance à fonctionner aussi
9
5- Biologie moléculaire des AAV
5.1- Nature du génomePour faire un rappel, l’AAV possède un génome d’ADN linéaire simple brin de 4,7 kb qui représente le quart de la masse du virion. Chaque brin d’ADN est flanqué d’une ITR de chaque côté ayant une tête d’épingle en forme de « T » de 145 paires de bases (pb) se terminant avec 125 pb, identiques des deux côtés, formant ainsi une partie double brin pour protéger l’ADN de certaines Dnases. Le génome viral peut être encapsidé aussi bien dans sa polarité positive que dans sa polarité négative et à la même fréquence.
5.2- Transcription des gènes du virus associé
Ce virus n’est pas capable de se répliquer tout seul sans avoir une aide extérieure qui vient, le plus souvent, sous la forme d’un virus auxiliaire. Il a été rapporté que les AAV pouvaient être aidés par les adénovirus, les virus de l’herpès simplex (HSV), le virus de la varicelle et le virus du papillome humain. Seuls les adénovirus et les HSV, les plus étudiés, seront discutés103.
Chez les adénovirus, il y a cinq gènes qui aident les AAV. Les protéines E1A et E2A jouent un rôle dans l’expression du génome en activant le site d’expression p5 qui se réprime lui-même en absence d’aide. E2A est aussi impliqué dans l’amélioration de la traduction de l’ARN viral et augmente la réplication du génome de l’AAV avec E4. Aussi, E4, en combinaison avec E1B, joue un rôle essentiel dans le transport et l’accumulation d’ARN viral dans le cytoplasme. Finalement, les ARN nommés VAI sont des augmentateurs de traduction par excellence pour les AAV. Il est aussi suggéré que l’adénovirus aide aussi au transport du virus vers le noyau et à l’épissage de l’ARN viral. Les gènes E2A, E4 et VAI se révèlent être plus que nécessaires lors de la production de AAV recombinants103.
Chez les HSV, il y a aussi plusieurs gènes susceptibles d’aider les AAV. Le gène UL29, qui produit la protéine ICP8, et le virus HSV sont nécessaires à la réplication virale. HSV
10
peut aussi interagir avec le REP des AAV pour aider à couper les ITR. Aussi, les gènes UL5, UL8 et UL52 (ils forment le complexe primase-hélicase) permettent à ICP8 de s’attacher à la bonne position sous-nucléique103.
Néanmoins, il existe des études qui affirment que les AAV peuvent se répliquer seuls si la cellule infectée est soumise à un stress génotoxique104.
5.3- Transcription et traduction des gènes des AAV
Les dependoparvovirus renferment des parvovirus avec un génome monosens dont la taille fait 4,7kb et les deux sens sont compactés dans les capsides dont l’expression des gènes suit trois modèles différents. Les trois modèles utilisent l’ADN double-brin pour la transcription.
Le premier modèle, concernant l’espèce dependoparvovirus adéno-associé A dont fait
partie AAV2, utilise deux cadres de lecture ouverts, trois promoteurs de transcription, qui sont p5, p19, p40, et un seul site de polyadénylation près de la fin de l’extrémité 3’. Le site p5 produit un ARN messager qui code pour Rep78 (ARNm non épissé) et Rep68 (ARNm épissé). Comme pour p5, le site p19 produit un ARNm qui code pour Rep52 (ARNm non épissé) et Rep40 (ARNm épissé). Le site p40 transcrit un ARNm ayant un premier site accepteur d’initiation de traduction qui produit VP1 en utilisant tout le gène, sans épissage. Pour VP2 et VP3, l’ARN est épissé et VP2 utilise le codon de démarrage alternatif ACG alors que VP3 est généré plus loin avec le codon traditionnel AUG58.
Le deuxième modèle concerne AAV5 et autres AAV non humains, pour lesquels le site
p7 (l’équivalent de p5) produit seulement Rep78 et Rep52 et Rep40 ne viennent pas de différents ARN épissés, mais de sites d’initiation de traduction différents59, 60 et 61.
Le troisième modèle et le plus rare, concernant les dependoparvovirus ansériformes 1 comme le parvovirus d’oie, présente une stratégie d’expression similaire au protoparvovirus. Ils ont tous les deux uniquement deux promoteurs (p9 et p42) où p42
11
fonctionne comme les autres modèles et p9 synthétise Rep1 ou Rep2 selon le traitement nucléique qu’aura reçu le pré-ARN p962 et 63.
Aussi, ce genre synthétise une autre protéine, la protéine activatrice d’assemblage (AAP), qui n’est pas présente dans les autres genres et qui est traduite dans un site d’amorçage non conventionnel (CTG) dans un ORF alternative du gène CAP42.
Figure 5 : Représentation schématique du génome des AAV.
Ce diagramme illustre l’organisation du génome des AAV. Il est composé de deux ORF majeurs qui codent pour les gènes CAP et REP qui sont flanqués par les ITR. Différents transcrits de REP et de CAP ont été produits par leurs promoteurs respectifs de fait que p5, p19 et p40, VP2 et VP3 viennent du même transcrit, mais ont des codons de démarrage
12
différents (ACG pour VP2 et AUG pour VP3). Le gène AAP utilise un ORF alternatif pour transduire.
5.4- Réplication des AAV
La réplication de l’ADN viral se produit dans le noyau et les ITR des AAV agissent en cis (et sont les seuls à agir en tant que cis élément dans la réplication) comme amorce pour l’ADN polymérase afin de synthétiser le second brin d’ADN. L’ADN double-brin qui a été produit sera appelé le monomère à forme réplicative (RFM). Lorsque le RFM est utilisé pour un second tour d’autoréplication à partir d’amorces, cela forme un dimère à forme réplicative (RFD). Le stade intermédiaire de RFM et RFD est répliqué par un mécanisme
de déplacement d’ADN simple-brin105. Rep68 et Rep78 vont ouvrir et couper les terminis
et réinitialiser la réplication2 et 5. Les protéines nécessaires en trans à la réplication virale in vitro sont les protéines Rep78 et Rep68, l’ADN polymérase ᵟ, les facteurs de réplication C, l’antigène de prolifération nucléaire cellulaire et le complexe de maintenance du minichromosome. Pour pouvoir se répliquer, le virus doit recevoir un stimulus venant soit d’un virus auxiliaire ou venant d’un stress cellulaire, car le virus n’est pas capable d’induire la phase S du cycle cellulaire, ce qui est nécessaire à sa réplication106. Dans le cas contraire, le virus reste latent jusqu’au moment où un virus auxiliaire infecte la cellule, Il existe deux écoles de pensée pour expliquer ce qui se produit dans ce cas. La première, et la plus populaire, stipule que le génome de l’AAV s’intègre dans le chromosome 19 au site AAVS1 où il attendrait en latence l’infection par un virus auxiliaire10. La seconde théorie
13
6- Produits du génome
6.1- Protéines structurales
Le génome des AAV possède deux gènes : REP et CAP. Le gène CAP est responsable de la génération des protéines de la capside. Comme la plupart des parvovirus, l’AAV possède une masse moléculaire d’ensembles de protéines structurales qui excède grandement la capacité codante du génome. Faisant partie des Parvovirinae, la capside est composée uniquement de trois protéines contrairement aux Densovirinae qui ont des capsides composées de quatre protéines. Néanmoins, les propriétés biophysiques et biochimiques
sont les mêmes pour les deux sous-familles89. La capside de l’AAV se compose de trois
polypeptides principaux appelés VP1, VP2 et VP3 qui ont une masse moléculaire respective de 87 kDa, 73 kDa et 61 kDa. Les trois protéines sont dans une proportion molaire de 1 VP1 :1 VP2 :10 VP3 et font partie de 75% de la masse du virion85 et 86. Aucune modification post-transcriptionnelle ou aucun ajout de résidu glucidique attaché la capside n’ont été observés. D’autres fonctions ont été observées avec ces protéines structurales. VP1 possède un domaine phospholipase A2 (PLA2) qui est utilisé pour sortir du compartiment endosomal91. De plus, VP1 et VP2 possèdent des signaux de localisation
nucléique (NLS) sur leurs N-terminus pour entrer dans le noyau92.
6.2- Protéines non structurales
Le gène REP permet de synthétiser quatre protéines : Rep78 et Rep68 qui sont impliquées
dans la réplication du génomeet Rep52 et Rep40 qui sont utilisés pour le compactage du
génome. Ces protéines fonctionnent en paires et elles ont toutes des activités hélicase et ATPase. Les protéines Rep78 et Rep68 s’attachent aux ITR du génome viral. Ces dernières vont défaire la conformation double brin des ITR avec leur activité hélicase et couper le termini avec une activité endonucléase à site spécifique (ATPase), ce qui est nécessaire pour la réplication du génome ou l’intégration du virus dans le génome2 et 5. Rep52 et Rep40 s’attachent aux ITR du génome viral et avec leurs activités hélicases, elles conduisent la translocation de tout le génome dans la précapside où le sens 3’ à 5’ est préféré3 et 4.
14 6.3 Protéines auxiliaires
Le gène CAP permet de synthétiser deux protéines non structurales qui sont AAP (protéine activatrice d’assemblage) et la protéine X. AAP permet de transporter les protéines VP3, qui se trouvent dans le cytoplasme, vers le nucléole, où se trouve VP1 et VP2, mais AAP joue aussi un rôle dans l’assemblage de la capside, cependant le débat fait encore rage quant à savoir si c’est une protéine d’échafaudage et/ou chaperonne42. Une étude a
démontré que la protéine est nécessaire à la stabilité de la capside virale88. La protéine X a
pour rôle d’amplifier le niveau de réplication du génome viral43. Les deux protéines
accessoires ont été découvertes récemment et il n’y a pas encore beaucoup d’informations à leur sujet, en particulier la protéine X.
15
7- Morphologie et structure
7.1- En général
Les capsides virales, dont l’utilité primaire est de protéger le génome contre l’environnement, sont de véritables bijoux d’architecture à l’échelle moléculaire. La structure de la capside est formée de copies identiques de quelques protéines virales, ce qui permet d’économiser au maximum l’information génétique. Les molécules de protéine sont arrangées pour avoir un maximum de contact avec le milieu tout en maintenant la capside de manière stable avec uniquement des interactions protéine-protéine. Pour avoir une capside symétrique tout en possédant le plus grand volume interne possible avec des protéines non symétriques, le moyen le plus économique pour construire une telle capside est d’arranger les protéines en des triangles équilatéraux qui vont former un icosaèdre. Un icosaèdre est une structure géométrique de 20 triangles équilatéraux arrangés autour de la face d’une sphère. Chaque triangle équilatéral est l’assemblage de trois sous-unités, ce qui porte le nombre de sous-unités de protéine dans la capside au nombre de 60. La symétrie icosaèdre permet d’obtenir une capside fermée avec un nombre minimal de sous-unités identiques. Chacune des sous-unités est reliée les unes aux autres par les axes de rotation 2, 3 et 5 qui définissent la symétrie icosaèdre. Il y a 15 axes d’ordre 2 qui passent au travers des arêtes et 10 axes d’ordre 3 s’allongeant au travers de chaque face de la capside. Cinq sous-unités font un contact d’ordre 5 pour obtenir un axe de symétrie d’ordre 5 aux 12 sommets. L’axe de symétrie 2 permet à un objet d’avoir une apparence identique à son point de départ après une rotation de 180 degrés. À l’axe 3, l’apparence de départ doit être restaurée avec les rotations de 120 degrés et 240 degrés et à l’axe 5, l’identité est restaurée avec des rotations de 72 degrés, 144 degrés, 216 degrés et 288 degrés. Une des caractéristiques d’une structure icosaédrique est que les interactions de toutes les molécules avec leurs voisines sont identiques93.
Chez les AAV, les protéines de la capside VP1, VP2 et VP3 sont présentes dans le virion dans un rapport approximatif de 1 :1 :10 pour un total de 60 sous-unités par particule. Les protéines VP1, VP2 et VP3 contiennent respectivement 735, 598 et 533 acides aminés pour AAV2 et ils ont tous la même extrémité C-terminale, ce qui constitue environ 520 acides
16
aminés. La capside de l’AAV de forme icosaédrique est composée en grande partie des C-termini des VP. Le cœur des protéines VP est constitué de huit tonneaux bêta antiparallèles et d’une hélice alpha86.
La première structure de capside de l’AAV qui a été observée est le sérotype 2 en 2002 à l’aide de la cristallographie aux rayons X avec une résolution de 3-Å, ce qui est la moyenne pour les icosaèdres. La structure tertiaire consiste en un cœur de feuillet bêta antiparallèle et d’hélices alpha, similaire aux autres parvovirus reliés entre eux par de longues boucles. Les brins bêta sont reliés par de longues boucles et ces boucles sont responsables des contacts entre les sous-unités et des caractéristiques à la surface des capsides86.
7.2 Les boucles chez les AAV
Les tonneaux bêta sont répartis dans deux feuillets appelés ‘BIDG’ et ‘CHEF’ qui sont composés des brins antiparallèles bêta-B, bêta-I, bêta-D et bêta-G pour le feuillet BIDG et bêta-C, bêta-H, bêta-E et bêta-F pour le feuillet CHEF. Les feuillets sont reliés entre différentes parties de larges boucles qui peuvent aussi contenir des petits brins de feuillet bêta qui forment 70% de la topologie de la protéine VP3. Une grande partie de ces boucles sont localisées à la surface de la capside. Les AAV possèdent trois boucles majeures, la boucle GH qui relie les bêta-G et bêta-H, la boucle HI qui relie les bêta-H et les bêta-I et la boucle DE qui relie les bêta D et les bêta-E qui comprend respectivement 230, 14 et 16 acides aminés86 et 94.
7.3 Les interactions des sous-unités.
Les 60 sous-unités de la capside virale peuvent interagir via les axes de symétrie d’ordre 2, 3 et 5, car elles sont présentées de manière uniforme tout le tour de la capside grâce à sa symétrie icosaédrique86.
17
L’extrémité C-terminale contenue dans bêta-I relie les VP monomères voisins pour former la majorité des interactions d’axes d’ordre 2. Ces extrémités se situent entre les dépressions et sont entourées des axes d’ordre 5 icosaédriques. L’alpha-A est aussi impliqué dans les axes d’ordre 2 et forme un mur de dépressions. Ces interactions ont la plus petite surface enterrée des trois axes, car il y a seulement une chaîne de polypeptides impliquée. Les interactions d’axes d’ordre 3 sont les plus étendues et impliquent la boucle majeure GH et les mini-boucles BC et EF. Les boucles GH s’interdigitent pour former des saillies en forme de pointes venant de l’axe d’ordre 3. Les AAV 1, 2, 3, 6, 7, 8 ont des saillies ressemblant à des doigts et les AAV 4, 5 et 9 ont des saillies plus rondes86.
Finalement, la plupart des interactions d’axe d’ordre 5 du monomère VP3 se font avec la boucle HI. La boucle est positionnée au-dessus des feuillets bêta des VP3 monomères et forme le plancher de la dépression conservée qui entoure le canal à l'axe. Le reste des interactions sont formées par le feuillet BIDG et les N-terminaux dans les monomères adjacents. Le canal cylindrique de la boucle DE sur l’axe d’ordre 5 va créer une tourelle à la surface à partir du feuillet bêta. Cette tourelle connecte l’intérieur et l’extérieur de la capside. La tourelle sert à exposer à l’extérieur la partie N-terminale de VP1 qui contient un domaine phospholipase A2 (PLA2) et le signal de localisation nucléaire (NLS) nécessaire à l’infectiosité86 et 95.
18
8- Trafic intracellulaire des AAV et leur entrée dans le noyau.
Le virus se réplique dans le noyau, de ce fait, il doit passer la membrane cellulaire et la membrane nucléaire. Les AAV passent la membrane cellulaire via des endosomes, mais comme ils sont non enveloppés, ils doivent utiliser une méthode alternative à la fusion de la membrane virale avec l’endosome. Le mécanisme d’évasion de l’endosome possède encore aujourd’hui de grandes zones d’ombres. La décapsidation du virion semble liée à l’acidification de l’endosome et les protéases d’endosome cathepsine B et L, mais le mécanisme est encore inconnu96 et 97. Pour briser l’endosome, le virus semble utiliser la voie enzymatique pour sortir de l’endosome et gagner le cytoplasme. VP1 possède à son N-terminus un domaine phospholipase A2 (PLA2) qui agit comme lipase et est nécessaire à l’infection et à la libération de la capside. Le domaine PLA2 reste à l’intérieur de la capside jusqu’aux compartiments de l’endosome tardif ce qui permet à la capside d’être relâchée en périphérie du noyau. Les mécanismes d’activation de PLA2 restent encore incompris. Dans le cytoplasme, d’autres évènements de trafic, de changement de conformation et d’organisation moléculaire se produisent avant d’atteindre le noyau et demeurent incertains encore86 et 91.
Les NLS se trouvent dans la partie N-terminale de VP1 et VP2 et ils sont exposés dans les endosomes tardifs. Le génome viral doit être dans le noyau pour se répliquer et pour y parvenir, il doit utiliser une voie d’import nucléique. L’implication des NLS qui se trouvent dans les VP1 et VP2 des AAV est nécessaire à l’import nucléique92. Il existe plusieurs
hypothèses quant à l’entrée du virus dans le noyau. La première hypothèse est que le virus forme un complexe de pore nucléique (NPC), mais n’a pas encore été observée chez les AAV, alors qu’elle l’a été chez d’autres parvovirus. Par exemple, le virus minute de la souris (MVM) perturbe la membrane nucléique dans les cellules de fibroblastes de souris et crée de petits trous où le virus passe98. La seconde hypothèse est que la petite taille de la capside lui permet de passer au travers des pores nucléiques avec la capside intacte. Cette hypothèse est soutenue par l’évidence que, lors d’une expérience sur AAV2, la cellule infectée s’est vue micro-injectée des anticorps spécifiques aux capsides d’AAV dans le
19
noyau et AAV2 a été neutralisé99. Que ce soit de l’ADN viral nu ou le virion intact de
l’AAV, le noyau est la destination finale du virus.
9-Les différentes méthodes utilisées pour isoler les AAV jusqu’à
aujourd’hui.
9.1- Les rongeurs
Les séquences d’AAV de rat et de souris ont été obtenues à partir d’ADN venant du foie de la souris et du rat qui a été purifié avec le DNeasy tissue kit (QIAGEN). La première opération était une PCR pour amplifier un fragment d’ADN de 1,5 kb ayant les 800 pb du C-terminus du gène REP et les 700 pb du N-terminus du gène CAP. Ensuite, trois arpentages chromosomiques ont été effectués afin d’obtenir des fragments d’environ 1400 pb, 550-400 pb et 360-300pb. Un fragment d’environ 3000 pb avec tout le gène CAP et la plupart de rep a été obtenu26.
9.2- Le Porc
Ces AAV ont été découverts par le laboratoire de mon directeur de recherche Dr Gary Kobinger et voici la méthode utilisée. Les séquences d’AAV venant du porc ont été obtenues à partir d’ADN venant de divers tissus de porc qui ont été purifiés avec le QIAamp DNA mini kit (QIAGEN). La première opération était une PCR emboîtée en deux étapes où dans la seconde PCR, les amorces sont à l’intérieur de la séquence amplifiée, pour amplifier un fragment d’ADN de 1,7 kb ayant les 900 pb du C-terminus du gène REP et les 800 pb du N-terminus du gène CAP. Ensuite, une PCR emboîtée en trois étapes thermoasymétriques entrelacées (TAIL) a été effectuée pour obtenir soit la fin de CAP de l’AAV ou le CAP au complet. Le gène CAP a été obtenu complètement pour tous13 sauf le
sérotype po1 qui a aussi la fin du gène REP32. Aussi, plusieurs de ces AAV partagent des similarités avec AAV5 et AAV2.
20
Pour l’AAV de serpent, les cellules venant d’un python malade ont d’abord été cultivées jusqu’à la lyse cellulaire, cette dernière a été centrifugée pour séparer les particules virales et l’ADN viral a été isolé via SDS/protéinase K. L’ADN a d’abord été digéré à l’aide de plusieurs enzymes de restriction, puis une PCR a été performée et l’ADN a encore été traité avec différentes enzymes de restrictions. L’AAV a ainsi été séquencé avec succès.
9.4- Les oiseaux
Pour les AAV d’oiseaux, le virus a d’abord été cultivé dans des cellules d’embryon de poulet avec un adénovirus aviaire. Ils ont été ensuite centrifugés pour isoler les particules virales et l’ADN viral a été isolé via SDS/protéinase K. L’ADN a été migré sur gel d’agarose 1% où une bande de 4,7 kb a été extraite et le second brin a été synthétisé à l’aide d’une polymérase ADN. L’AAV été séquencé. 15.
9.5- Les Chèvres
Pour les AAV de chèvres, les AAV ont été cultivés sur des cellules de chèvres avec des adénovirus. Quatre PCR ont été effectuées pour avoir la séquence de CAP, de REP, du côté droit des ITR et du côté gauche des ITR. Cet AAV a été séquencé avec succès. 27 et 33.
9.6- Le bœuf
Pour les AAV de bœuf, deux méthodes différentes ont été utilisées. Les AAV ont été cultivés sur des cellules de rein de bœuf avec des adénovirus. La première méthode était une PCR pour amplifier un fragment d’ADN de 1,4 kb ayant les 700 pb du C-terminus du gène REP et les 700 pb du N-terminus du gène CAP. La seconde PCR a été faite des amorces dégénérées situées au début du gène REP et à la fin du gène CAP et la majorité de la dégénérescence se trouvait du côté amplifié. Pour l’autre méthode, impliquant des poumons de bœuf ayant des maladies respiratoires, une métagénomique virale a été effectuée pour y détecter différents virus, dont des AAV. La première méthode a permis d’obtenir toute la séquence de l’AAV, sauf les ITR.34 La seconde a donné presque le même
21 9.7- Les primates non humains
Pour les AAV de singes, plusieurs méthodes ont été utilisées pour les trouver. Pour les AAV 10 et 11, l’ADN a été extrait du singe crabier avec le kit d’extraction d’ADN
QIAamp. Une région signature de 250 bp a été amplifiée vers le début de CAP pour
indiquer la présence d’AAV et deux fragments d’ADN ont été amplifiés partant des deux extrémités de l’AAV (et en amplifiant le milieu pour AAV10), donnant des fragments assez longs pour qu’ils se chevauchent24. Les AAV 7, 8, pi.1 et 9 (humain), ont été détectés de la même manière que 10 et 11. Pour le gène CAP, deux PCR ont été faits avec de chaque côté de la région signature avec cette même région en commun la même chose a été fait avec le gène REP. La séquence a été reconstruite à partir des plasmides créés à partir des PCR. Le résultat a été confirmé avec l’aide d’une PCR commençant au milieu de REP jusqu’à la fin de CAP21 et 23. Pour AAV12, les cellules infectées ont été lysées avec le kit
d’ADN QIAamp et deux PCR amplifiant respectivement tout le gène REP et le gène CAP ont été effectuées36. Pour AAV1, le virus a été cultivé dans des cellules de singe 293 avec
adénovirus. Une enzyme a ensuite été utilisée pour réparer l’ADN et pour couper les extrémités, suivi d’une autre enzyme pour couper le virus en trois fragments qui ont été insérés dans trois plasmides différents. Les trois plasmides ont été séquencés38. Pour
AAV4, le virus a été cultivé dans des cellules humaines HeLa avec adénovirus. Il a été purifié avec le CsCl et un transfert de protéines hybridées avec une sonde contenant AAV2 a été effectué. Le fragment capturé a été mis dans un plasmide et a été envoyé au séquençage39. AAV 7, 8, 10, 11, 12, pi.1 ont tout le gène REP et CAP. AAV 1 et 4 a tout le gène REP et CAP et les ITR.
22
Pour les AAV humains, le premier à avoir été séquencé est AAV2 qui a été cultivé dans des cellules humaines HeLa avec adénovirus. Il a été purifié avec du CsCl après avoir été lysé par trypsine et deoxycholate et les virions ont été marqués avec de la [3H]thymidine. L’ADN a ensuite été digéré par diverses enzymes de restriction et extrait sur un gel de polyacrylamide37. Pour AAV3, il a d’abord été cultivé dans des cellules humaines KB avec adénovirus. L’ADN a été digéré par une paire d’enzymes de restriction avant d’être inséré dans des plasmides. Les plasmides ont ensuite été soumis à une hybridation de type Southern avec des sondes AAV2 marquées sur les atomes phosphore pour finalement être séquencés. Afin de séquencer les ITR, l’ADN a été digéré avec une enzyme de restriction appropriée selon les résultats du premier séquençage. Ils ont été migrés sur gel agarose et les bandes appropriées ont été excisées et l’ADN a été isolé par gel extraction. Les fragments d’ADN ont ensuite été introduits dans des plasmides et ont été séquencés40. Pour
AAV5, l’ADN a été extrait avec la protéinase K. Le génome a été digéré par des enzymes de restriction visant des sites de restriction uniques et l’ITR droit a été coupé par une polymérase et a été digéré par une autre enzyme. Les ITR ont ensuite été clonés dans des plasmides avant d’être séquencés41. Pour AAV6, il a été cultivé sur des cellules 293 avec
adénovirus avant d’être lysé par protéinase K, extrait par phénol-chloroforme et précipité par acétate de sodium. L’ADN de AAV6 a été laissé hybrider pendant plusieurs heures avec son génome complémentaire. Les fragments de 5kb ont été extraits sur gel, leurs extrémités ont été remplies par une polymérase et par des enzymes XbaI avant d’être mis dans un plasmide. Ils ont été transférés par Southern avec une sonde AAV6 et ils ont été ensuite séquencés20. Le génome a été complètement séquencé avec les ITR pour tous les AAV humains.
23
10- Raisons pour rechercher des AAV dans les tissus de porcs.
Le porc a été choisi comme source potentielle d’AAV pouvant être utile à la thérapie génique pour plusieurs raisons.
10.1- Similarité entre l’humain et le porc
La première raison de favoriser le porc est la grande similarité entre l’homme et le porc au niveau anatomique. Tout d’abord, les cochons possèdent, en grande partie, les mêmes muscles que l’humain, tels que les muscles de la poitrine majeure et de l’abdomen. Il y a néanmoins quelques différences, dues au fait que les cochons sont quadrupèdes et les humains sont bipèdes et qui affectent l’emplacement des muscles de la poitrine qui se fixent à la ceinture scapulaire. Le cochon et l’homme possèdent aussi les mêmes organes, dans les mêmes positions et ils ont environ la même taille, mais il existe toute même quelques petites disparités telles que la présence d’un lobe de foie supplémentaire chez le porc. Au niveau du système sanguin, il y a seulement deux différences notables : la présence d’un tronc bicarotide et l’absence d’artères iliaques chez le porc. Des études ont démontré que les tissus humains et porcins sont assez proches pour permettre un certain niveau de
xénogreffe cochon-homme, par exemple la greffe de valve cardiaque de cochon110. En plus
qu’il soit, comme l’homme, un mammifère, ce qui signifie une similarité raisonnable au niveau génétique, le porc possède également une physiologie du corps similaire, ce qui implique que les AAV devraient présenter une bio distribution similaire100. Aussi, ils sont assez dissimilaires pour éviter une réaction croisée comme avec les AAV venant de singes qui sont reconnus par la pré-immunité par réaction croisée.119
10.2 Le porc sert d’hôte intermédiaire pour plusieurs virus
Deuxièmement, le porc sert fréquemment d’hôte intermédiaire pour plusieurs virus pouvant infecter l’homme et les AAV porcins pourraient en faire partie. Le premier exemple prouvé est celui de l’influenza, un virus à ARN segmenté, qui utilise le porc
24
comme hôte. Cet hôte peut aussi bien être infecté par l’influenza humaine que l’influenza provenant d’autres espèces comme les oiseaux, ce qui rend possible la recombinaison entre
différents virus résultant en de nouvelles souches pouvant être transmises à l’homme101.
Un autre exemple est l’hépatite E, pour laquelle le porc agit comme réservoir naturel pour
ce virus alors que de multiples cas de transmission cochon-homme ont été rapportés102.
Bien que le risque de transmission du porc à l’humain soit présent, les AAV porcins ne se retrouvent pas au sein de la population en général. Il est possible d’en déduire deux hypothèses : qu’ils sont incapables d’infecter l’homme, ce qui est peu probable, car ils sont capables d’infecter des souris dans des situation expérimentales ou que les AAV porcins ne sont pas capables de transmission d’homme à homme. Alors, la transmission se limiterait aux personnes habitant en campagne. Donc, il y a peu de risques d’une présence de pré-immunité au sein de la population.
25
Chapitre 1 : Objectifs et hypothèses
Les vecteurs dérivés des AAV peuvent délivrer des transgènes dans une variété de tissus in vivo, ce qui leur confère un grand potentiel pour la thérapie génique. Il existe quatre raisons qui font des AAV l’un des meilleurs vecteurs de thérapie génique : l’absence de pathogénicité, la faible immunogénicité, l’expression dans les cellules mitotiques et post-mitotiques et la spécificité tissulaire des sérotypes. Le vecteur AAV est à présent utilisé dans deux traitements de thérapie génique : le Luxturna pour traiter la neuropathie optique héréditaire de Leber de type 2 (produit en 2018)107 et le Zolgensma pour traiter l’atrophie
musculaire spinale (produit en 2019)108. Des vecteurs AAV sont également étudiés en
essais cliniques pour leur potentiel thérapeutique sur de multiples autres maladies génétiques. Des études sur l’usage de AAV comme vecteurs de vaccins similaires au VSV pour le vaccin contre l’Ebola sont en cours13 et 120. La majorité des vecteurs d’AAV
recombinants (rAAV) sont basés sur l’AAV2 humain, qui est le plus fortement caractérisé et contre lequel les populations des États-Unis et de l’Europe de l’Est possèdent des anticorps neutralisants dans une proportion de 54%31. Il est d’une grande importance d’obtenir un vecteur d’AAV de porc aussi performant que ceux humains, mais qui n’est pas reconnu par les anticorps de la population afin qu’il ne soit pas neutralisé avant de délivrer ses effets thérapeutiques. Il est déjà établi que les AAV du porc ne sont pas reconnus par les anticorps de la population et ils pourraient être aussi performants que les
AAV humains13.
Le tropisme cellulaire est principalement déterminé par les protéines de la capside encodées par le gène CAP et la capside est une cible parfaite pour la réponse immunitaire. Les meilleurs candidats à la thérapie génique sont les virus peu immunogéniques comme certains sérotypes d’AAV, ce qui fait d’eux de mauvais vecteurs de vaccination. L’inverse est également vrai des virus fortement immunogéniques. Les premiers AAV ont été découverts dans des réserves d’adénovirus contaminés ou provenant d’enfants ou animaux malades80, 40, 24, 22. Plus récemment, les nouveaux AAV ont été découverts via des PCR
visant à amplifier l’ADN génomique de différentes espèces animales26, 32 et 33. Il existe une
26
digestion à la Dnase dépendant de l’ATP sûre préserverait les plasmides tout en éliminant le génome de l’hôte. Cette hypothèse pourrait faciliter sa détection11.
Notre laboratoire a isolé de nouvelles séquences d’AAV venant de tissus et de selles de porc. Les nouvelles séquences ont été trouvées par PCR emboîtée et des vecteurs basés sur ces séquences ont été produits et caractérisés in vitro et in vivo.
Nous voulons tester l’hypothèse de la présence, au Québec, de souches d’AAV porcins produisant des capsides différentes de celles mentionnées ci-dessus. À partir d’échantillons de tissus frais et de purin, ce projet vise à identifier et à tester de nouvelles capsides d’AAV porcins.
Les objectifs de ces études constituant ce mémoire sont de :
1- Trouver de nouvelles séquences du gène CAP d’AAV venant de tissus ou de fèces de porc.
2- Évaluer l’efficacité de la digestion de Dnase dépendant de l’ATP sûre pour les plasmides sur les résultats PCR
3- Produire des AAV recombinés avec les nouveaux gènes CAP et le transgène GFP ou LacZ
Aux nouvelles constructions d’AAV porcins s’est ajouté AAV8 avec l’expression du transgène LacZ qui sera utilisé pour les tests de transduction.
27
Chapitre 2 : Matériel et méthodes
1-Isolation de l’ADN génomique des fèces de porc, détection des
AAV et amplification du gène CAP
Les différents échantillons de fèces de porc ont été criblés pour détecter la présence de séquences d’AAV en collaboration avec Monsieur Claude Lamarre. Les échantillons de porc ont été obtenus par Jean-François Rivest le 28 février 2018 à la ferme Les Élevages Bonneau, à Saint-François-de-la-Rivière-du-Sud. Les échantillons de cochon ont été réduits en poudre à l’aide d’azote liquide et d’un pilon et mortier, les cellules ont été lysées par du SDS 1% à 95 degrés Celsius durant 10 minutes, l’ADN a été isolé à l’aide de multiples traitements au phénol et chloroforme, le produit a été ensuite digéré par la RNAse A à 37 degrés Celsius durant 30 minutes avant de faire un dernier traitement au phénol et chloroforme.
Étant donné la faible quantité d’ADN viral présent dans les échantillons et la grande quan-tité d’ADN génomique du porc et de ses bactéries intestinales qui génèrent de l’interférence, les PCR doivent être, au minimum, semi-emboîtées pour pouvoir isoler l’ADN des AAV, car ce type de PCR réduit considérablement les interférences. Une première PCR a été effectuée avec les amorces sens UniversalFWD (5’-CGTGGTCTT-GTACTGCCTGGATATAATATCTGGGACCC-3’) ou UniversalFWDcap.1 (5’-GGCG-CCGACGGAGTGGGTAATGCCTCGGGAAATTGGCATTGCGATTCC3') combinées avec les amorces anti-sens RCAP3(RCAP3.1 : 5’-TTACAGATTACGAGTAAGGTAGC-3’, RCAP3.2 : 5’-TTACAGATTACGAGTAAGGTAAC-5’-TTACAGATTACGAGTAAGGTAGC-3’, RCAP3.3 : 5’-TTACAGAT-TACGAGTAAGGTATC-3’, RCAP3.4 : 5’-TTACAGGGGACGAGTAAGGTAAC-3’ et RCAP3.5 : 5’-TCACAAGTTTCGTGTGAGATACC-3’) et CED-H (5’-ACTGAMACG-AATAMMCGGTTTATTGA-3’) ou CED+H (5’-ACTGAMACGAATHAMMCGGTTT-ATTGA-3’). Pour détecter la présence des AAV, une deuxième PCR emboîtée a été effectuée avec les amorces FcapA(5’-GGTAATGCCTCGGGAAATTGGCATT-3’) et Rcap+ (5’-TGTTGTTGATGAGCCGCTGCCAGT-3’) qui ont la capacité d’amplifier une région signature d’environ 200 pb pour identifier la présence d’AAV21. À partir des
28
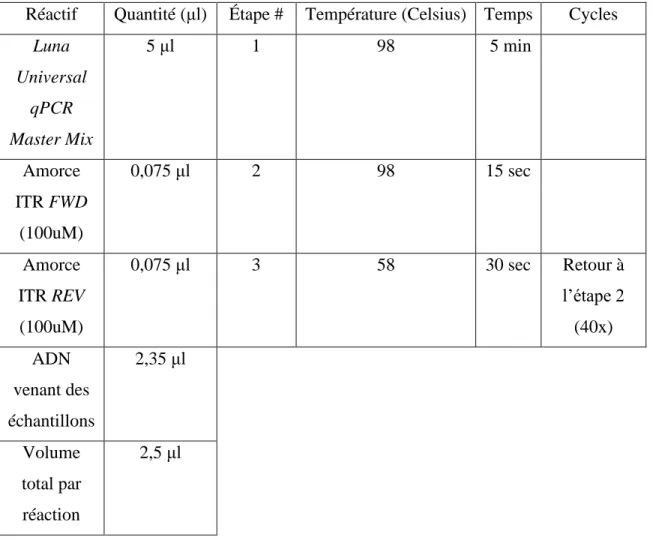
amplicons la première PCR, une seconde PCR a été effectuée avec les amorces sens universalFWDcap.1 ou universalFWDcap.2 (5’-TGCCTGGAGTACTTTCCGTCGCGA-TGCTGCGGACGGGGAACAAC-3’) en fonction de la combinaison d’amorces utilisées lors de la première PCR afin d’isoler les 1,2 kb de la fin de la région du gène CAP. Une analyse BLAST a été utilisée afin d’évaluer le degré de différence que possèdent les nouveaux AAV par rapport aux séquences disponibles dans GenBank. Les fragments isolés ont été nommés AAVpo5v, AAVpo4v et AAVpop1v1-15 en collaboration avec Monsieur Claude Lamarre. Pour AAVpo5v, une PCR semi-emboitée a été performée dans le but d’isoler le gène CAP au complet ainsi que la fin du gène REP et le fragment d’ADN généré avait la taille de 3kb. La première PCR utilise l’amorce sens FunivREP1 (5’-TGCTTATCTGGTGGGAGGAG-3’) et les amorces antisens CED-H ou CED+H et la deuxième PCR utilise l’amorce sens FunivREP2 (5’-GTGGGAGGAGGGCAAGAT-3’) et les amorces antisens CED-H ou CED+H. Les PCR ont fait appel à la polymérase PrimeSTAR MAX DNA (Takara, Clontech, JA, USA) et le cycle PCR est présenté dans la table 2.1.
29
Figure 2.1- Survol de l’isolation de nouveaux AAV porcins
Le flux du diagramme décrit sommairement les différentes étapes nécessaires à l’isolation du gène CAP de nouveaux AAV venant de fèces de porc.
30
Table 2.1 : Le cycle PCR pour l’isolation du gène CAP d’AAV
Réactifs Quantité (μl) Étapes # Températ ure (Celsius)
Temps Cycles
Amorce sens (10uM) 1,5 1 98 30 sec Amorce antisens (10uM) 1,5 2 98 10
PrimeSTAR MAX DNA Polymerase
25 3 70 15
DMSO 1,5 4 72 20 sec/kb Retour à
l’étape 2 (5x) H2O Volume pour atteindre
50
5 98 10
ADN porcin (200μg) Volume pour 200μg 6 65 15
Volume total 50 7 72 20 sec/kb Retour à l’étape 5 (5x) 8 98 10 9 60 15 10 72 20 sec/kb Retour à l’étape 8 (5x) 11 98 10 12 Tm des amorces -5 15 13 72 20 sec/kb Retour à l’étape 11 (30x) 14 72 6 min 15 4 infini
31
2- Traitement de l’ADN génomique à la Dnase dépendant de l’ATP
sûre pour les plasmides
Après l’extraction de l’ADN génomique décrit précédemment, 5 μg d’ADN génomique a été coupé par l’enzyme de restriction EcoRV durant un temps d’incubation de 1h à 37 degrés Celsius. Ensuite, 20% de l’ADN coupé a été digéré avec la Dnase dépendant de l’ATP sûre pour les plasmides (VWR) durant 30 minutes à 37 degrés Celsius pour être ensuite neutralisée par une température de 70 degrés Celsius durant 30 minutes. Une PCR semi-emboîtée a été effectuée avec les amorces sens Funivrep1 et FunivREP2 et l’amorce antisens RCAP3. Les bandes d’environ de 3 kb ont été envoyées au séquençage.
32
3-Clonage du gène CAP des nouveaux AAV et insertion du gène
CAP dans le plasmide DJ
Tous les fragments de PCR obtenus lors de l’étape précédente ont migré dans un gel d’agarose 1% sous 90 V durant environ 50 minutes dans du tampon TAE. Le marqueur ADN utilisé est le 1 kb Plus DNA Ladder (New England BioLabs). Après la migration sur gel, le gel a été visualisé à l’aide d’une machine à photographie à UV et les bandes ayant la taille voulue ont été coupées et purifiées sur gel en utilisant le PCR cleanup and gel extraction kits (TakaraBio) en suivant les recommandations du protocole du fabricant. Les produits PCR extraits du gel ont été plus tard clonés dans le vecteur pCR®-Blunt II-TOPO® (ThermoFisher Scientific)
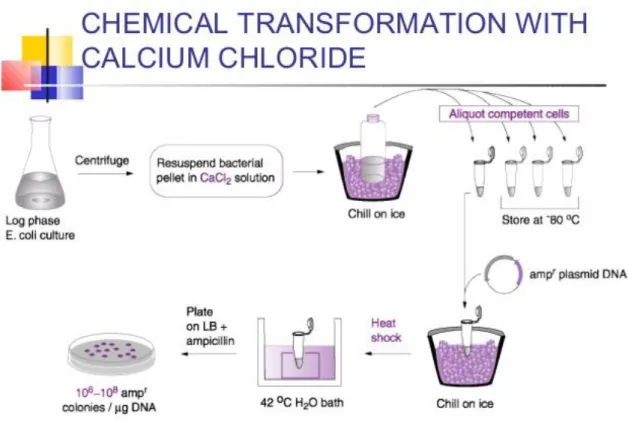
Le clonage des produits PCR a été réalisé en utilisant le Zero Blunt™ TOPO™ PCR Cloning Kit et le vecteur pCR®-Blunt II-TOPO® comme décrit. Après l’incubation du mélange réactif, les plasmides fraîchement ligués ont été transformés dans des cellules E.coli compétentes (Stellar chemically competent cells, TakaraBio) en utilisant les recommandations du fabricant. La transformation a été effectuée en les soumettant à un choc thermique de 42 degrés Celsius. Les cellules transformées ont été inoculées dans des plaques d’agar contenant un bouillon lysogène (LB) supplémenté avec 50 ul/ml de kanamycine, puis les plaques ont été incubées à 37 degrés Celsius durant toute la nuit. Le lendemain, plusieurs colonies de chaque plaque ont été repiquées dans 3 ml de bouillon LB et placées dans un incubateur-agitateur à 225 rpm à 37 degrés Celsius durant toute la nuit.
33
Figure 2.2 : clonage de plasmide avec gène CAP par transformation chimique de E.coli competent
Représentation schématique de la technique de la transformation chimique pour le clonage de plasmide. Une colonie sera repiquée dans 3 ml de LB résistant à l’antibiotique de sélection avant d’être récoltée. Les bactéries récoltées vont voir leurs plasmides extraits.
34
Tous les plasmides produits ont été purifiés en utilisant le NucleoSpin Plasmid, Mini kit for plasmid DNA (Macherey-Nagel) et le protocole du fabricant a été suivi à la lettre. Les échantillons ont été élués dans 50 µl de tampon EB (Macherey-Nagel) et les digestions restrictives ont été performées à l’aide d’enzymes de restriction venant de Fisher Scientific selon les recommandations du fabricant.
Les plasmides contenant le gène CAP ont été filtrés en digérant avec EcoRI (Fisher Scientific) et les plasmides contenant des insertions ont été envoyés au séquençage. Le séquençage a été performé par la Plateforme de séquençage SANGER du centre de recherche du CHU de Québec-Université Laval (CHUL) (Québec, Québec, Canada) en utilisant des amorces disponibles dans leurs librairies et des amorces commandées à IDT DNA Technologies. Tous les échantillons d’ADN envoyés ont été dilués à 50 ng/μl pour les plasmides et à 1,6 uM pour les amorces en utilisant de l’eau microfiltrée. Après la confirmation par séquençage, le gène CAP entier d’AAVpo5v a été ligué dans le vecteur pAAV-DJ, un plasmide ayant le gène REP de AAV2 permettant d’insérer le gène CAP d’un AAV afin de produire sa capside.
Pour mettre le gène CAP dans le plasmide DJ (voir annexe A1), une PCR en une étape avec le plasmide topo AAVpo5v a été effectuée avec l’amorce sens cF2 (5’- TGACTGCATCTTTGAACAATAAATGATTTAAATCAGGTATGTCGTTTGTTGA-3’) et l’amorce antisens cR2 (5’-CTGAAACGAATCAACCGGTTTATTGATTAACAG-GCAATTACAGAGGTCGGGTAAGGTAGC-3’) afin d’obtenir complètement et uniquement le gène CAP d’AAVpo5v. Ensuite, une autre PCR en une étape avec le plasmide DJ a été effectuée avec l’amorce sens pF2 (5’-GCTACCTTACCCGACCTC-TGTAATTGCCTGTTAATCAATAAACCGGTTGATTCGTTTCAG-3’) et l’amorce antisens pR2 (5’-TCAACAAACGACATACCTGATTTAAATCATTTATTGTTCAAA-GATGCAGTCA-3’) afin d’avoir tout le plasmide DJ sans le gène CAP. Une migration sur gel a été effectuée afin d’évaluer que le produit PCR possédait la taille attendue et d’en
35
vérifier la pureté. En utilisant le Gibson Assembly® Master Mix (NEB), la proportion de gènes CAP obtenus par rapport au plasmide DJ était de 3 :1 et les consignes du fabricant ont été suivies à la lettre. Après une incubation à 50 degrés Celsius durant 15 minutes, le nouveau plasmide a été transformé dans des cellules E. coli compétentes (Stellar chemically competent cells, TakaraBio) avec la même méthode de transformation. Les cellules ont été inoculées sur des géloses d’agar supplémentées de 100μg/ml de carbénicilline et ont été incubées à 37 degrés Celsius durant toute la nuit. Le lendemain, plusieurs colonies de chaque plaque ont été repiquées dans 3 ml de bouillon LB et placées dans un incubateur agitateur à 225 rpm à 37 degrés Celsius durant toute la nuit. Tous les plasmides produits ont été purifiés en utilisant le NucleoSpin Plasmid, Mini kit for plasmid DNA (Macherey-Nagel) précédemment expliqué.
36
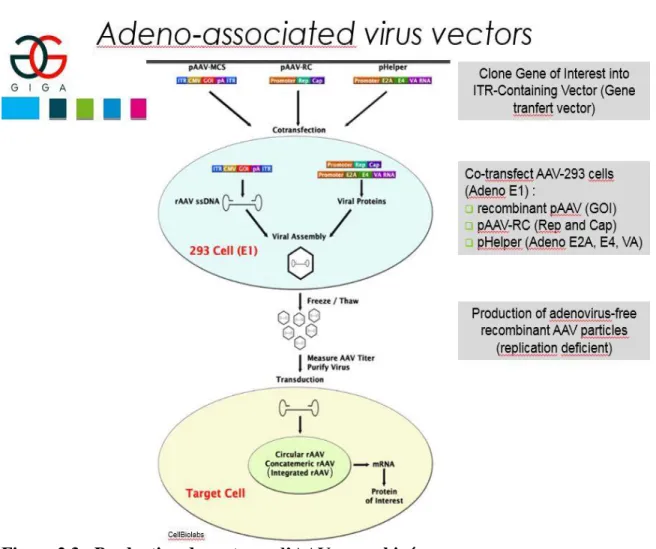
4-Production de capsides d’AAV avec un transgène et purification
4.1- Les plasmides impliqués dans la production des AAV recombinés.Les particules d’AAV hybrides ont été générées en utilisant trois plasmides différents. L’un des plasmides contient les gènes E2A, E4 et VA de l’adénovirus pour activer et aider la production de particules d’AAV dans la cellule nommer pHelper (voir annexe A2), un autre plasmide exprimant le transgène de manière cis et un dernier plasmide avec le gène REP de AAV2 et exprime de manière trans la capside des nouveaux AAV porcins (AAVpo5v, AAVpop1 et AAVpop2). Le gène CAP du sérotype AAV8 venant de macaques rhésus a été produit pour servir de témoin positif. Les gènes CAP des AAV ont été clonés à l’emplacement exact où se trouvait le gène CAP d’AAV2 en utilisant le Gibson assembly décrit ci-haut pour produire les plasmides DJ/AAVpo5v, pop1 et pop2 (voir annexe A1).
Un plasmide nommé pAAV.CMV.LacZ.bGH (voir annexe A3) exprime le gène rapporteur LacZ, lequel possède un promoteur CMV et est flanqué d’ITR d’AAV. Cela a pour effet d’exprimer le plasmide en cis et que le transgène soit contenu dans la capside recombinée. Le plasmide pAAV.CMV.LacZ.bGH sera impliqué dans des études de transduction in vitro dans cinq lignées cellulaires différentes.
Les génomes des AAV recombinés contenant soit le gène rapporteur LacZ flanqués des ITR de AAV2, dont l’expression est rendue possible par le promoteur CMV, ont été compactés par triple transfection dans des cellules de rein d’embryon humain (HEK) 293T.