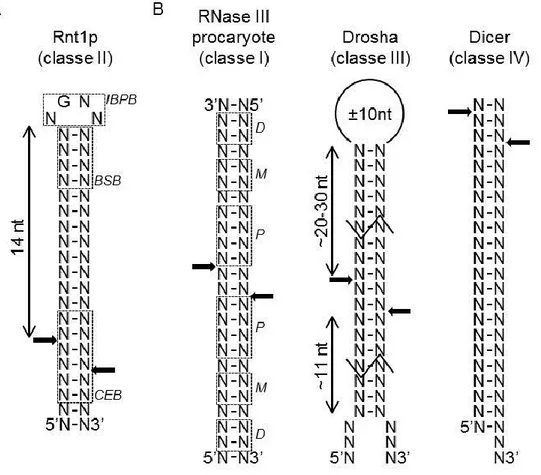
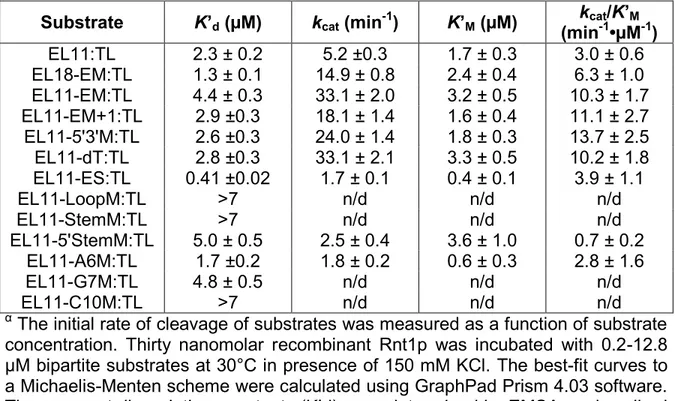
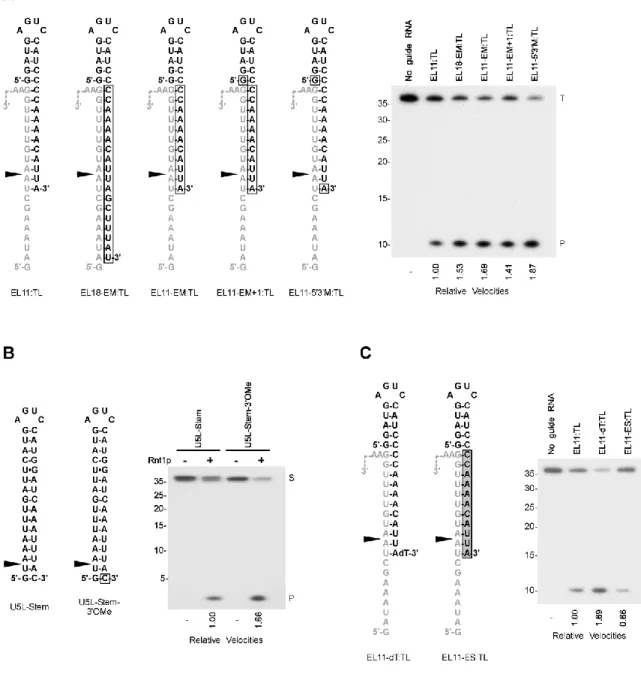
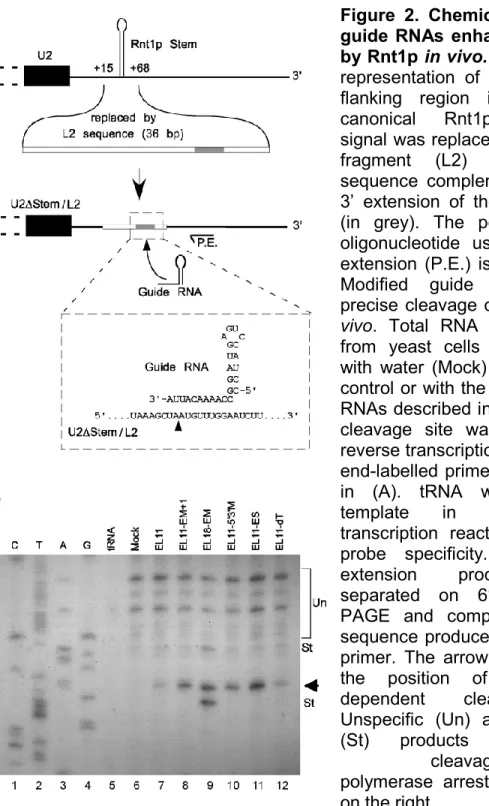
Étude des mécanismes de dégradation sélective de l’ARN par la RNase III de Saccharomyces cerevisiae
Texte intégral
Figure

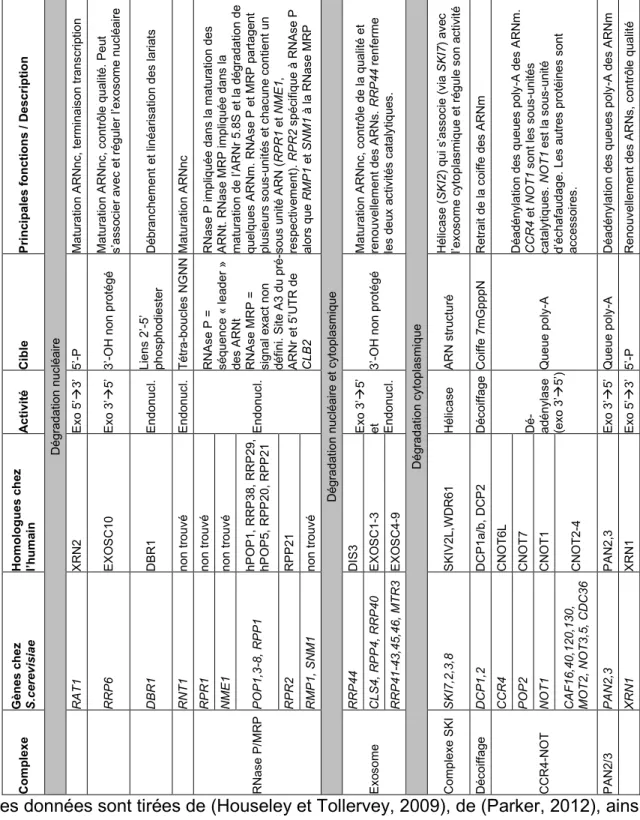

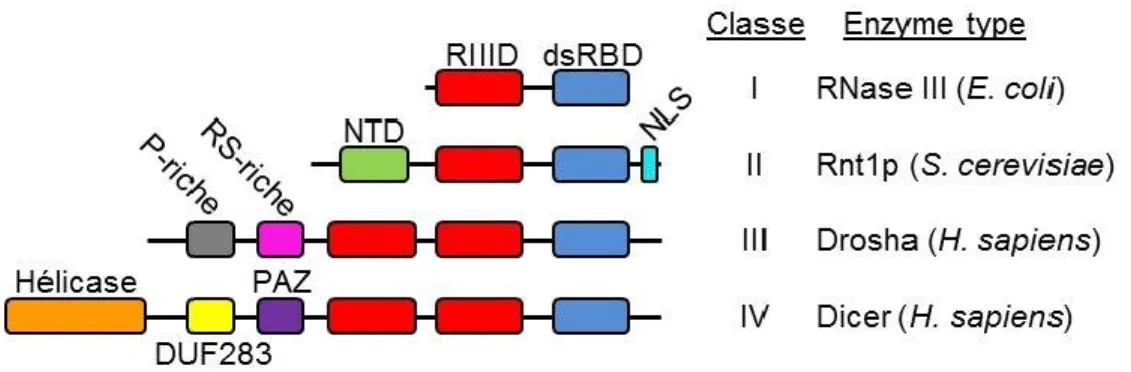
Documents relatifs
Table S1 and Figure S2: Temperature dependence of layer spacing d of compound 10/10 determined from the 2D X-ray measurements (solid squares) and tilt angle τ obs observed from the
Left panel: Some miRNA mimics as well as the negative control have no effect on the JAK/STAT3 signaling pathway, therefore a « normal » luciferase activity was measured in
Bound
In the following, we will also need the fact that if the set of vectors given as input to the LLL algorithm starts with a shortest non-zero lattice vector, then this vector is
Dans ce chapitre, nous avons détaillé la technique de mesure en cavité résonante tant en termes d’élaboration du système que l’utilisation de relations théoriques de
Pour cela, les entrées du modèle sont modulées puis les résultats de simulation analysés afin de définir une plage de variation de la vitesse du véhicule au cours de la
In the first section we introduce hyperk¨ ahler manifolds and we present the known examples. A fundamental result is Theorem 2.3, which relates the study of hyperk¨ ahler manifolds
Table S3 Biological process Gene Ontology (GO) term analysis of differentially expressed transcripts shared in the comparisons of non-transgenic NT:NT leaves versus non-transgenic and