© Patrick Gagné, 2020
Élaboration d’une méthodologie d’analyses
bio-informatiques pour des données de séquençage
Illumina dans un contexte de metabarcoding
Mémoire
Patrick Gagné
Maîtrise en microbiologie - avec mémoire
Maître ès sciences (M. Sc.)
Élaboration d’une méthodologie d’analyses
bio-informatiques pour des données de séquençage
Illumina dans un contexte de metabarcoding
Mémoire
Patrick Gagné
Sous la direction de :
Dr. Simon Hardy, directeur de recherche
Dr. Jean A. Bérubé, codirecteur de recherche
ii
Résumé
Le metabarcoding, un domaine alliant les technologies de séquençage à haut débit et l’identification d’organismes biologiques via l’ADN, a permis d’ouvrir les portes à un grand nombre de nouveaux projets scientifiques. Il existe une multitude de programmes capables d’effectuer les analyses, mais ces programmes ne sont souvent pas adaptés pour des projets particuliers comme la détection de pathogènes. ADAPTI a été développé afin de répondre à ce manque. ADAPTI est une méthodologie complète d’analyse adaptée pour la détection de pathogènes, mais capable de s’adapter à différents contextes de recherche grâce à son développement flexible et extensible. Toute la méthodologie a été mise à l’épreuve afin d’évaluer sa capacité, tant au niveau de l’efficacité en temps, mémoire vive et en espace disque, qu’au niveau de la validité et l’exactitude des résultats. Il a été démontré qu’ADAPTI est non seulement capable de fournir des résultats sur des jeux de données de grande taille, mais il le fait avec une grande sensibilité tout en conservant une efficacité multifilaire acceptable. ADAPTI a ensuite été comparé à des programmes open source en utilisant un jeu de données standardisé et il a été déterminé qu’il permet d’effectuer de meilleures analyses que les autres programmes testés. Il a été déterminé que la haute sensibilité fait la force d’ADAPTI, mais aussi l’une de ses faiblesses en permettant une plus grande quantité de séquences « bruits », lesquelles nécessitent des traitements supplémentaires. Certains programmes composant la suite d’ADAPTI nécessitent de l’optimisation au niveau de l’utilisation de la mémoire vive, ce qui est souvent un enjeu en bio-informatique. Il sera aussi nécessaire d’implémenter des capacités de calcul parallèle massif, car l’évolution rapide des technologies de séquençage rendra ADAPTI inefficace dans sa forme actuelle d’ici quelques années. Somme toute, ADAPTI est une méthodologie fonctionnelle capable de répondre à la demande en pathologie.
iii
Table des matières
Résumé ... ii
Liste des figures ... vii
Liste des tableaux ... viii
Liste des abréviations ... ix
Introduction... 1
1 Chapitre 1 : Concepts, revue de littérature et objectifs ... 4
1.1 Code Barre (barcode) ... 4
1.2 Metabarcoding ... 5
1.3 Séquençage haut débit ... 6
1.3.1 Pyroséquençage 454 ... 6
1.3.2 Illumina ... 7
1.3.3 Ion Torrent ... 8
1.3.4 PacBio et Nanopore MinION ... 8
1.3.5 Tableaux résumés ... 9
1.3.6 Capacité Paired-End ... 10
1.4 Traitement des données ... 10
1.4.1 Les boîtes à outils ... 10
1.4.2 Les pipelines ... 11
1.5 Amorces de séquençage ... 12
1.5.1 Les amorces dégénérées ... 14
1.6 Architecture informatique ... 15
1.6.1 Mémoire partagée ... 16
1.6.2 Mémoire distribuée ... 16
1.7 Développement informatique ... 18
iv
1.7.2 Programmation parallèle (architecture mémoire partagée) ... 21
1.7.3 Programmation distribuée (architecture à mémoire distribuée) ... 24
Efficacité ... 26
Données massives ... 26
Adaptabilité et extensibilité ... 27
Simplicité de mise en place ... 28
Facilité d’utilisation ... 28
Objectifs du projet ... 28
2 Chapitre 2 : Matériel et méthodes ... 29
2.1 Données ... 29
2.1.1 Préparation des données d’évaluation ... 29
2.2 ADAPTI ... 31
2.3 Processus d’essai ... 31
2.4 Étapes de traitement ... 32
2.4.1 Démultiplexage ... 32
2.4.2 Réassemblage ... 33
2.5 Nettoyage des séquences ... 36
2.6 Déréplication ... 37
2.7 Retrait des homopolymères ... 38
2.8 Élimination des chimères ... 41
2.9 Traitements spécifiques pour les champignons et les insectes ... 43
2.9.1 Traitement des ITS1 fongiques ... 43
2.9.2 Traitement spécifique aux COI ... 46
2.10 Regroupement (clusterisation) ... 47
v
2.10.2 Swarm ... 50
2.11 Identification des OTUs ... 50
2.12 Contexte de génération des résultats ... 52
3 Chapitre 3 : Résultats ... 53
3.1 Efficacité multifilaire... 53
3.2 Efficacité mémoire vive (RAM) ... 56
3.2.1 L’approche du pire cas ... 56
3.2.2 L’approche en temps réel ... 57
3.3 Efficacité espace disque ... 60
3.4 Comparaison avec d’autres programmes ... 61
4 Chapitre 4 : Discussion ... 65
4.1 Efficacité multifilaire... 65
4.2 Efficacité mémoire vive ... 67
4.3 Efficacité espace disque ... 68
4.4 Comparaison avec d’autres programmes ... 69
4.5 Calcul parallèle massif ... 71
4.6 Remplacement des OTUs par les Exact Sequence Variant (ESV) ... 72
Conclusion... 74
Retour sur les objectifs ... 74
Efficacité ... 75 Données massives ... 75 Adaptabilité et extensibilité ... 75 Simplicité d’installation ... 75 Facilité d’utilisation ... 76 Développements futurs ... 76 Checkpointing et résilience ... 76
vi
Capacité parallèle massive ... 77
Optimisation en mémoire vive ... 77
Exploration des rejets ... 77
Ajout de modules pour Ion Torrent ... 78
Application à des projets d’écologie ... 79
vii
Liste des figures
Figure 1: Fragment Illumina Paired-End typique ... 12
Figure 2 : Amorce spécifique aux organismes fongiques (Nelsen, 2008) ... 13
Figure 3 : Décalage de l’amorce ITS1F ... 13
Figure 4 : Architecture à mémoire partagée (Semantic Scholar) ... 16
Figure 5 : Architecture à mémoire distribuée (Washington University in St.Louis) 17 Figure 6 : Principe du calcul parallèle de base ... 22
Figure 7 : Transmission de message par MPI ... 25
Figure 8 ; Amélioration de la qualité grâce au paired-end et risque d’erreur pour un fragment trop long ... 34
Figure 9 : Principe du réassemblage et de l’amélioration de la qualité ... 35
Figure 10 : Principe de fenêtre coulissante ... 36
Figure 11 : Principe de la cartographie de fichier ... 40
Figure 12 : Fonctionnement de HomopRemover ... 40
Figure 13 : processus de génération de k-mers ... 42
Figure 14 : Fonctionnement de CoxFilter avec une limite de 0 codon stop ... 47
Figure 15 : Défaut du regroupement par algorithme glouton selon l’ordre des séquences ... 49
Figure 16 : Graphique de performance de blastn (Pascal, 2014) ... 51
Figure 17 : Efficacité multifilaire de ADAPTI pour le jeu de donnée 25M ... 54
Figure 18 : Efficacité multifilaire d’ADAPTI pour le jeu de données 25M par rapport au nombre de fils d’exécution (4 fils d’exécution comme base de comparaison) .. 54
Figure 19 : Efficacité multifilaire de ADAPTI pour le jeu de donnée 250M ... 55
Figure 20 : Graphique de la demande en mémoire vive en fonction du temps ... 56
Figure 21 : Graphique d’évaluation de la mémoire vive en fonction du temps avec 17 millions de séquences en entrée du programme ... 58
Figure 22 : Graphique d’évaluation de la mémoire vive en fonction du temps avec 129 millions de séquences en entrée du programme ... 58
Figure 23 : Graphique d’évaluation de la mémoire vive en fonction du temps avec 250 millions de séquences en entrée du programme ... 59
Figure 24 : Graphique de l’espace disque nécessaire à ADAPTI en fonction du nombre de séquences brutes en entrée ... 61
viii
Liste des tableaux
Tableau 1 : Résumé des différentes technologies de séquençage haut débit (Besser, Carleton, Gerner-Smidt, Lindsey, & Trees, 2018). Le pyroséquençage n’est pas été inclus dans le tableau, car cette technologie n’est plus supportée. .... 9 Tableau 2 : Résumé des coûts et temps de traitements des différentes technologies de séquençage haut débit (Bleidorn, 2016) ... 10 Tableau 3 : Tableau des bases dégénérées selon la nomenclature de l’IUPAC (modifié par Patrick Gagné afin d’inclure la notation I et X) (bioinformatics.org) ... 15 Tableau 4 : Sommaire des jeux de données uniques utilisé pour évaluer ADAPTI29 Tableau 5 : Composition des différentes combinaisons de jeux de données pour tester ADAPTI ... 30 Tableau 6 : Composition du jeu de donnée Mock ... 31 Tableau 7 : Processus de sélection du motif SSU ... 44 Tableau 8 : Synthèse résultats (échantillons uniques, données provenant de Hoggart 2018) ... 62 Tableau 9 : Synthèse résultats (échantillons assemblé) comparaison des pipelines ADAPTI, DADA2, Snakemake AAFC et Hoggard 2018 ... 63 Tableau 10 : Tableau de décompte des OTUs pour ADAPTI, AAFC_Snakemake et dada2 ... 63 Tableau 11 : Synthèse des caractéristiques, capacités et performances des différents pipelines de metabarcoding ... 64
ix
Liste des abréviations
ADN: Acide désoxyribonucléique COI: Cytochrome oxidase I COX: Cytochrome oxidase I ITS: Internal Transcribed Spacer SSU: Small Subunit
LSU: Large Subunit
PCR: Polymerase Chain Reaction SHD: Séquançage Haut-débit CPU: Core Processing Unit
MPI: Message Passing Interface OTU: Operational Taxonomical Unit Pb: Pair de bases
NCBI: National Center for Biotechnology Information Go: Gigaoctet
RAM: Random Access Memory I/O: Input/Output
ESV: Exact Sequences Variants SQL: Structured Query Language
1
Introduction
Depuis plusieurs années, l’évolution très rapide des technologies de séquençage ADN a permis le développement de nouvelles branches dans l’étude des sciences de la vie et notamment de la biodiversité. Grâce aux avancées technologiques, il est désormais possible d’obtenir plusieurs millions de séquences ADN d’un échantillon environnemental, permettant ainsi d’identifier les organismes présents dans celui-ci. Cette nouvelle branche se nomme le metabarcoding et est un domaine alliant les technologies de séquençage à haut débit (pyroséquençage 454, Illumina, Ion Torrent, PacBio, etc.) et l’identification d’organismes biologiques via l’ADN.
Comme le metabarcoding existe depuis plusieurs années, il existe une multitude de programmes et de techniques permettant l’analyse et l’interprétation des résultats. Malgré tout, la prévalence des études écologiques a contribué à une sous-représentation des autres types de projets comme la recherche de pathogènes, ce qui rend ce type d’étude plus difficile avec les outils disponibles. C’est de ce manque d’outils que ce projet est né, car, dans un contexte de recherche en pathologie, l’approche par metabarcoding est particulièrement intéressante puisqu'elle permet le suivi de dizaines de pathogènes simultanément, et non pas un à la fois comme avec les sondes moléculaires. Ce projet vise à développer une méthodologie adaptée à la détection de pathogènes forestiers et ainsi contribuer à combler le manque dans ce domaine particulier.
Le développement d’une nouvelle méthodologie de traitement de données de metabarcoding n’est pas une mince affaire. Il est nécessaire de non seulement comprendre le principe et le fonctionnement des différentes étapes d'obtention des données et des technologies s’y rattachant (type de séquençage, format des données etc.), mais aussi de posséder une connaissance avancée en programmation et en algorithmique. Une fois ces éléments maitrisés, le travail est loin d’être terminé. En effet, il est nécessaire d’identifier les éléments structuraux
2
qui composeront la méthodologie, comme le type de langage à utiliser, les dépendances à inclure, les outils à développer et plus.
Ce mémoire traite du développement et de la composition d’une telle méthodologie. Il part de l’acquisition des connaissances requises, en passant par le développement algorithmique pour conclure avec l’évaluation des performances puis de la validité des résultats produits. Il contient tous les éléments nécessaires à la conception, la mise-en-place, l’évaluation et l’optimisation d’une méthode bioinformatique appliquée à un domaine précis. Il faut cependant noter qu’en programmation et en algorithmique, il peut exister une infinité de solutions à un problème précis et qu'il existe probablement toujours une solution plus efficace et/ou moins coûteuse que la solution appliquée à ce problème. C’est pourquoi il est généralement impossible d’assurer à l’utilisateur (ou au programmeur) que la méthode qu’il préconise soit la meilleure. Si l’on considère aussi que chaque projet de recherche possède des objectifs différents, la bonne méthode pour un projet peut être inadéquate pour un autre projet.
Afin de saisir le développement et le fonctionnement de la méthodologie, il est nécessaire de non seulement comprendre les concepts du metabarcoding, mais aussi de comprendre les concepts bio-informatiques, incluant les outils existants et les notions de programmations afin de saisir les différents choix faits lors du développement et pourquoi ces choix ont été faits. Ce mémoire traitera de certains programmes composant la méthode, généralement les programmes principaux et les raisons de leur développement, mais ne traitera pas de tous les algorithmes, car la méthode est composée de plus d’une quarantaine de scripts développés uniquement pour son fonctionnement et il serait trop ambitieux de les décrire exhaustivement.
Tout développement nécessite une phase test, où les composants du système (ou dans le cas de ce projet: de la méthode) doivent être évalués en condition contrôlée pour déterminer leur validité, robustesse, efficacité, et bien sûr, leurs faiblesses. Étant donné que ce projet vise le développement d’une nouvelle méthode de traitement, il est nécessaire de la tester exhaustivement afin d’assurer
3
sa qualité. Ces tests seront principalement des tests d’efficacité informatique comme l’efficacité multifilaire, l’efficacité en mémoire vive, l’efficacité d’espace disque et la comparaison de ces valeurs avec ceux d’outils accessibles sur Internet, mais aussi l’évaluation de la validité des résultats en comparaison avec des études récentes dans le domaine.
4
1 Chapitre 1 : Concepts, revue de littérature et objectifs
Les prochaines sections traiteront des concepts reliés au metabarcoding, aux définitions des outils du domaine et des concepts informatiques généraux nécessaires à la création de la méthodologie.
1.1 Code Barre (barcode)
Il est récent de pouvoir identifier un organisme, autrement que de le catégoriser selon ses caractéristiques morphologiques et biologiques. Pour les champignons, par exemple, la classification demande une connaissance très approfondie de leur morphologie et des types de reproductions. De plus la planète est composée de plusieurs millions d’espèces et seule une poignée de taxonomistes sont en mesure d’identifier au plus 0.01% de ces espèces (Hebert, Cywinska, Ball, & Dewaard, 2003). Dans les années 80, une nouvelle technique voit le jour: l’identification micro génétique. Elle permit la discrimination des organismes difficiles à caractériser par le biais d’une petite portion d'ADN du génome de l’organisme cible, dans ce cas-ci des protozoaires (Nanney, 1982). Bien que cette méthode ait été à la base développée pour la discrimination des organismes pour qui la caractérisation causait des problèmes, cette technique a commencé à prendre de l’ampleur vers les années 2000 où elle a été appliquée à des organismes plus complexes, comme des lépidoptères (Brown, Emberson, & Paterson, 1999). Le terme code-barre génétique est utilisé dans ce contexte, car les portions d’ADN ciblées permettent l’identification et la discrimination d’organismes. Selon le type d’organisme à identifier, la région code-barre ciblée sera différente; pour les animaux, les insectes et certains protistes, une portion d’environ 600 paires de bases du gène mitochondrial de la cytochrome oxidase I (aussi appelé COI ou
COX) est utilisée (Ji et al., 2013). Pour les champignons, les portions non codantes
intercalant les gènes codants pour les sous-unités ribosomiques SSU, 5.8S et LSU sont utilisées. Ces portions servent d’espaceurs entre ces gènes d’où leur nom Internal Transcribed Spacers (ou ITS). Ces séquences sont un bon code-barre pour les champignons, car ils sont soumis à moins de pression sélective que les SSU et LSU, et donc accumulent plus facilement les mutations. Malgré l’apparente
5
puissance des codes-barres, il ne faut pas oublier que certaines espèces sont plus difficiles que d’autres à différencier, par exemple, Chalara fraxinea, causant la Chalarose du frêne, est un champignon possédant une similarité de 98.17% avec
Hymenoscyphus albidus, alors que celui-ci n’est pas pathogène (Queloz et al.,
2011).
1.2 Metabarcoding
Grâce aux avancées technologiques du séquençage à haut débit, il est désormais possible d’obtenir des millions de séquences d’un échantillon environnemental. En couplant cette capacité de production de séquences avec le principe du code-barre génétique, il est possible d’obtenir une quantité importante de codes-barres des différentes espèces présentes dans un échantillon donné. Cette approche porte le nom de metabarcoding.
Le metabarcoding est très utilisé de nos jours, et ce dans une multitude de contextes. L’usage principal est dans les études de biodiversité, et ce pour plusieurs types d’organismes, comme les poissons (Civade et al., 2016), les insectes (Ji et al., 2013), les bactéries (Sapp et al., 2016), les champignons (Schmidt et al., 2013), etc.
Un projet utilisant le metabarcoding commence par l’identification du type d'organismes cibles et du ou des codes-barres qui lui sont associés. Cette étape demande normalement une revue de littérature afin d’identifier les codes-barres appropriés.
Une fois le code-barre identifié, il est nécessaire de sélectionner les amorces qui permettent l’amplification par PCR de la cible. Une fois les amorces de séquençage prêtes, l’ADN contenu dans les différents échantillons doit être extrait par le biais d’un kit d’extraction ADN. Ces ADN doivent ensuite être amplifiés, purifiés, dosés et finalement mis en commun pour former le « pool » de séquençage, qui sera envoyé à la plateforme de séquençage pour traitement. Une fois les données brutes générées, il est nécessaire de procéder à l’analyse bio-informatique pour obtenir les résultats sur l’objet de l’étude.
6
1.3 Séquençage haut débit
Successeurs à la technologie Sanger, les méthodes de séquençage haut débit (SHD) sont la pierre angulaire de la plupart des projets portant sur l’ADN. Il existe plusieurs méthodes de séquençage à haut débit qui seront décrites dans les prochaines sections
1.3.1 Pyroséquençage 454
Le pyroséquençage 454, considéré comme l’une des premières méthodes de séquençage de seconde génération, a été développé par la compagnie Roche en 2005 (Margulies et al., 2005). Contrairement au séquençage Sanger, le 454 est en mesure de séquencer un grand nombre de fragments d’ADN différents simultanément (d’où l’appellation de séquençage haut débit). Le séquençage 454 commence par la ligation et l’amplification des fragments d’ADN sur support solide (l’amplification permet d’augmenter la puissance du signal lumineux et donc de permettre aux capteurs de fonctionner correctement). Une fois l’amplification terminée, l’appareil envoie, à chaque cycle, des flux de dNTP (des bases azotées A, C, T et G couplé à une molécule fluorescentes) non aléatoires. Lorsque, par exemple, l’appareil envoie un flux d’adénine, toutes les séquences possédant une thymine recevront l’adénine dans leur chaîne complémentaire. De cette incorporation résultera la libération d’un pyrophosphate qui, par le biais d’autres réactions chimiques, permettra l’émission de lumière. Cette lumière est ensuite captée par les caméras pour ensuite être converti en base ADN par l’appareil. L’avantage principal du 454 était la taille des séquences pouvant atteindre jusqu’à 700 Pb et un faible taux d’erreur (meilleur que l’Illumina). Le principal défaut du pyroséquençage (mis à part son coût élevé) est au niveau de l’imprécision lors du séquençage des homopolymères. Un homopolymère est une succession de bases ADN identiques (exemple AAAAAAA). La cause de l’imprécision en 454 est due au principe de flow, car lorsqu’un dNTP est ajouté, un homopolymère incorporera autant de dNTP que sa longueur, ce qui résultera en une augmentation du signal. Si les homopolymères sont trop longs, les capteurs seront surchargés et la prise de mesure deviendra imprécise, il sera alors impossible de déterminer la longueur réelle de l’homopolymère en question. Cette technologie est aujourd’hui désuète et
7
n’est plus supportée par Roche dû notamment à la performance nettement supérieure et au coût réduit des technologies plus récentes.
1.3.2 Illumina
Aussi appelé séquençage par terminateur réversible, le séquençage Illumina voit le jour en 2008 (Bentley et al., 2008). La technologie Illumina utilise un principe similaire à celui de la technologie 454, c’est-à-dire qu’elle emploie des nucléotides émettant de la lumière qui est captée et convertie en signal numérique pour devenir des bases de l’ADN. Comme le 454, les séquences doivent avant tout être amplifiées sur un support solide (cellule flow) afin d’augmenter la puissance du signal, pour ensuite être fixées à une matrice de séquençage. Le système injecte ensuite les 4 bases terminatrices et récupère les signaux lumineux. Comme les bases sont terminatrices, chaque cycle de séquençage fournit qu’une base par séquence, et ce même si la séquence en question possède une succession de la même base (homopolymères). Donc contrairement au 454, Illumina n’aura pas de problèmes avec les homopolymères trop longs. Le cycle se répète alors jusqu’à la complétion du séquençage. Il existe 3 types de séquençage Illumina : le MiSeq, le HiSeq et le GAIIx. Il y a peu de différences au niveau du principe de séquençage pour les 3 méthodes, la principale différence étant l’appareillage utilisé (par exemple, Illumina Hiseq utilise un appareil beaucoup plus imposant et beaucoup plus coûteux pour effectuer le séquençage). Cette différence explique l’écart important entre le nombre de séquences fournies par MiSeq par rapport au HiSeq. La technologie Illumina est devenue l’une des méthodes les plus utilisées aujourd’hui, principalement en raison de sa précision, de son rendement, qui est d’environ 1-2Gb par opération en 27 heures avec un taux d’erreur d’environ 0.80% pour la technologie MiSeq , environ 600Gb par opération en 11 jours avec un taux d’erreur de 0.27% pour la technologie HiSeq (Bleidorn, 2016) (Quail et al., 2012) et jusqu’à 6000 Gb avec un taux d’erreur similaire au HiSeq pour la technologie NovaSeq (Besser et al., 2018) .
8
1.3.3 Ion Torrent
La technologie Ion torrent utilise un principe fondamentalement différent de l’Ilumina et du 454 pour effectuer le séquençage. Il s’agit de la technologie de séquençage par semi-conducteur Ion Torrent (Rothberg et al., 2011). Apparu vers 2011, Ion Torrent n’est pas basé sur la lecture d’un signal lumineux pour reconnaître les bases ADN, mais plutôt sur un circuit intégré capable de détecter les variations de pH dans des cellules contenant des billes sur lequel est fixé l’ADN à séquencer. Ion Torrent reprend le principe des flow (ou flux) en 454 (une succession non aléatoire de dNTP) et mesure la variation en pH induite par l’incorporation d’un dNTP par une polymérase à une chaîne d’ADN. Le système de détection purement électronique utilisé par Ion Torrent a permis de grandement diminuer les coûts de l’appareil et le coût d’opération. Au niveau de la vitesse d’exécution, Ion Torrent est nettement plus rapide qu’Illumina (2 heures par opération contre 27 heures pour Illumina) (Bleidorn, 2016). Le principal défaut de cette technologie est son taux d’erreur de 1.76%, ce qui est assez élevé pour des études de metabarcoding sachant que certaines espèces ne sont différenciables que par quelques nucléotides seulement.
1.3.4 PacBio et Nanopore MinION
Les technologies PacBio et MinION, bien que très prometteuses en génomique, ne sont malheureusement pas très bien applicables à des recherches utilisant le metabarcoding, principalement en raison des taux d’erreurs très élevés par rapport aux autres technologies; jusqu’à 15% pour le PacBio (Rhoads & Au, 2015) et vers 38% pour le MinION (Laver et al., 2015).
9
1.3.5 Tableaux résumés
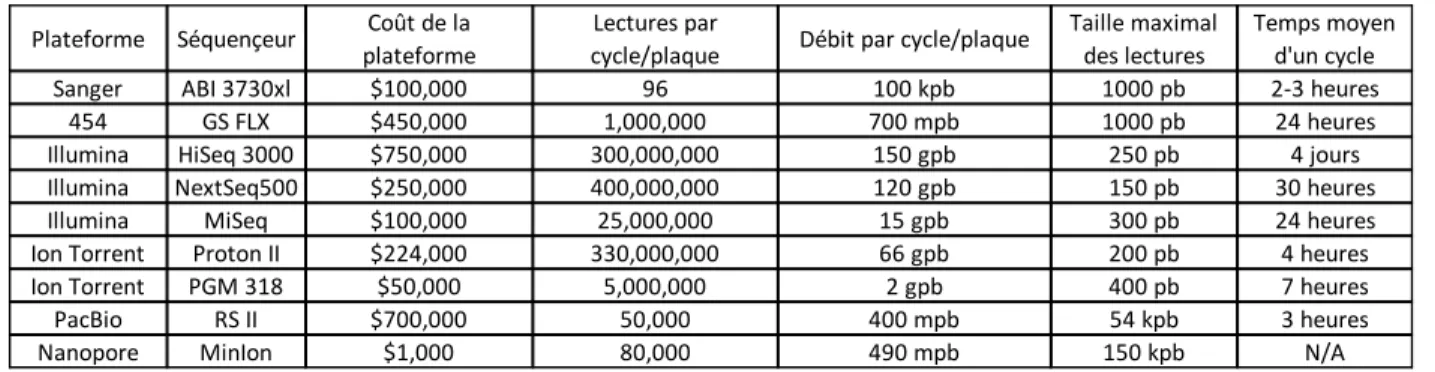
Tableau 1 : Résumé des différentes technologies de séquençage haut débit (Besser, Carleton, Gerner-Smidt, Lindsey, & Trees, 2018). Le pyroséquençage n’est pas été inclus dans le tableau, car cette technologie n’est plus supportée.
Plateforme
Instrument Débit (Gb)
Longueur de
séquences (pb) Forces Faiblesses
ABI 3500/3700 0.0003 Jusqu’à 1 Kb Précision de lecture et
longueur Coût et débit MiniSeq 1.7-7.5 1x75 à 2x150 Investissement initial
bas
Temps de traitement et longueur
MiSeq 0.3-15 1x36 à 2x300 Longueur des lectures,
extensibilité Temps de traitement NextSeq 10-120 1x75 à 2x150 Débit Temps de traitement et
longueur HiSeq (2500) 10-1000 1x50 à 2x250 Précision de lecture,
débit
Investissement initial élevé et temps de traitement
NovaSeq 5000/6000 2000-6000 2x50 à 2x150 Précision de lecture, débit
Investissement initial élevé et temps de traitement
PGM 0.08-2 Jusqu’à 400 Longueur des lectures,
vitesse Débit, homopolymères S5 0.6-15 Jusqu’à 400 Longueur des lectures,
vitesse homopolymères Proton 10-15 Jusqu’à 200 Vitesse, débit homopolymères PacBio RSII 0.5-1b Jusqu'à 60Kb Longueur de lecture,
vitesse
Taux d'erreur élevé, coût initial élevé
Sequel 5-10b Jusqu'à 60Kb Longueur de lecture,
vitesse Taux d'erreur élevé MInION 0.1-1 Jusqu'à 100Kb Longueur de lecture,
portabilité
Taux d'erreur élevé, temps de traitement Séquençage Sanger
Illumina
IonTorrent
Pacific BioScience (PacBio)
10
Tableau 2 : Résumé des coûts et temps de traitements des différentes technologies de séquençage haut débit (Bleidorn, 2016)
1.3.6 Capacité Paired-End
La capacité paired-end est une forme de séquençage particulière pour les technologies Illumina et Ion Torrent. La méthode consiste à effectuer le séquençage dans les deux sens pour obtenir des fragments plus longs et de meilleure qualité. L’utilisation d’une telle méthode permet donc de presque doubler la taille des séquences et donc d’aller chercher plus d’information génétique. Pour les ITS fongiques, cette méthode est nécessaire, car la plupart des ITS sont plus longs que la longueur d’amplicons maximal qu’Illumina ou Ion Torrent peuvent fournir avec un seul brin.
1.4 Traitement des données
Une analyse metabarcoding Illumina MiSeq complète génère en moyenne 25 millions de séquences. Afin d’extraire les informations importantes de ces données, il est nécessaire de procéder à leur traitement pour isoler les caractéristiques recherchées qui sont différentes dépendamment du projet. Depuis plusieurs années, un grand nombre d’outils bio-informatiques ont été développés à cet effet. Les programmes d’analyse peuvent être séparés en 2 catégories : les pipelines et les boîtes à outils.
1.4.1 Les boîtes à outils
Les boîtes à outils sont un regroupement d’outils, mais sans structure d’analyse définie où l’utilisateur décide quelles étapes doivent s’enchainer pour produire les résultats voulus. Ces outils sont la plupart du temps difficiles à utiliser pour des utilisateurs non-initiés, car il est nécessaire de comprendre leurs fonctions pour
Plateforme Séquençeur Coût de la plateforme
Lectures par
cycle/plaque Débit par cycle/plaque
Taille maximal des lectures
Temps moyen d'un cycle Sanger ABI 3730xl $100,000 96 100 kpb 1000 pb 2-3 heures
454 GS FLX $450,000 1,000,000 700 mpb 1000 pb 24 heures Illumina HiSeq 3000 $750,000 300,000,000 150 gpb 250 pb 4 jours Illumina NextSeq500 $250,000 400,000,000 120 gpb 150 pb 30 heures Illumina MiSeq $100,000 25,000,000 15 gpb 300 pb 24 heures Ion Torrent Proton II $224,000 330,000,000 66 gpb 200 pb 4 heures Ion Torrent PGM 318 $50,000 5,000,000 2 gpb 400 pb 7 heures PacBio RS II $700,000 50,000 400 mpb 54 kpb 3 heures Nanopore MinIon $1,000 80,000 490 mpb 150 kpb N/A
11
réaliser une analyse cohérente. Souvent, les boîtes à outils vont contenir des exemples d’utilisation que l’utilisateur pourra suivre pour faciliter son travail, mais cela reste que c’est l’utilisateur qui doit décider si les lignes directrices répondent à ses besoins.
Usearch/Uparse (Edgar, 2013) est l’un des outils les plus populaires pour les analyses de metabarcoding, mais en raison du prix de la licence qui est très élevé (plus de 1000$ pour une version de Usearch, un montant qui doit être repayé pour obtenir les versions plus récentes), une alternative a été préféré : Vsearch (Rognes, Flouri, Nichols, Quince, & Mahé, 2016). Ce programme contient les mêmes outils que Usearch, mais il est à source ouverte et n’a pas de limitations au niveau de la mémoire. Selon sa documentation, Vsearch donne des résultats égaux ou supérieurs à Usearch et possède pratiquement les mêmes fonctions. La plupart des fonctions produisent des fichiers eux même compatibles avec Usearch. La boîte à outils la plus populaire est très probablement Qiime (Caporaso et al., 2010). Cette suite de programme est largement utilisée dans des projets d’écologie où il est nécessaire de caractériser des environnements complexes.
1.4.2 Les pipelines
Les pipelines sont un regroupement d’outils s’enchainant les uns à la suite des autres où la sortie d’un programme devient l’entrée d’un autre. Un pipeline a l’avantage d’être souvent plus facile à utiliser pour les utilisateurs non-initiés, car la compréhension des traitements effectués sur les données à chaque étape n’a pas besoin d’être exhaustive pour obtenir des résultats. Souvent, chaque pipeline est ajusté pour un type de projet, ce qui les rend moins versatiles que les boîtes à outils.
Les pipelines sont souvent statiques, c’est-à-dire qu’ils ne sont pas modifiables dans leurs étapes, mais chaque étape reste relativement paramétrable. La méthode d’implémentation est variable, mais les deux méthodes les plus populaires restent l’implémentation Bash et l’implémentation Python.
12
L’implémentation Bash consiste en un développement direct où les commandes sont exécutées les unes à la suite des autres suivant une structure logique pour effectuer l’analyse complète. Cette forme de pipeline est très simple à mettre en place et nécessite peu de logiciels externes aussi appelés dépendances (mise-à-part les programmes composant le pipeline), mais demande un développement parfois exhaustif pour obtenir une structure complète.
L’implémentation en Python fait normalement appel à des librairies particulières conçues spécialement pour la mise en place de pipelines, des librairies comme Snakemake (Köster & Rahmann, 2012) ou GenPipe (Bourgey et al., 2019), où des structures de pipelines sont déjà programmées et où il suffit de développer les différentes étapes d’analyses pour obtenir un pipeline complet. Bien qu’alléchant, ces librairies sont souvent difficiles d’installation étant donné le grand nombre de dépendances à installer et la courbe d’apprentissage assez abrupte pour le développement des étapes d’analyses.
1.5 Amorces de séquençage
Lors d’un séquençage Illumina Paired-end, il est nécessaire de préparer les amplicons d’ADN de manière adéquate. Pour ce faire, chaque fragment ADN d’un échantillon doit être amplifié de manière à posséder les caractéristiques présentes dans la figure 1. Les amorces spécifiques notées ici ITS1F et ITS7G sont des amorces universelles pour les gènes ribosomiques des champignons, mais peuvent être remplacées par d'autres amorces dépendamment du type d'organismes étudiés.
Figure 1: Fragment Illumina Paired-End typique
Les amorces Illumina sont spécifiques à la matrice de séquençage des plaques Illumina, elles permettent aux fragments de se lier à la matrice pour initier le séquençage. Les index avant et arrière sont les étiquettes d’identification des différents échantillons, car lors de la préparation au séquençage, les différents
13
échantillons doivent être mélangés de manière équimolaire. Sans les étiquettes, il serait impossible de les différencier suite au séquençage. Les amorces sont spécifiques au type d’organismes étudiés, elles permettent d’isoler le fragment d’intérêt dans le mélange d’ADN génomique suivant l’extraction d’ADN. Pour les organismes fongiques, le fragment d’intérêt est l’ITS (décrit à la section 1.1). Pour bien cibler ce fragment, l' amorce ITS1F (Gardes & Bruns, 1993) située dans une région très conservée du gène 18S (codant pour une sous-unité ribosomique) et l’amorce ITS7G, située dans une région très conservée du gène 5.8S (Ihrmark et al., 2012) sont utilisées.
Figure 2 : Amorce spécifique aux organismes fongiques (Nelsen, 2008)
Les amorces spécifiques sont aussi décalées, c’est-à-dire que le début des amorces contient un nombre variant de 0 à 3 nucléotides aléatoires selon cette figure :
Figure 3 : Décalage de l’amorce ITS1F
Le décalage (Wu et al., 2015) est très important lors du séquençage, car la prise de mesure de la luminosité en Illumina commence toujours à la première base de l’amorce spécifique à l’espèce. Sans le décalage toutes les séquences émettraient de la lumière au même moment, et ce tout le long de l’amorce, ce qui surchargerait
14
les capteurs de l’appareil et rendrait toute lecture des bases impossible. Le décalage permet de déphaser le cadre de lecture des différentes séquences et donc d’éviter la surcharge des capteurs. Il est toutefois important de considérer le décalage lors des analyses subséquentes, car la délétion directe de l’amorce par une méthode de type détection/remplacement laissera les bases de décalage dans la séquence ADN.
1.5.1 Les amorces dégénérées
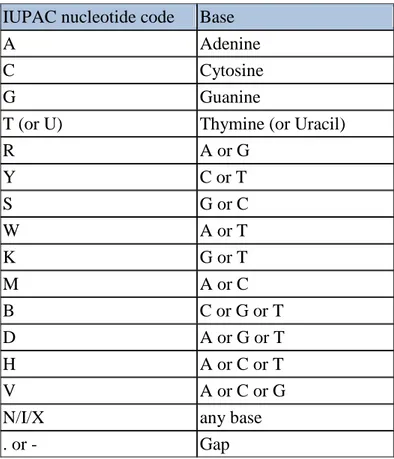
Lors du développement d’une amorce universelle pour un organisme cible, il est parfois impossible de trouver une portion ne comportant aucune variation entre les différentes espèces du type d’organismes cibles. Lors d’une amplification typique, plus une amorce possède une affinité pour une séquence ADN X (c’est-à-dire que la séquence de l’amorce est très proche de la séquence ADN, à amorcer), plus cette séquence sera favorisée par rapport aux autres possédant moins d’affinité. Si une séquence d’amorce consensus est utilisée (donc une séquence moyenne), il est très probable que des espèces ne soient pas amplifiées et donc n’apparaitront pas durant l’analyse bio-informatique. Pour éviter ce biais, il est nécessaire de faire appel à l’amorce dégénérée. Dans une amorce dégénérée. Les bases ambiguës sont remplacées par des bases correspondant à plusieurs possibilités selon le tableau 3.
15
Tableau 3 : Tableau des bases dégénérées selon la nomenclature de l’IUPAC (modifié par Patrick Gagné afin d’inclure la notation I et X) (bioinformatics.org)
IUPAC nucleotide code Base
A Adenine
C Cytosine
G Guanine
T (or U) Thymine (or Uracil)
R A or G Y C or T S G or C W A or T K G or T M A or C B C or G or T D A or G or T H A or C or T V A or C or G
N/I/X any base
. or - Gap
Lors de la synthèse de l’amorce, un mélange équimolaire des différentes possibilités est créé (exemple, si l’amorce est ATTCYCCT, il y aura 50% du mélange qui contiendra l’amorce ATTCCCCT et 50% qui contiendra l’amorce ATTCTCCT). Cette technique permettra de réduire le biais de sélection lors de l’amplification, mais demandera des programmes appropriés lors du traitement bio-informatique.
1.6 Architecture informatique
Il existe plusieurs types d’architecture pour les systèmes informatiques. Dans le cadre de ce projet, deux d’entre elles sont importantes : mémoire partagée et mémoire distribuée.
16
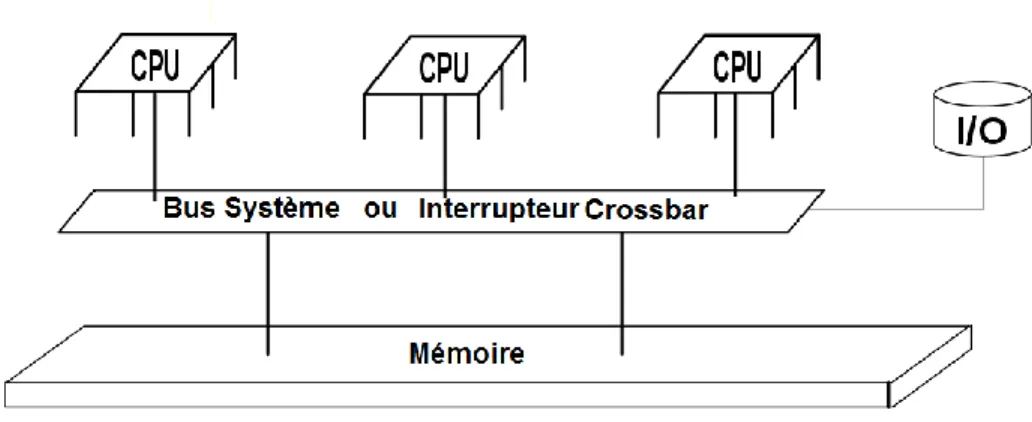
1.6.1 Mémoire partagée
L’architecture à mémoire partagée est probablement la plus commune; c’est l’architecture de pratiquement tous les ordinateurs domestiques sur le marché. Le terme partagé est utilisé dans ce contexte, car tous les processeurs de l’ordinateur partagent la même mémoire.
Figure 4 : Architecture à mémoire partagée (Semantic Scholar)
Cette architecture est avantageuse au niveau du développement de programme parallèle, car le partage de mémoire assure que chaque processeur peut accéder à la même information qu’un autre processeur sans étape de programmation supplémentaire. Le principal défaut de cette architecture est qu’elle est limitée en puissance. Pour le moment, une carte mère ne peut accueillir au plus que quatre Quadcore contenant chacun 16 processeurs logiques (c’est-à-dire aux maximum 64 processeurs logiques). Cette limitation n’est pas un problème en soi, car la plupart des outils n’ont pas besoin d’autant de puissance pour fonctionner efficacement. Toutefois, des programmes demandant une grande puissance de calculs (pour la dynamique moléculaire par exemple) ne pourront pas être exécutés sur ce genre d’architecture. Pour ce type d’application, il faut faire appel à une architecture alternative.
1.6.2 Mémoire distribuée
L’architecture à mémoire distribuée est l’architecture par excellence des supercalculateurs. Elle consiste en plusieurs ordinateurs à mémoire partagée
17
(communément appelés nœuds ou nodes) interreliés par de la réseautique de haute performance (ce qui forme la grappe ou cluster).
Figure 5 : Architecture à mémoire distribuée (Washington University in St.Louis)
Cette architecture est avantageuse au niveau de la performance, car il est possible d’effectuer une analyse en parallèle sur un nombre de cœurs important (certains supercalculateurs sont composés de plusieurs centaines de nœuds et donc de plusieurs milliers de processeurs). Étant à mémoire distribuée, chaque nœud est indépendant dans son exécution, il est donc nécessaire que les programmes utilisés sur un supercalculateur soient en mesure de tirer profit de cet environnement. Le principal défaut de ce type d’architecture est sa grande complexité; en effet, la manipulation du système est très différente d’un ordinateur conventionnel, par exemple, le lancement d’une tâche sur un supercalculateur demande normalement d’interagir avec l’ordonnanceur de tâches, qui gère l’allocation des ressources (principe inexistant sur un ordinateur conventionnel). De plus, le niveau de programmation nécessaire pour développer des programmes capables de fonctionner sur un supercalculateur est très élevé, car les librairies sont plus complexes, les principes plus abstraits et les erreurs beaucoup plus fréquentes. Ces points seront expliqués plus en détails dans la section 1.7.2.
18
1.7 Développement informatique
Dans le but de développer une nouvelle méthodologie d’analyse bio-informatique, il est nécessaire de maîtriser différents langages et différentes méthodes de programmation. Le choix du langage de programmation approprié repose sur plusieurs critères :
La performance du langage (un langage compilé comme le C++ sera plus efficace qu’un programme interprété comme Python)
Les opérations à effectuer (selon le type d’opérations, un langage peut être plus approprié qu’un autre. Exemple: des calculs statistiques seront plus faciles à réaliser en R plutôt qu’en C++)
La simplicité (certains langages, comme le C++, sont très complexes, autant au niveau syntaxique qu’au niveau algorithmique. Dans ces cas, un langage moins complexe permet de diminuer le temps de développement, surtout si le programme effectue des opérations simples)
La versatilité (certains langages plus spécialisés ne sont pas en mesure d’effectuer certaines opérations que des langages de plus bas niveau sont en mesure de faire).
La programmation séquentielle, bien que simple, n’est pas toujours la plus efficace. Il est possible d’obtenir un gain de performance important si certaines opérations sont implémentées de manière parallèle. Bien que cela augmente la complexité du développement, le calcul parallèle n’est pas négligeable pour obtenir une meilleure performance. De plus il est possible d’obtenir encore plus de performance si la parallélisation est effectuée sur plusieurs ordinateurs connectés en parallèle, il s’agit de la programmation distribuée
1.7.1 Langage de programmation
1.7.1.1 Bash
Le langage Bash est un langage propre aux systèmes de type UNIX/Linux, c’est le langage pour l’exécution de l’invite de commande Linux. Ce langage est très souvent utilisé dans la mise en place de pipelines, car il est très simple à
19
implémenter. En général, il suffit d’entrer les commandes dans un script Bash et ensuite d’exécuter ce script pour que le terminal lance chaque commande les unes à la suite des autres. Malgré sa simplicité, le Bash regorge de possibilités. Ce langage possède la plupart des capacités des autres langages de programmation de plus bas niveau (boucles, définition de fonctions, etc.). Le langage est cependant limité pour la programmation d’outils. Souvent le Bash fera office de contrôleur pour des programmes plus complexes, c’est-à-dire qu’il permettra aux programmes de fonctionner au bon moment, un peu comme un pipeline.
1.7.1.2 C++
Il n’est pas vraiment possible de parler de programmation sans parler de C++. Ce langage est un langage compilé, c’est-à-dire que l’exécution du programme nécessite de transformer le langage utilisé par les programmeurs en langage binaire afin que l’ordinateur puisse exécuter le code. La compilation est un avantage au niveau de la vitesse d’exécution, mais cela rend le développement des gros programmes plus difficile, car un programme imposant peut demander beaucoup de temps pour compiler et chaque modification du code nécessite une recompilation, ce qui n’est pas nécessairement un problème, mais peut rapidement le devenir lorsque le code source augmente en taille. Le principal défaut du C++ est sa grande difficulté d’implémentation, car ce langage utilise une syntaxe parfois ardue à comprendre et nécessite une connaissance approfondie de la programmation pour le maîtriser. La plupart des outils performants utilisent le C++ comme langage de prédilection.
Un avantage du C++ est le niveau de liberté qu’il donne à l’utilisateur; principalement au niveau de la gestion de la mémoire, car c’est l’utilisateur qui doit définir comment son programme interagit avec les différentes composantes de l’ordinateur. Mais cet avantage est aussi un défaut, car cela ajoute à la difficulté d’implémentation.
1.7.1.3 Python
Le langage Python (version 3) est un langage très populaire auprès des programmeurs. Contrairement au C++, le python est un langage interprété (et ne
20
nécessite donc pas d'être compilé). Ce qui signifie que chaque instruction est convertie en langage machine au fur et à mesure que les lignes de commandes défilent. L’absence de compilation améliore le temps de développement, mais cette amélioration est au détriment de la vitesse d’exécution, ce qui peut constituer un défaut de Python dépendamment du contexte.
La popularité de Python est aussi en raison de sa facilité d’implémentation (c’est d’ailleurs le langage utilisé pour former les nouveaux programmeurs). Malgré cette simplicité, Python reste un langage de bas niveau capable de réaliser n’importe quelle tâche bio-informatique. Un autre avantage de Python est la disponibilité d’une grande quantité de librairies bio-informatique (notamment biopython) qui facilitent grandement le développement d’outils.
Un autre défaut important de Python est sa difficulté d’implémentation au niveau du parallélisme, car ce langage est restreint par un mécanisme interne nommé le GIL (Global Interpreter Lock). Ce mécanisme permet de protéger les différents objets de Python en mémoire en empêchant plusieurs cœurs d’y accéder. Comme le système de gestion de la mémoire en Python n’est pas en mesure d’assurer la protection de la mémoire en situation multi cœur, ce mécanisme est nécessaire, et donc incontournable. Ceci constitue probablement le plus gros défaut de Python, car la seule manière d’effectuer du calcul parallèle en Python est d’utiliser le parallélisme par implémentation externe (voir section 1.7.2.3) ou par MPI (voir section 1.7.3).
1.7.1.4 R
Le langage R a été développé afin de faciliter les analyses statistiques dans tous les domaines. Depuis plusieurs années, ce langage est devenu très populaire en bio-informatique, surtout en raison de la grande importance des mathématiques dans les sciences de la vie. Le langage R a l’avantage d’avoir une importante communauté de développeurs permettant très souvent d’obtenir des réponses à différents problèmes sans avoir à programmer. R est cependant difficile d’approche pour les programmeurs chevronnés, car sa syntaxe est souvent très différente par rapport aux autres langages. De plus, le langage R est un langage
21
de plus haut niveau, c’est-à-dire qu’il y a certaines méthodes de programmation qui n’existent pas ou qui peuvent être très difficiles d’implémentation en R, notamment le calcul parallèle massif, qui sera discuté dans la section 1.7.3 « Programmation parallèle et distribuée ».
1.7.2 Programmation parallèle (architecture mémoire partagée)
Le calcul parallèle est une méthode d’amélioration de performance calculatoire très utilisée de nos jours. Depuis plusieurs années, les processeurs ont considérablement évolué; par rapport aux processeurs de l’époque (un seul cœur), les processeurs modernes sont capables de traiter une grande quantité d’information en utilisant leurs multiples cœurs en parallèle.
Afin de bénéficier des améliorations de performance, il est nécessaire de choisir une doctrine de programmation adaptée et de l’implémenter dans le programme en cours de développement. Il existe trois doctrines principales de programmation parallèle sur un ordinateur unique: la programmation pthread (ou manuelle), openMP (implémentation par instructions) et l’implémentation externe. Pthread et openMP sont des librairies C/C++, elles sont utilisées à titre d’exemple pour faciliter la compréhension.
1.7.2.1 Pthread (implémentation manuelle)
La doctrine de programmation pthread, ou programmation manuelle, consiste à utiliser une librairie de calcul parallèle (pthread dans ce contexte) et à développer le code du programme de manière parallèle. Cette méthode, bien que potentiellement très puissante, est aussi l’une des bêtes noires des programmeurs, car la gestion de chaque cœur doit être gérée par le programmeur. Dans ce cas, il y a beaucoup de pièges que le programmeur doit éviter; par exemple : les courses critiques. Ce type d’erreur survient quand un fil d’exécution tente d’accéder à une variable alors qu’un autre fil d’exécution tente d’écrire dans cette variable au même moment. Dans le meilleur cas, le programme subira un échec complet et retournera un message d’erreur, mais dans le pire des cas, le programme continuera de calculer, mais les calculs n’aboutiront pas aux bonnes valeurs. Ce
22
type d’erreur est souvent difficile à détecter et peut causer beaucoup de problèmes lors du développement d’outils.
Afin d’éviter les courses critiques, il est nécessaire que le programmeur implémente un contrôle d’accès aux ressources, c’est-à-dire une série d’instructions qui permette à un cœur de « verrouiller » une ressource afin d’y écrire sans risquer qu’un autre cœur y accède. Une autre façon d’éviter les courses critiques est par isolement complet des cœurs, c’est-à-dire que chaque processeur possède ses propres ressources et ces propres variables, indépendamment des autres cœurs.
Le principe de base de la programmation parallèle dans un contexte bio-informatique est illustré dans la figure 6.
Figure 6 : Principe du calcul parallèle de base
La figure 6 représente le cas simple ou parfait de la programmation parallèle. Le cas le plus simple est que le résultat du traitement d’une donnée est obtenu
23
indépendamment du traitement d’une autre donnée. Ce type de cas permet notamment de séparer le jeu de données d’entrée en autant de parties qu’il y a de cœur disponible.
1.7.2.2 OpenMP (implémentation par instruction)
La doctrine de programmation OpenMP (ou implémentation par instruction) (Dagum & Menon, 1998) a été inventée en 1998 afin de faciliter le développement de programmes multifilaires. Pour développer un programme parallèle avec OpenMP, le programmeur fait appel à une série d’instructions sous la forme « #pragma omp parallel instruction ». Ces instructions sont appliquées directement sur le bloc de code suivant, par exemple une boucle.
La programmation OpenMP rend le développement parallèle beaucoup plus simple en rendant transparentes les méthodes parallèles pour le programmeur. De plus, un ordinateur n’ayant pas la capacité OpenMP sera quand même en mesure d’exécuter le programme (les instructions seront tout simplement ignorées par le compilateur). Cependant, cette transparence est obtenue au détriment de la clarté et du contrôle, car il est difficile (voire parfois impossible) de savoir comment OpenMP effectue la parallélisation. Il est possible qu’une instruction (ou une série d’instructions) ne soit pas bien adaptée ou soit mal placée, ce qui résultera souvent en des erreurs dans l’analyse ou en une chute drastique de la performance du programme.
1.7.2.3 Implémentation externe
Une parallélisation externe consiste à utiliser un programme n’ayant pas de capacité parallèle et de la lui donner sans modification sur son code. La méthode de base est de démarrer plusieurs instances du programme en question et d’ajuster leurs paramètres d’entrée afin que chaque instance travaille sur une partie différente des données. Ce genre de parallélisme demande souvent des programmes secondaires afin d’effectuer les traitements nécessaires sur les données (des programmes permettant de diviser le fichier d’entrée ou de concaténer les fichiers de sortie, par exemple). L’avantage de ce type de parallélisme (lorsqu’il est applicable) est qu’il peut s’adapter à n’importe quel
24
système sans connaissance exhaustive en programmation parallèle. De plus, il est possible de profiter de la puissance d’une architecture à mémoire distribuée et de lancer un grand nombre d’instances sur plusieurs nœuds de calcul.
1.7.3 Programmation distribuée (architecture à mémoire distribuée)
La programmation distribuée est fondamentalement différente de la programmation parallèle standard. Dans une architecture à mémoire distribuée (comme mentionné dans la section 1.6.2), les différents nœuds de calculs ne partagent pas la même mémoire. Afin de synchroniser les processeurs et leur fournir les données nécessaires aux calculs, il est nécessaire d’avoir des méthodes de communication adaptées à ce type d’architecture.
L’interface par passage de message (ou MPI) (Gropp, Gropp, Lusk, Lusk, & Skjellum, 1999) est une librairie de fonctions permettant la communication entre les différents processeurs d’une architecture à mémoire distribuée. Dans cette situation, le « message » peut être n’importe quel type de données, des séquences ADN par exemple. La figure 7 suivante illustre le fonctionnement d’une analyse utilisant le passage par message.
25 Figure 7 : Transmission de message par MPI
Il est possible d’utiliser le principe de passage par message sur un ordinateur à mémoire partagée afin d’éviter la réécriture du programme, mais cette compatibilité accrue sera au détriment de la performance, car le passage de message est associé à une latence élevée (le temps de transmission du message).
L’avantage de la programmation distribuée est qu’il n’y a pas de risque de course critique, car la plupart des fonctions de transmission de message sont bloquantes, c’est-à-dire que le programme sur le nœud transmettant et recevant est automatiquement mis en attente jusqu’à la fin de la transmission. Une fois le traitement effectué sur chaque nœud, il est nécessaire que les résultats soient transmis par ces mêmes nœuds vers un nœud maître afin d’être concaténés et retournés.
26
Compte tenu de l’évolution très rapide des technologies de séquençage, les jeux de données deviennent de plus en plus massifs et complexes. Dans cette optique, il certain que les outils actuellement disponibles ne seront pas en mesure d’effectuer le travail dans le futur; c’est pourquoi il est nécessaire que de nouveaux outils et de nouvelles méthodologies soient constamment développés. La méthodologie développée dans le cadre de ce mémoire s'attache à respecter les concepts clefs d'une bonne méthodologie bio-informatique. Ces concepts sont :
Efficacité
L’efficacité de la méthodologie est premièrement basée sur la vitesse d’exécution: plus le programme s’exécute rapidement, plus il sera efficace. Afin d’atteindre des niveaux de vitesse supérieure, des techniques de calcul parallèle ont été utilisées lors du développement des outils les plus lourds. La sélection des programmes externes a aussi été faite en tenant compte de leur efficacité.
L’efficacité en informatique peut aussi être interprétée au sens de la mémoire vive (RAM). Un programme efficace en mémoire vive ne surchargera pas celle-ci inutilement. Dans le cadre du projet, cet aspect de l’efficacité n’est pas prioritaire, le matériel informatique utilisé lors du développement possédant assez de mémoire vive pour rendre le développement de méthodes légères en RAM inutile. Certaines méthodes ont cependant été développées dans cette optique pour les futures implémentations de la méthodologie.
En plus de l’efficacité au niveau de la vitesse d’exécution et de la RAM, la parallélisation d’un algorithme est aussi associée à une valeur d’efficacité. Un programme efficace au niveau du parallélisme est en mesure de profiter d’un gain de vitesse proportionnel au nombre de processeurs utilisés.
Données massives
Il est de plus en plus difficile de parler de séquençage ADN sans parler de données massives. Les données massives sont souvent définies par des données dont la taille est un problème en soi. Ce type de données demande souvent des méthodes particulières pour être traité, car leur taille importante rend la plupart des
27
programmes standards non fonctionnels. De plus, les données massives causent surtout des problèmes au niveau de la mémoire vive, car il est normalement impossible d’importer un jeu de données massif directement dans la mémoire vive d’un système. Les données massives, dans le cadre de ce projet, n’ont pas la même signification, car les jeux de données en metabarcoding ne sont pas encore assez imposants pour être de véritables données massives (un jeu de données de 20 Go n’est pas considéré comme massif au sens propre). Cependant, la combinaison de plusieurs jeux de données afin de réaliser une analyse combinée peut correspondre à des données massives.
La méthodologie doit donc être en mesure d’analyser des données de l’ordre des centaines de millions de séquences dans un temps raisonnable.
Adaptabilité et extensibilité
Il est désirable que la méthodologie possède une bonne adaptabilité, car les projets de recherche en metabarcoding sont nombreux et variés. Un programme adaptable doit être en mesure de considérer plusieurs possibilités de projets et permettre à l’utilisateur de choisir quel type d’analyse lui convient le mieux. Le développement du projet a été réalisé principalement dans un contexte de recherche de pathogènes forestiers exotiques, de type fongique, mais il est possible pour l’utilisateur de réaliser une analyse pour des COI d’insectes ou des études écologiques.
En plus de l’adaptabilité au niveau des projets de recherche, le programme doit être adaptable au niveau des étapes d’analyse composant sa structure (plusieurs choix sont disponibles selon l’étape, et l’utilisateur peut choisir le programme qui lui convient dans la liste de possibilités).
Le programme doit être extensible, c’est-à-dire qu’il ne doit pas être trop difficile d’ajouter des étapes de traitement ou des modules au programme. Cela n’est pas une priorité pour le moment, mais certaines pratiques de programmation utilisées dans le développement du pipeline ont été choisies dans cette optique.
28
Simplicité de mise en place
L’obstacle majeur de beaucoup de pipelines et boîtes à outils est qu’il peut être parfois ardu de les installer, surtout en raison du nombre élevé de dépendances que chaque outil nécessite pour fonctionner. À titre d’exemple, Qiime est l’une des boîtes à outils les plus difficiles à installer en raison des dépendances.
Un pipeline utilisant un très petit nombre de dépendances est désirable. Les seules dépendances nécessaires sont les programmes externes au projet. Tous les programmes développés dans le cadre de ce projet ont été implémentés en utilisant presque exclusivement des librairies de base des langages de programmation, la seule exception étant la librairie tclap (Smoot, 2003), une libraire de gestion des arguments de programme en C++, mais elle est fournie avec le code source de chaque programme qui l’utilise.
Le pipeline doit aussi avoir un script de configuration, d’installation et de vérification automatisé afin de faciliter toutes ses étapes de mise en place.
Facilité d’utilisation
Tout pipeline vise à rendre les analyses plus faciles à réaliser pour les utilisateurs débutants. Cette simplification vient toutefois limiter le potentiel des pipelines en question, car toute étape de simplification est souvent au détriment de la flexibilité. Il est bon d’avoir une utilisation simple, principalement grâce aux analyses prédéfinies et optimisées, mais aussi sans trop sacrifier la flexibilité du programme en permettant à l’utilisateur de modifier aisément les paramètres de chaque analyse et de réutiliser ces changements dans les analyses subséquentes.
Objectifs du projet
L’objectif principal du projet est de développer une méthodologie d’analyse bio-informatique capable de répondre aux demandes de divers types de projet de recherche pour des données de type Illumina/Ion Torrent dans un contexte de metabarcoding. Elle se doit aussi d'être efficace, capable d’analyser des jeux de données imposants, extensible et adaptable, simple d’installation avec peu de dépendances et facile d’utilisation.
29
2 Chapitre 2 : Matériel et méthodes
2.1 Données
Pour le développement de la méthodologie, 11 jeux de données de metabarcodage fongiques basés sur l’ITS1 ont été utilisés. Ces jeux de données proviennent de sources et de projets variés. Afin d’évaluer la capacité du pipeline à traiter des données massives, la plupart de ces jeux de données ont été combinés afin de créer un jeu de données de grande taille (la procédure de génération des jeux de données d’évaluation est décrite dans la section 2.2)
Comme une grande partie de ces jeux de données appartiennent à d’autres projets, les résultats des analyses ne sont pas divulgués dans le cadre de ce projet, seuls les données de performances et les décomptes de séquences le sont.
2.1.1 Préparation des données d’évaluation
Pour évaluer correctement ADAPTI, 11 jeux de données environnementales d’ITS1 ont été utilisés pour les différents tests (tableau 4).
Tableau 4 : Sommaire des jeux de données uniques utilisé pour évaluer ADAPTI
Ces jeux de données ont été regroupés de manière progressive pour obtenir des dimensions progressives allant de 21Go à 305Go (tableau 5)
Dataset # Éch Taille Compressé (G) Taille Décompressé Compte Séquence F+R Compte Séquences Complètes
Combinaison1 139 6.4 21.017 34257038 17128519 Combinaison2 191 6.5 22.297 36287596 18143798 Combinaison3 101 7.6 24.257 39498822 19749411 Combinaison4 97 12 30.52 50003520 25001760 Combinaison5 115 13 31.65 51712928 25856464 Combinaison6 185 9.7 28.86 47028512 23514256 Combinaison7 128 13 32.14 52332466 26166233 Combinaison8 155 9.3 27.336 44489614 22244807 Combinaison9 153 12 30.051 48920348 24460174 Combinaison10 155 9.3 27.036 44487416 22243708 EO_Dataset 128 11 29.469 48042232 24021116
30
Tableau 5 : Composition des différentes combinaisons de jeux de données pour tester ADAPTI
Dans le contexte de l’évaluation de la performance multifilaire, l’utilisation de données à dimensions progressives n’est pas possible, car les temps d’analyses à complexité élevée sont beaucoup trop élevés. Pour pallier à ce problème, seuls deux sous-groupes ont été utilisés (Combinaison 4 du tableau 4, appelé 25M et la sommation de tous les jeux de données, dernière ligne du tableau 5 appelé 250M). Tous les jeux de données ci-dessus proviennent de projets de recherche en détection de pathogènes forestiers fongiques et de projets d’études en écologie fongique des sols. Ce sont donc des jeux de données ITS environnementaux dont les provenances sont multiples.
Bien que ces jeux de données soient adéquats pour évaluer la performance d’ADAPTI, il n’en demeure pas moins que leur nature environnementale les rend inadéquats pour évaluer la validité de la méthode, leur composition étant inconnue. Pour correctement évaluer la validité des résultats, un jeu de données normalisé (appelé communément Mock) a été téléchargé sur NCBI (Tableau 6); ils proviennent d’une étude comparant l’efficacité des différents codes-barres pour les champignons (Hoggard et al., 2018).
Nombre de jeu de données # Échantillons # Séquences Taille compressé (Go) Taille décompressé (Go)
1 139 17128519 6.4 21.017 2 330 35272317 12.9 43.314 3 431 55021728 20.5 67.571 4 528 80023488 32.5 98.091 5 643 105879952 45.5 129.741 6 828 129394208 55.2 158.601 7 956 155560441 68.2 190.741 8 1111 177805248 77.5 218.077 9 1264 202265422 89.5 248.128 10 1419 224509130 98.8 275.164 11 1547 248530246 109.8 304.633
31 Tableau 6 : Composition du jeu de donnée Mock
2.2 ADAPTI
ADAPTI ou Automatic Dna Analysis & Processing Tool for Illumina est le pipeline développé dans le cadre de ce projet. ADAPTI est une continuité du pipeline ADAPT développé pour le pyroséquençage 454 (pipeline non publié). Mis à part le nom, ADAPTI est complètement différent de son prédécesseur, autant au niveau des étapes d’analyses fondamentalement différentes en raison du changement de technologie de séquençage qu’au niveau des méthodes de programmation implémentées dans sa structure. Les deux pipelines utilisent le langage Bash comme langage d’implémentation, mais les fonctions ainsi que les méthodes de programmations utilisées sont différentes.
2.3 Processus d’essai
ADAPTI a été testé de manière interne et externe. Les tests internes consistent en une vérification de son efficacité multifilaire au niveau du temps et de la mémoire vive utilisée par rapport au nombre de séquences en entrée. Ces tests permettent d’évaluer si ADAPTI répond aux objectifs du projet.
Type de séquençage Illumina Paired-End Nombre Échantillons 24
Nombre Séquences 3667939
Code-Barre ITS 1
Taille compressé 0.963 Go Taille décompressé 3.75 Go
32
Les tests externes consistent à effectuer la comparaison de la performance d’ADAPTI aux autres programmes et boîtes à outils. Les programmes suivants ont testés :
AAFC Snakemake-Qiime (version développée par Agriculture Canada (Korol & Lowe, 2017)
Pipecraft (Anslan, Bahram, Hiiesalu, & Tedersoo, 2017) Dada2 (Callahan et al., 2016)
Les critères d’évaluation sont a) la performance au niveau de la vitesse d’exécution b) la performance au niveau de la mémoire vive c) La capacité à traiter une quantité massive de données en un temps raisonnable et d) le nombre d’espèces théoriques, noté ici OTU ou unité taxonomique opérationnelle, sortant après les traitements. Des tests ont aussi été effectués sur la capacité à détecter des pathogènes forestiers exotiques dans une série d’échantillons environnementaux, car ADAPTI a été développé à cet effet à la base. Chaque programme a été installé sur un supercalculateur à mémoire distribuée et les analyses ont été effectuées sur un seul nœud (les programmes ci-dessus ainsi qu’ADAPTI n’ayant pas la capacité d’utiliser plusieurs nœuds de calculs).
2.4 Étapes de traitement
Dans des projets de metabarcoding, les pipelines regroupent très souvent des étapes similaires : le démultiplexage, le réassemblage, le nettoyage, le regroupement (clustering) en unités taxonomiques opérationnelles (OTUs) et l’assignation taxonomique des OTUs par blast.
2.4.1 Démultiplexage
Le démultiplexage consiste à extraire l’information des index de séquençage pour déterminer quelle séquence appartient à quel échantillon. Cette étape est cependant de moins en moins nécessaire, car les améliorations des plateformes de séquençage permettent d’automatiquement ségréger les séquences brutes par échantillons avant de rendre les données disponibles aux utilisateurs. C’est d’ailleurs pourquoi ADAPTI ne contient pas de module pour travailler avec des