HAL Id: hal-02806078
https://hal.inrae.fr/hal-02806078
Submitted on 6 Jun 2020HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Etude de l’influence de l’hydromorphie des sols sur les
communautés microbiennes à l’échelle d’un versant
agricole
Quentin Audouit
To cite this version:
Quentin Audouit. Etude de l’influence de l’hydromorphie des sols sur les communautés microbiennes à l’échelle d’un versant agricole. [Stage] France. Université de Bourgogne (UB), FRA. 2013, 28p. �hal-02806078�
Quentin AUDOUIT
Master 1 Sciences de l’Environnement
Maîtres de stage :
Lionel Ranjard
Cédric Le Guillou
RAPPORT DE STAGE
ETUDE DE L
'
INFLUENCE DE
L
'
HYDROMORPHIE DES SOLS SUR LES
COMMUNAUTES MICROBIENNES A
L
'
ECHELLE D
'
UN VERSANT AGRICOLE
Remerciements
Je tiens dans un premier temps à remercier Mr Lionel Ranjard, directeur de l’équipe BioCom, pour m’avoir proposé ce stage et accueilli au sein de l’UMR Agroécologie.
Je remercie tout particulièrement mon maitre de stage Cédric Le Guillou qui a été présent à mes côtés pendant toute la durée de mon étude sur le transect de Naizin. Egalement pour sa pédagogie et son aide précieuse en matière de bibliographie et d’analyses statistiques.
J’adresse mes sincères remerciements à Virginie Nowak pour la formation qu’elle m’a apportée sur les manipulations en laboratoire malgré mes faibles connaissances en biologie ainsi que pour son aide sur l’étude du transect « piégeométrique ».
Merci à Mélanie Lelièvre pour la formation, le soutien moral et l’aide fournis en matière de PCR et d’analyses LiCor.
Je remercie aussi Emmanuel Tete d’Agrocampus Ouest de Rennes pour nous avoir accompagné sur le terrain et fourni les résultats extraits de sa thèse.
Je salue également tous les membres de l’équipe BioCom pour leur accueil, leur aide et la bonne ambiance de travail.
Enfin, je remercie le séquenceur LiCor, merveilleux appareil, sans quoi la fin de mon étude n’aurait pas eu la même intensité.
Sommaire
Avant-propos...………1
1. Présentation de l’organisme d’accueil : l’INRA………1
2. Le site INRA de Dijon………...1
3. L’UMR Agroécologie………1 4. L’équipe Biocom………...1 5. La plateforme GenoSol……….2
Introduction………3
I. Le projet MOSAIC……….4
1. Objectifs généraux………..42. Objectifs spécifiques du stage……….5
II. Matériel et méthodes………...6
1. Présentation du site d’étude………6
a) Le bassin versant de Naizin b) Le transect piézométrique 2. Campagne d’échantillonnage au terrain……….8
3. Analyses en laboratoire……….10
a) Conditionnement des échantillons pour le conservatoire b) Quantification de l’ADN microbien c) Caractérisation de la structure génétique des communautés microbiennes
III. Résultats et Discussion………..19
1. Caractérisation du régime hydrique des sols………..19
2. Profil de flux de respiration des sols………..20
3. Quantification de l’ADN microbien………21
4. Caractérisation de la structure génétique des communautés microbiennes………..22
IV. Conclusion……….25
Liste des illustrations
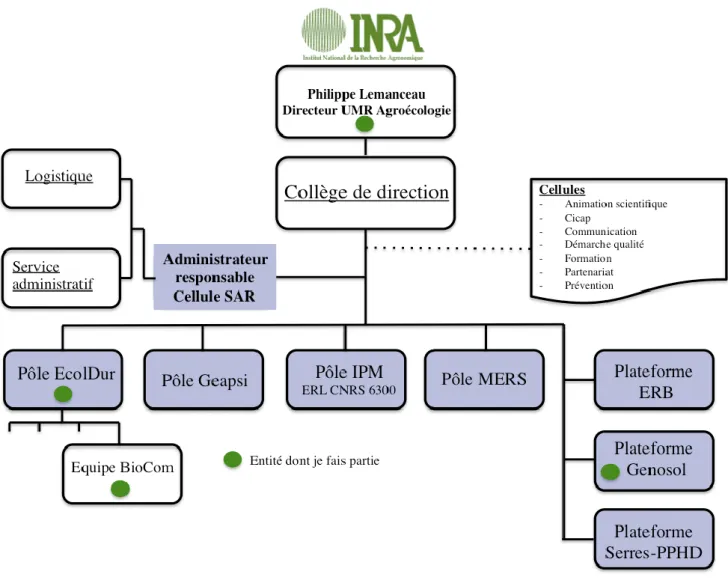
Figure 1 : Organigramme de l'UMR Agroécologie ... 2
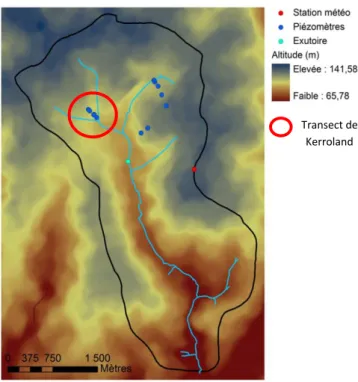
Figure 2 : Localisation du site de Naizin ... 6
Figure 3 : Vue aérienne du bassin versant de Naizin ... 6
Figure 4 : Carte d'occupation des sols du bassin versant ... 6
Figure 5 : Carte topographique ... 7
Figure 6 : Photo aérienne et emplacement des sites de prélèvement ... 8
Figure 7 : Photographie de la zone d'étude ... 8
Figure 8: Prélèvement à la tarière ... 9
Figure 9 : Prélèvements à la tarière dans la parcelle de blé ... 9
Figure 10 : Lyophilisateur ... 10
Figure 11 : Tamisage des échantillons ... 10
Figure 12 : Congélateurs du conservatoire GenoSol ... 10
Figure 13 : Quartage des échantillons ... 10
Figure 14 : Agitateur FastPrep ... 11
Figure 15 : Lysats d'ADN "sale" obtenus ... 11
Figure 16 : Etuve ... 12
Figure 17 : Electrophorèse sur gel d'agarose ... 13
Figure 18 : Cuves pour traitement au BET ... 13
Figure 19 : Armoire UV et caméra ... 13
Figure 20 : Image de gel après migration ... 14
Figure 21 : Pré-purification par PVPP (T.Regnier, 2010) ... 15
Figure 22 : Purification GeneClean (T.Regnier, 2010) ... 15
Figure 23 : Schéma synthétique technique PCR-ARISA ... 16
Figure 24 : Déroulement d'une PCR ... 17
Figure 25 : Système d'électrophorèse verticale du LiCor ... 18
Figure 26 : Séquenceur LiCor ... 18
Figure 27 : Suivi temporel de l'humidité volumétrique des sols ... 19
Figure 28 : Humidité pondérale des sols lors de la campagne de prélèvement ... 20
Figure 29: Suivi temporel de la respiration des sols ... 20
Figure 30 : Quantification de l'ADN microbien ... 21
Figure 31 : ACP des profils ARISA des bactéries ... 23
Glossaire
ACP : Analyse en Composantes Principales ADN : Acide DésoxyriboNucléique.
ARISA : Automated Ribosomal Intergenic Spacer Analysis. ARNr : Acide RiboNucléique ribosomique.
BET : Bromure d’Ethidium.
CO2 : Dioxyde de carbone ou Gaz carbonique.
EDTA : Acide Ethylène Diamine Tétra-Acétique.
EPST : Établissement Public à caractère Scientifique et Technologique. H20 UP : Eau Ultra Purifiée.
MOS : Matière Organique du Sol. NaCl : Chlorure de sodium.
ORE : Observatoire de Recherche en Environnement. PCR : Polymerase Chain Reaction.
qPCR : Qauntitative Polymerase Chain Reaction. PVPP : Poly-Vinyl Poly-Pirrolidone.
RBV : Réseau de Bassins Versants.
RISA : Ribosomal Intergenic Spacer Analysis. SDS : DodecylSulfate de Sodium.
SOERE : Systèmes d’Observation et d’Expérimentation pour la Recherche en Environnement.
UMR : Unité Mixte de Recherche. UV : Rayons Ultraviolets.
1
Avant-propos
1. Présentation de l’organisme d’accueil : L’INRA
L'Institut National de la Recherche Agronomique (INRA) est un organisme français de recherche en agronomie fondé en 1946, placé sous le statut d’Établissement public à caractère scientifique et technologique (EPST), et sous la double tutelle du ministère chargé de la Recherche et du ministère chargé de l’Agriculture. Ses recherches concernent trois domaines fortement imbriqués : l'alimentation, l'agriculture et l'environnement.
2. Le site INRA de Dijon
Le site de Dijon existe depuis la création de l’institut. Il est porteur de 4 thématiques principales : « Des qualités sensorielles des aliments aux comportements alimentaires », « Communautés végétales et microbiennes des agro-écosystèmes : fonctionnement, interactions, durabilité des systèmes agricoles », « Territoire et développement » et « Écologie des systèmes aquatiques et microbien ». Ces recherches sont menées conjointement avec l’Université de Bourgogne, l’Agrosup (Institut national supérieur des sciences agronomiques, de l’alimentation et de l’environnement) et le CNRS (Centre National de la Recherche Scientifique).
3. L’UMR Agroécologie 1347
Cette UMR (Unité Mixte de Recherche) récente est issue de la fusion des anciennes UMR BGA (Biologie et Gestion des Adventis), MSE (Microbiologie du Sol et de l’Environnement) et PME (Plantes, Microbes, Environnement). Elle réunit donc logiquement les activités et axes de recherche scientifique des 3 UMR précédentes. J’ai réalisé mon stage au sein de l’UMR Agroécologie, dans l’équipe BioCom (figure 1).
4. L’équipe BioCom
Cette équipe comporte 8 membres permanents dont l’animation et la direction scientifique est assurée par Lionel Ranjard. L’équipe BioCom « Distribution spatiale, dynamique et traduction fonctionnelle de la biodiversité des communautés microbiennes telluriques » a pour but :
i. De déterminer les facteurs naturels et anthropiques qui déterminent la distribution spatiale et la diversité des communautés microbiennes du sol.
ii. D’étudier les liens entre la biodiversité microbienne d’un sol et son fonctionnement. iii. D’évaluer les modes d’usages des sols selon une approche environnementale.
2
5. La plateforme GenoSol
L’UMR MSE a créé en 2008 la plateforme GenoSol. Centre de ressource unique en France et en Europe, son objectif est de fournir une structure logistique assurant l’acquisition, la conservation, la caractérisation et la mise à disposition des ADN de sols issus des échantillonnages de grande envergure. Cette plateforme a pour mission d’établir un véritable référentiel des caractérisations moléculaires microbiennes sur les sols ainsi que des interprétations de ces analyses à grande échelle. Ces analyses doivent permettre de conférer un caractère plus générique aux études portant sur l’évaluation des ressources génétiques des sols et la traduction de ces ressources en services agro-écosystémiques.
3
Introduction
Le sol est une ressource essentielle à l’humanité, il est le support de l’activité anthropique pour la construction mais aussi et surtout pour la production alimentaire par l’agriculture. Comprendre le fonctionnement du sol est un enjeu majeur de la science agronomique moderne à l’heure où se posent les questions de développement durable et la nécessité de répondre à la demande mondiale en nourriture qui devrait augmenter de 70% en 2050 (Dequiedt et al., 2011 ; FAO, 1998 ; FAO, 2009). Le défi des agrosystèmes pour les années à venir est donc d’augmenter la productivité tout en préservant les ressources naturelles et en réduisant l’impact sur l’environnement (sol, eau, air et biodiversité). Ce défi doit, en plus, se réaliser dans un contexte de changements globaux qui menacent la durabilité des systèmes agricoles.
Actuellement, la diminution des stocks de matière organique des sols (MOS) est considérée comme une menace majeure pour la durabilité des agrosystèmes (Commission Européenne, 2002). En effet, la MOS est capital pour la fertilité des sols (Tiessen et al., 1994) car elle affecte à la fois la capacité de production végétale et les fonctions environnementales du sol (Lal, 2009). Ces fonctions environnementales englobe notamment son rôle de milieu tampon dans les transferts de nutriments (NO3-, carbone organique dissous), la fonction de « puits » de carbone dans la régulation des émissions des gaz à effet de serre (Johnston et al., 2004) et le rôle d’interface dans les dynamiques des cycles biogéochimiques d’éléments majeurs (C, N, P).
Les recherches menées ont considérablement amélioré la connaissance des propriétés et de la dynamique des MOS. Cependant, une des difficultés majeures pour parvenir à prédire les changements à long-terme de la dynamique des MOS est de comprendre les potentiels effets additifs et les interactions dans des agrosystèmes présentant des pratiques culturales très variées (Rees et al. 2005). Pour combler ce manque de connaissance, il est nécessaire d’entreprendre une évaluation plus intégrée de la dynamiques des MOS (Schmidt et al., 2011). Ainsi, l’échelle du paysage est considérée comme pertinente pour évaluer les interactions entre les facteurs naturels et anthropiques qui contrôlent la dynamiques des MOS et pour aborder de façon combinée l’impact des MOS sur la productivité agricole et les fonctions environnementales du sol (Viaud et al., 2010).
Le rôle majeur des communautés microbiennes dans la dynamique des MOS est depuis longtemps reconnu (Swift et al., 1979). Les communautés microbiennes ont par ailleurs la capacité de réagir rapidement aux stress environnementaux affectant le sol ce qui en fait un bio-indicateur pertinent de la qualité du sol (Bressan et al., 2008). Cependant, elles ne sont pas explicitement prises en compte dans les modèles actuels de dynamique de la MOS. Un changement de composition des communautés microbiennes dues aux changements climatiques pourraient avoir des conséquences sur la dynamique des MOS. Cela pourrait partiellement expliquer les différences de prédiction observées sur l’évolution des MOS en contexte de changement climatique.
4
(Dumont et Murrell, 2005 ; Hättenschwiler et al., 2005 ; Morales et Holben, 2011). Les récents développements méthodologiques permettent d’étudier la diversité microbienne du sol sans à priori (Ranjard et al., 2000 ; Maron et al., 2011) et à grande échelle ce qui permet d’intégrer l’hétérogénéité du milieu et la distribution spatiale des communautés microbiennes ce qui in fine améliorent la compréhension du lien entre diversité microbienne et services écosystémiques (Martiny et al., 2006).
C’est dans cette thématique que se positionne le projet MOSAIC, plus précisément son axe de recherche « Quantifier la variabilité et co-variabilité de l'organisation existante des systèmes de culture, les facteurs environnementaux et les stocks de MOS, à l’échelle d’un paysage agricole» pour lequel l’équipe BioCom et la plateforme Genosol dans lesquels j’ai effectué mon stage apportent leur expertise en matière de microbiologie du sol.
I. Le projet MOSAIC
(Approche à l’échelle du paysage de la dynamique des Matières Organiques des Sols, dans des systèmes Agricoles Intensifs liés à l’élevage et dans un contexte de Changements globaux)
1) Objectifs généraux
L’objectif général du projet est d’étudier (i) la dynamique des MOS à l’échelle d’un paysage
agricole et (ii) l’impact de changements climatiques sur cette MOS :i. Le projet se focalise sur la quantification et la compréhension de l’hétérogénéité des dynamiques des MOS à l’échelle d’un paysage présentant des systèmes de cultures très variés.
ii. La notion de changement climatique englobe les variations de température et de précipitations. Dans ce projet, le paysage étudié présente des gradients de régime hydrique des sols allant des sols bien drainés sur les hauts versants jusqu’aux sols saturés temporairement ou en permanence dans les bas de versants. L’étude se concentre sur l’impact du changement des précipitations (ayant pour effet une altération des régimes hydriques des sols) sur les dynamiques des MOS à l’échelle du paysage.
Les objectifs spécifiques à la microbiologie des sols attribués à l’équipe BioCom et à la plateforme GenoSol sont l’établissement du lien entre évolution des MOS, systèmes de culture, hétérogénéités du milieu et diversité microbienne ainsi que la détermination de la hiérarchie des processus et facteurs contrôlant l’activité microbienne et la dynamique de la MOS à l’échelle du paysage.
5
Une finalité de ce projet est de déterminer, à l’échelle du paysage, quels systèmes de culture associés à quelle gestion de la fertilisation et à quels régimes hydriques (c’est-à-dire l’emplacement dans le paysage) sont les plus adaptés pour une productivité agricole qui préserve l’environnement. Il sera ainsi possible de proposer une réorganisation spatio-temporelle des systèmes culturaux pour une gestion optimisée du paysage agricole.
2) Objectifs spécifiques du stage
Le stage au sein de la plateforme GenoSol s’inscrit dans le projet MOSAIC, dans le volet sur l’influence des changements globaux sur la dynamique des MOS. Il a pour but d’appréhender l’influence du gradient d’hydromorphie sur les communautés microbiennes du sol.
Pour évaluer l’influence du gradient d’hydromorphie, un transect sur le versant agricole du site de Naizin a été étudié. Ce site d’étude est instrumenté (piézomètres) dans le cadre de projets de l’Observatoire de Recherche en Environnement (ORE) AgrHys. Les échantillons ont été prélevés en 3 points de la pente en fonction de niveau d’humidité du sol attendu : en haut de versant (régime hydrique de sol bien drainé), en milieu de pente (régime hydrique alternant saturation et désaturation) et en bas de pente (régime hydrique ennoyé durant l’hiver et proche de la nappe).
Durant le stage, l’objectif a donc été d’évaluer l’influence de l’hydromorphie sur l’abondance microbienne et la structure génétique des communautés bactérienne et fongique.
6
II. Matériel et méthodes
1.
Présentation du site d’étude
a) Le bassin versant de Naizin
Le site retenu pour le projet MOSAIC est un petit bassin versant de 12 km² (Morbihan, Bretagne) (figures 2 et 3) où l’activité agricole est importante et représentative des bassins versants bretons. Le bassin porte le nom de la commune principale, Naizin.
Le climat est typique de l’Ouest de la France, c’est-à-dire un climat tempéré et océanique. Les agriculteurs y pratiquent une polyculture-élevage intensive : 85% de la surface de bassin sont dédiés à l’agriculture (figure 4).
Rennes
Naizin
Figure 2 : Localisation du site de Naizin Figure 3 : Vue aérienne du bassin versant de Naizin
7
Transect de Kerroland
Les pratiques agricoles sont associées à d’importants apports organiques (tels que l’épandage de lisier de porc) et azotés (fertilisation minérale), ce qui impacte les cycles de l’azote (en très fort excès dans les sols et les eaux de Bretagne) et du carbone organique.
Pour ces raisons, le site fait l’objet d’un suivi scientifique important depuis les années 1990. En effet, il fait partie des 30 ORE (Observatoire de Recherche en Environnement) en France et plus précisément de l’ORE AgrHys (temps de réponse dans les agro-hydrosystèmes). Il fait également partie, au niveau hydrogéochimie, du SOERE (Systèmes d’observation et d’expérimentation pour la recherche en environnement) et du réseau RBV (réseau de bassins versants). Grâce à ces nombreux cadres d’étude sur le site, l’historique du site est bien renseigné pour le projet MOSAIC et permet des comparaisons sur une vingtaine d’année, par exemple pour les stocks de carbone organique de ces sols.
b) Le transect piézométrique de Kerroland
L’étude réalisée lors du stage se situe sur un transect piézométrique mis en place par E.Tete, doctorant à Agrocampus Ouest (Rennes), dans le cadre de sa thèse sur la mesure d’émissions de CO2 des sols.
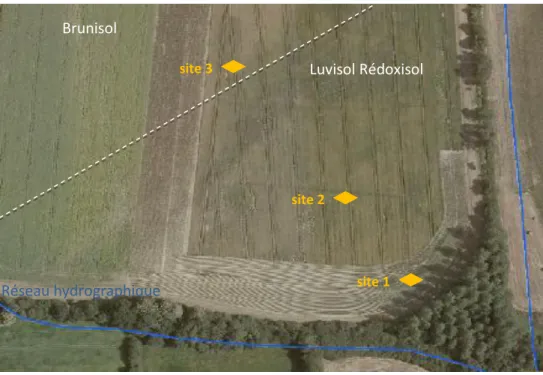
Le site d’étude est un versant agricole qui se situe au lieu-dit de Kerroland (figure 5). On y distingue une juxtaposition de deux domaines pédologiques :
-un domaine de sol bien drainé caractérisé par un Brunisol luvique de plus de 80 cm de profondeur, situé en haut de versant et dont l’horizon A n’est jamais saturé au cours de l’année.
-un domaine de sol hydromorphe en bas de versant caractérisé par un Luvisol-Rédoxisol faiblement dégradé sur altérite hydromorphe de 40-60 cm de profondeur. Le sol de mi- versant subit pendant l’hiver des alternances saturation-désaturation et celui de bas de versant est saturé de façon permanente durant l’hiver.
8 Réseau hydrographique Luvisol Rédoxisol Brunisol site 3 site 2 site 1
La photographie aérienne suivante nous montre la disposition des 3 points de prélèvement de notre étude en haut, milieu et bas de versant (figure 6) :
Par ailleurs les données de texture et de carbone des sols (sur 20 cm de profondeur) ont été défini à partir des travaux de thèse de M. Zida (Structure et fonctionnement hydrique d'un système pédologique armoricain - Rôle de l'espace poral et de l'agencement des horizons, 1998) :
Haut de versant : argile 17.5%, limons fins 34.5%, limons grossiers 36.0%, sables fins 7.3%, sables grossiers 4.7%. Teneur en C : 17.8 g/kg
Bas de versant : argile 18.7%, limons fins 35.1%, limons grossiers 30.0%, sables fins 8.5%, sables grossiers 7.7%. Teneur en C : 28.6g/kg
2. Campagne d’échantillonnage au terrain
La campagne sur le terrain (figure 7) a été réalisée le 21 Mai 2013, en condition climatique sèche (pas de pluie ce jour-là) et tempérée (environ 16°c).Des échantillons issus d’une précédente campagne de prélèvements réalisée par E.Tete le 28 mars 2013 ont également été utilisés pour la suite des analyses. Les échantillons de ces deux dates de prélèvement ont été
Figure 6 : Photo aérienne et emplacement des sites de prélèvement
9
poolés pour donner un échantillon global représentatif des différents sites.
Pour chaque point de prélèvement (haut, milieu et bas de pente) 3 répétitions ont été réalisées. Chaque répétition est le résultat de 5 carottages (composites) à la tarière (diamètre 5x20cm) (figure 8). Les échantillons composites ont été homogénéisés et quartés pour obtenir un échantillon (une répétition) représentatif de notre point de prélèvement.
Figure 8: Prélèvement à la tarière
Les points en bas et milieu de pente ont été réalisés aux localisations prévues, tandis que pour le point en haut de versant nos prélèvements ont été réalisés au sein de la parcelle de blé en se plaçant entre les rangées de blé pour minimiser l’effet du réseau racinaire sur nos échantillons (figure 9).
Figure 9 : Prélèvements à la tarière dans la parcelle de blé
Les échantillons ont été transportés en glacière (avec de la carboglace). De retour au laboratoire, ils ont été placés au congélateur à -40°C durant la nuit pour être conditionnés le lendemain pour la suite des analyses.
10
3. Analyses en laboratoire
a) Conditionnement des échantillons
Les échantillons ont été placés dans le lyophilisateur (figure 10) durant 30 heures pour obtenir des échantillons secs. La lyophilisation permet d’éliminer l’eau dans les sols par sublimation et donc dans les cellules pour une meilleure conservation du métagénome microbien. Les échantillons ont ensuite été tamisés à 4 mm (tamis à mailles carrées) (figure 11), le but étant d’extraire du sol les éléments grossiers comme les graviers mais aussi de « casser » les plus gros agrégats de sol.
Les échantillons ont ensuite été quartés (figure 12) et environ 50g de sol tamisé sont conservés dans des piluliers étiquetés et référencés au code propre à l’échantillon pour être stockés à long terme dans les congélateurs à -40°C du conservatoire (cryo-pédothèque GenoSol) (figure 13). La congélation rapide permet de stabiliser les matériaux biologiques (forme, structure et composition) et empêcher toute activité métabolique. Ces sols conservés pourront servir par la suite pour d’autres analyses de métagénome.
.
Figure 11 : Tamisage des échantillons Figure 10 : Lyophilisateur
Figure 12 : Congélateurs du conservatoire GenoSol Figure 13 : Quartage des échantillons
11
b) Quantification de l’ADN microbien
i. Extraction d’ADN à partir d’1 gramme de sol.
Cela a été réalisé par l’action simultanée d’une lyse mécanique (billes de différents diamètres) et d’une lyse chimique. La lyse mécanique a pour but de broyer les cellules des microorganismes, d’éclater les enveloppes et les tissus pour que la lyse chimique puisse libérer complètement l’ADN en solution.
1 gramme de sol sec lyophilisé est ajouté dans un tube contenant des billes de diamètres 4mm, 1.4mm et 0.1mm ainsi qu’une solution « tampon » à base de Tris-HCl, d’EDTA (Acide Ethylène Diamine Tétraacétique), de NaCl, de SDS (sodium dodecyl sulfate) et d’H2O
UP (eau ultra-purifiée).
La solution tampon a été préalablement filtrée à 0,2µm à l’aide d’une pompe à vide. Les tubes ont ensuite été vortexés puis placés dans un agitateur FastPrep (MPBio medicals) (figure 14) qui leur applique un mouvement alternatif à très haute vitesse. C’est au sein du FastPrep que s’effectue, grâce à l’agitation, la lyse mécanique.
A la sortie du FastPrep les tubes ont été vortexés une seconde fois puis placés dans un bain-marie à 70°C durant 30 min dans lequel s’effectue la lyse chimique. Enfin, les échantillons ont été centrifugés à 7000g pendant 5min pour obtenir la solution surnageante dans laquelle est concentré l’ADN de l’échantillon de sol : le lysat (figure 15).
Figure 15 : Lysats d'ADN "sale" obtenus
12
La totalité du lysat a été récupérée et stockée à -30°c dans le conservatoire GenoSol dans ce qui constitue la « lysa-thèque ». Le protocole d’extraction d’ADN se poursuit à partir d’1mL de ce lysat.
Etape de déprotéinisation, elle consiste à faire précipiter les protéines des lysats par l’ajout d’acétate de potassium dans une cuve de glace pour favoriser la formation de cristaux qui piègent les protéines qui ainsi précipitent. On obtient alors une solution surnageante claire qui contient l’ADN.
Etape de précipitation de l’ADN par l’ajout d’isopropanol à -20°c. Cela a pour effet « d’alourdir » l’ADN qui précipite au fond du tube. Les tubes sont ensuite centrifugés pour favoriser le dépôt d’ADN en « culot » et le surnageant ne contenant plus d’ADN est évacué.
Etape de lavage à l’éthanol du culot d’ADN qui permet de dissoudre les impuretés encore présentes dans le culot. Le surnageant est ensuite éliminé et les tubes contenant les culots d’ADN sont placés à l’étuve pendant 15 min (figure 16) pour éliminer par évaporation toute trace d’éthanol (qui est un inhibiteur d’analyses).
Etape de dissolution du culot d’ADN, elle a pour but de remettre l’ADN contenu dans le culot sec en solution par l’ajout d’H20 UP à 4°c
A la suite de ces étapes, une solution d’ADN dit « sale » (de nombreuses molécules n’ont pas été éliminées et sont mélangées à la solution d’ADN) a été obtenue. C’est à partir de cet ADN que l’on va pouvoir mesurer la biomasse moléculaire microbienne.
ii. Estimation de la quantité d’ADN par électrophorèse sur gel d’agarose 1%.
L’électrophorèse est une méthode de séparation des acides nucléiques selon leur poids moléculaires. Les ADN sont chargés négativement et la technique utilise cette propriété pour les séparer sous l’action d’un courant électrique.
Une électrophorèse sur gel d’agarose à 1% des solutions d’ADN « sales » a été effectuée en comparaison avec une gamme d’ADN de thymus de veau pour estimer leur quantité. Le courant électrique circulant dans la cuve remplie de tampon TBE 1X de l’anode à la cathode fait migrer les échantillons préalablement mélangés à un bleu de dépôt qui permet la visualisation du front de migration et d’alourdir les échantillons lors du dépôt (figure 17).
Figure 16 : Etuve Figure 16 : Etuve
13
Après un temps de migration compris entre 1 et 2 heures, le gel a été plongé durant 30 min dans un bain de BET (Bromure d’éthidium), qui est un intercalant de l’ADN fluorescent sous l’action d’UV, puis rincé à l’eau durant 20 min (figure 18). Le gel a ensuite été visualisé sur une table UV associée à une caméra haute précision (figure 19). L’image fluorescente du gel est obtenue grâce au logiciel BioCapt.
Puits ou sont déposés les échantillons +
bleu de dépôt Cuve + solution
tampon de TBE 1X Gel d’agarose à 1%
Cathode
Anode
Sens de migration des échantillons
Figure 17 : Electrophorèse sur gel d'agarose Figure 17 : Electrophorèse sur gel d'agarose
14
iii) Quantification d’ADN sur gel par Image Quant TL
Pour déterminer la concentration en ADN dans chaque échantillon, l’image du gel obtenue a été analysée par le logiciel ImageQuant (figure 20). Celui-ci mesure l’intensité de la fluorescence sous rayons UV émise par le BET fixé sur l’ADN des échantillons.
La mesure d’intensité de fluorescence permet de calculer la quantité d’ADN extraite grâce aux quantités connues de la gamme d’étalonnage. En prenant en compte toutes les étapes d’extraction d’ADN, la quantité d’ADN extraite est exprimée en ng d’ADN/g de sol.
c) Caractérisation de la structure génétique des communautés microbiennes
La technique ARISA (Automated Ribosomal Intergenic Spacer Analysis) permet d’obtenir une empreinte moléculaire des communautés microbiennes de chaque échantillon de sol. Cette technique complexe s’est déroulée en plusieurs étapes distinctes :Figure 20 : Image de gel après migration Ladder 1kb
Ladder 1kb Gamme d’ADN de
thymus de veau
Echantillons d’ADN « sale » Puits de dépôt
15
Pré-purification des ADN « sales » sur colonne de PVPP
Cette étape consiste en un tamis moléculaire réalisé sur colonne Microbiospin (Biorad) de PVPP. Le PVPP (PolyVinyl PolyPirrolidone) est un polymère de poids moléculaire très élevé qui se complexe avec les composés phénoliques et les alcaloïdes. Cette étape a permis d’obtenir une solution d’ADN débarrassée de ces composés inhibiteurs d’analyses moléculaires qui restent fixés sur la colonne de PVPP (figure 21).
Purification par Kit GeneClean sur colonne de silice
C’est une méthode de lavage d’ADN extrait grâce à une colonne de silice utilisant le principe d’affinité/exclusion. Lors de cette étape l’ADN se lie à la membrane de silice (Geneclean) en présence de fortes concentrations en sels puis est élué en faibles concentrations en sels après avoir été lavés avec une solution à base d’éthanol (figure 22).
Figure 21 : Pré-purification par PVPP (T.Regnier, 2010)
16
Ces deux étapes ont été complémentaires pour permettre d’éliminer d’éventuels éléments tels que les protéines, sucres, macromolécules phénoliques, acides humiques.
L’ADN en solution aqueuse obtenu est appelé ADN « propre », il est conservé à -20°C au sein du conservatoire GenoSol (DNAthèque).
PCR-ARISA
La technique ARISA (figure 23) permet l’étude de la structure des communautés microbiennes (bactéries/champignons) d’un sol. Cette méthode exploite le polymorphisme de taille de l’espace intergénique (IGS) entre les gènes codant pour l’ARNr 16S et l’ARN 23S des bactéries et les gènes codant pour l’ARNr 18S et l’ARNr 25-28S des champignons. Ce polymorphisme varie pour la grande majorité des espèces procaryotes entre 50 et 1500 paires de bases.
17
La base de la technique ARISA repose sur la PCR (Polymerase Chain Reaction) qui est une technique permettant d’amplifier in vitro des séquences d’ADN cibles par répétition de réactions d’élongation en présence d’amorces nucléotidiques spécifiques et d’une ADN polymérase (figure 24).
Le principe de la PCR repose sur la répétition de 3 processus :
La dénaturation des deux brins d’ADN à température élevée (environ 95°C) afin d’obtenir des molécules d’ADN monobrin.
L’hybridation d’amorces oligonucléotidiques (ou « primers ») complémentaires d’une séquence d’ADN monobrin cible à température permettant la réaction (environ 50°C).
La réaction d’élongation par une ADN polymérase thermostable (« Taq polymérase ») à partir des amorces à température optimale (72°C).
Les produits de premier cycle de PCR sont dénaturés par la chaleur et le cycle recommence n fois. A chaque cycle, le nombre de copies du fragment d’ADN est doublé. La PCR suit donc la formule mathématique : 2n molécules obtenues après n cycles. Deux types de PCR, PCR-B ARISA pour l’analyse des communautés bactériennes et une PCR-F ARISA pour l’analyse des communautés fongiques de nos échantillons, ont été réalisées.
18
Purification des produits PCR par Kit Qiagen
Cette purification permet l’élimination des nucléotides, primers, polymérases et sels en excès sur colonne d’affinité (MinElute Spin Columns). Cette purification est nécessaire pour l’étape suivante : le séquençage LiCor.
La purification par le kit Qiagen MinElute repose sur le principe d’exclusion-affinité. La première étape consiste à fixer l’ADN sur une membrane de silice grâce à une forte concentration en sels, puis l’ADN est lavé à l’aide d’une solution à base d’alcool avant d’être élué avec de l’eau UP.
Quantification des PCR purifiées
Les PCR purifiées ont ensuite été quantifiées sur gel agarose 2% pour déterminer la dilution nécessaire des échantillons pour la suite des analyses.
Migration électrophorétique sur gel de polyacrylamide : LiCor
L’électrophorèse des produits purifiés des deux PCR a été réalisée sur séquenceur LiCor (figure 26) dans le but d’obtenir les profils ARISA ou « fingerprints » des communautés bactérienne et fongique des échantillons.
La migration se fait sur gel de polyacrylamide. Le principe est le même que l’électrophorèse sur gel d’agarose mais la distance de migration est bien plus grande (environ 60 cm) (figure 25) et la maille du gel de polyacrylamide est bien plus fine, ce qui produit une migration de meilleur qualité car la séparation des molécules d’ADN selon leur taille est plus efficace.
Figure 26 : Séquenceur LiCor Figure 25 : Système d'électrophorèse
19
Les échantillons de PCR purifiée et dilués ont été associés à un rose de dépôt (stop IR²) et déposés dans les puits du gel de polyacrylamide. La migration a été effectuée durant 14h.
Les profils ARISA des communautés bactériennes et fongique des échantillons sont analysés par le logiciel One-Dscan qui permet la conversion des profils ARISA en matrices de données intégrant la taille et l’intensité des 100 bandes majoritaires pour chaque échantillon. Ces matrices sont analysées sous R en composantes principale (ACP).
III. Résultats et discussion
1.
Caractérisation du régime hydrique des sols.
Un suivi de l’humidité volumétrique des sols par sondes TDR a été réalisé sur la période du 21/02/2013 au 18/04/2013 par E. Tete dans le cadre de ses travaux de thèse (figure 27).
La figure montre que l’humidité volumétrique du sol est, sur l’ensemble de la période de suivi, plus élevée en bas de versant qu’en haut de versant.
20
Une mesure de l’humidité pondérale (figure 28) a été réalisée sur les échantillons de sol de la campagne d’échantillonnage du 21 Mai.
La figure 28 montre que l’humidité pondérale des sols est plus élevée en bas et milieu de versant qu’en haut de versant. Il y a un gradient d’humidité des sols croissant du haut de versant vers le bas de versant.
Ces données d’humidité associées aux données pédologiques confirment un des postulats de départ, à savoir le fait que les sols présentent un gradient d’hydromorphie croissant du haut vers le bas de versant. Ce profil de régime hydrique est en adéquation avec l’influence attendue de la nappe d’eau sur le profil de sol en fonction de la topographie (versant agricole).
2.
Profil de flux de respiration des sols.
Un suivi in situ de la respiration hétérotrophe des sols a été réalisé à différentes dates par E. Tete dans le cadre de ses travaux de thèse (figure 29).
.
Figure 28 : Humidité pondérale des sols lors de la campagne de prélèvement
21
La figure 29 montre que la respiration hétérotrophe (flux de CO2) est plus élevée en haut qu’en bas
de versant.
Le phénomène de respiration hétérotrophe est lié à l’activité de décomposition de la MOS par les microorganismes (processus de minéralisation et humification). La décomposition de la MOS est notamment liée à la quantité, à l’activité et à l’efficacité des microorganismes (Liu et al., 2010).
Il s’agit donc maintenant de déterminer si ces observations peuvent être associées à des différences de communautés microbiennes entre les 3 sites.
3.
Quantification de l’ADN microbien
La figure 30 présente les résultats de quantification d’ADN microbien, soit un équivalent de la biomasse microbienne :
La figure montre une différenciation des sites : le sol de haut de versant présente la plus faible quantité d’ADN donc une plus faible quantité de microorganismes, le sol de milieu de versant présente la plus forte quantité d’ADN donc la plus forte quantité de microorganismes, le sol de bas de versant présente quant à lui des quantités intermédiaires. Le sol de bas de versant ayant une teneur en carbone plus élevée, il est cohérent que la biomasse microbienne de ce site soit plus élevée.
Il apparaît que le sol de bas de versant présentant une humidité et une teneur en carbone plus élevées est également celui qui présente une biomasse microbienne plus élevée. Les populations
22
microbiennes sont donc plus importantes quantitativement en milieu humide et avec une teneur en carbone élevée.
Cependant, ce même sol présente des valeurs de respiration hétérotrophe inférieures à celui de haut de versant. Cela montre que le paramètre quantitatif (biomasse microbienne) n’est pas déterminant dans le processus de respiration hétérotrophe. La respiration n’est pas simplement reliée de manière linéaire à la biomasse microbienne.Cela peut s’expliquer par le fait que les microorganismes ont besoin d’une certaine disponibilité en oxygène pour respirer. Si la porosité est occupée par l’eau, le sol entre en conditions anaérobiques ce qui a pour conséquence une inhibition du processus de respiration aérobie. C’est probablement le cas du sol de bas de versant où la granulométrie doit être plus fine (microagrégats) et la porosité moins élevée et donc occupée par l’eau (espace poral saturé), contrairement au cas du sol de haut de versant où les conditions de régime de sol bien drainé seraient plus favorables à la respiration aérobie. En zone de bas de versant où le sol est saturé en eau la majeure partie de l’année les conditions rédox sont telles que le processus de dénitrification doit être plus important.
Il s’agit maintenant de déterminer si les communautés microbiennes des sols présentent des différences dans leur structure génétique qui pourraient être reliées à nos observations.
4.
Caractérisation de la structure génétique des communautés
microbiennes
Les profils ARISA bactériens et fongiques des échantillons ont été analysés en composantes principales (figure 31 et 32).
23
La figure 31 montre, suivant l’axe 2, une différenciation du sol de bas de versant par rapport aux sols de milieu et haut de versant.
Figure 31 : ACP des profils ARISA des bactéries
24
On constate également sur la figure 32 une différenciation de la structure génétique de la communauté fongique du sol de bas de versant suivant les axes 1 et 2.
Une différenciation des communautés à la fois bactériennes et fongiques est observée en zone de bas de versant. Cela est sans doute associé aux conditions particulières de ce milieu (teneur en C, et engorgement en eau) qui ont orienté les communautés vers des populations spécifiques adaptées aux conditions. Cette zone de bas de versant constitue un habitat écologique spécifique. La respiration en haut de versant, supérieur aux autres sites et malgré une biomasse plus faible, peut être associée à des populations spécifiques plus efficaces dans le processus de respiration.
25
IV. Conclusion
L’influence du gradient d’hydromorphie sur les communautés microbiennes du sol est un domaine de recherche relativement nouveau qui s’inscrit parmi les actuels projets phares en agronomie : l’étude des dynamiques des MOS à l’échelle des paysages agricoles intensifs. L’étude menée a cherché à vérifier s’il existe des différences entre les communautés microbiennes d’un sol de bas de versant agricole présentant un régime hydrique de sol saturé et celles d’un sol de haut de versant présentant un régime hydrique de sol bien drainé.
L’étude a d’abord démontré une différence d’humidité entre ces deux sols, corroborant la présence d’un gradient d’hydromorphie croissant du haut vers le bas de versant. Elle a également démontré une distinction de ces sols en matière de respiration hétérotrophe ce qui traduit une différence d’activité de décomposition de la MOS par les microorganismes.
L’analyse de la biomasse et de la structure génétique des communautés a montré une différenciation des communautés bactériennes et fongiques entre le sol de bas de versant et celui de haut de versant. Cela a confirmé l’hypothèse de départ à savoir une différence des communautés microbiennes des sols selon le gradient d’hydromorphie.
Cependant il serait judicieux de poursuivre cette étude par des expériences au laboratoire en conditions contrôlées. En appliquant aux sols de l’étude les mêmes conditions d’humidité et de structure du sol (taille des agrégats), il serait alors possible de confirmer ou d’infirmer le lien entre la composition des communautés microbiennes et des fonctions du sol. Lors de la suite du stage pendant le mois de juin, nous allons poursuivre l’étude en se basant sur la technique de PCR quantitative (qPCR) qui fournira une nouvelle information à savoir le ratio bactéries/champignons pour chacun des sols et ainsi voir si le gradient d’hydromorphie influence le développement d’une de ces deux communautés en particulier.
D’un point de vue personnel ce stage au sein de l’INRA de Dijon m’a permis de comprendre le fonctionnement de la recherche scientifique dans le domaine de l’agroécologie. Il m’a permis d’appréhender des techniques novatrices en matière d’extraction et d’analyses de l’ADN microbien d’un sol. Ce stage m’aura également permis de me perfectionner dans la gestion et l’interprétation de données ainsi que dans la rédaction de rapport scientifique.
26
Références bibliographiques
.
Sites internet :
http://www2.dijon.inra.fr/plateforme_genosol/presentation.php consulté le 30/04/13 et 02/05/13 http ://www.rennes.inra.fr/Toutes-les-actualites/MOSAIC-gerer-la-matiere-organique-des-sols-en-contexte-de-changements-globaux consulté le 02/05/13 http://www.sesep.uvsq.fr/formation/methodes.html consulté le 05/06/13Publications scientifiques :
Bressan, M., Mougel, C., Dequiedt, S., Maron, P.-A., Lemanceau, P., Ranjard, L., 2008. Response of soil bacterial community structure to successive perturbations of different types and intensities. Environmental Microbiology 10, 2184-2187.
Dequiedt, S., Saby, Lelievre, M., Jolivet, C., Thioulouse, J., Toutain, B., Arrouays, D., Bispo, A., Lemanceau, P., Ranjard, L., 2010. Biogeographical patterns of soil molecular microbial biomass as influenced by soil characteristics and management, global ecology and biogeography, (global ecol.biogeogr.).
Dumont, M.G., Murrell, J.C., 2005. Stable isotope probing - linking microbial identity to function. Nature Reviews Microbiology 3, 499-504.
FAO/ISRIC/ISSS, 1998. World Reference Base for Soil Resources. Food and Agriculture Organization of the United Nations, Rome, Italy
FAO, 2009. The state of food insecurity in the world 2009. Economic crisis-impacts andlessons learned (FAO, Rome, 2009).
Hättenschwiler, S., Tiunov, A., Scheu, S., 2005. Biodiversity and litter decomposition in terrestrial ecosystems. Annu.Rev.Ecol.Syst.2005 36:191-218
Lal, R., 2009. Soil quality impacts of residue removal for bioethanol production. Soil and Tillage Research 102, 233-241.
27
Maron, P.A., Ranjard, L., Mougel, C., Lemanceau, P., 2007. Metaproteomics: A new approach for studying functional microbial ecology. Microbial Ecology 53, 486-493
Martiny. J, B. Bohannan, J.Brown, R.Colwell, J Fuhrman, J.Green, M.Horner-Devine, M Kane, J. Krumins, C.Kuske, P.Morin, S. Naeem, L. Øvreås, A. Reysenbach, V.Smith & J. Staley, 2006. Microbial biogeography: putting microorganisms on the map.Nature Reviews Microbiology 4, 102-112
Morales, S., Holben, W., 2010. Linking bacterial identities and ecosystem processes: can ‘omic’ analyses be more than the sum of their parts? FEMS Microbiology Ecology Volume 75, Issue 1, pages 2–16
Ranjard, L., Poly, F., Combrisson, J., Richaume, A., Gourbière, F., Thioulouse, J., Nazaret, S., 2000. Heterogeneous Cell Density and Genetic Structure of Bacterial Pools Associated with Various Soil Microenvironments as Determined by Enumeration and DNA Fingerprinting Approach (RISA). Microbial Ecology (2000) 39:263-272
Rees, R.M., Bingham, I.J., Baddeley, J.A., Watson, C.A., 2005. The role of plants and land management in sequestering soil carbon in temperate arable and grassland ecosystems. Geoderma 128: 130-154.
Swift, M.J., Heal, O.W., Anderson, J.M., Greig, S.D.J., Pitelka, P., Frank, A., 1979.Decomposition in terrestrial ecosystems. Blackwell, Oxford.
Tiessen, H., Cuevas, E., Chacon, P., 2000. The role of organic matter in sustaining soil fertility. Nature 371, 783 – 785
Weixing Liu, Wenhua Xu, Jianping Hong, Shiqiang Wan, 2010. Interannual variability of soil microbial biomass and respiration in responses totopography, annual burning and N addition in a semiarid temperate steppe. Geoderma 158: 259–267
Ouvrages:
D. Targu, C.Moussard, éds. Principes et techniques de biologie moléculaire, 2ème édition revue et augmentée. INRA éditions.
28
Quentin AUDOUIT M1 SE 2012/2013
Etude de l'influence de l'hydromorphie des sols sur les
communautés microbiennes à l'échelle d'un versant agricole
RESUME
Le projet MOSAIC vise à étudier les facteurs contrôlant la dynamique des MOS à l’échelle d’un paysage agricole dans un contexte de durabilité des systèmes culturaux.
Le but du stage était d’étudier l’impact du gradient d’hydromorphie sur les communautés microbiennes des sols à l’échelle d’un versant agricole du site d’étude de MOSAIC. Les échantillons de sol étudiés sont issus de campagnes de prélèvement datant du 28 mars et du 21 mai 2013.
Les analyses en laboratoire ont démontré la présence effective d’un gradient d’humidité croissant du haut vers le bas de versant ainsi que des différences dans les valeurs de respiration hétérotrophe de ces sols. Par la suite, l’étude a mis en évidence des différences de quantité d’ADN microbien des sols donc de taille des populations microbiennes des sols ainsi que des différences dans la structure génétique des communautés bactériennes et fongiques via la technique de PCR-ARISA.
La zone de bas de versant, avec ses caractéristiques de sol (humidité, teneur en carbone, structure) constitue un habitat écologique spécifique.
Mots-clés : sol, versant agricole, gradient d’hydromorphie, communautés microbiennes, PCR-ARISA, habitat écologique.