Caractérisation de la protéine Cérès :
À la frontière entre polarité épithéliale et
contrôle énergétique
Mémoire
Émilie Hardy
Maîtrise en biologie cellulaire et moléculaire
Maître ès sciences (M.Sc.)
Québec, Canada
© Émilie Hardy, 2015
RÉSUMÉ
La polarité apico-‐basale des cellules épithéliales est essentielle aux fonctions des tissus épithéliaux. La protéine Yurt promeut l’identité de la membrane basolatérale et régule négativement l’activité de Crumbs par des mécanismes encore inconnus. Crumbs est un déterminant apical qui réprime la croissance tumorale et prévient la transition épithélio-‐mésenchymateuse (EMT).
Nous avons identifié la protéine CG5964 (ALBATROSS chez l’humain) comme un nouvel interacteur physique de Yurt. L’objectif de ma maîtrise est de réaliser l’analyse fonctionnelle de cette protéine (renommée Cérès chez la drosophile), par des méthodes biochimiques et génétiques (génération d’un allèle mutant et d’une lignée de surexpression).
Nos résultats suggèrent que la protéine Cérès n’est pas impliquée dans la polarité épithéliale mais plutôt dans le maintien de l’équilibre énergétique. Mon projet apportera les données de base sur la protéine de drosophile Cérès qui n’avait jamais été caractérisée. La compréhension du rôle fonctionnel de l’interaction entre Cérès et Yurt pourrait révéler de nouvelles fonctions de Yurt. Dans l’optique d’utiliser Yurt comme cible thérapeutique pour lutter contre le cancer, il est important de connaître les différents processus biologiques dans lesquels Yurt est impliquée.
Table des matières
Résumé …... iii
Table des matières ... v
Liste des tableaux ... ix
Liste des figures ... xi
Listes des abréviations ... xiii
Épigraphe ... xvii
Remerciements ... xix
1. Introduction ... 1
1.1 Les tissus épithéliaux ... 1
1.2 Les jonctions cellules-‐cellules ... 2
1.3 La polarité apico-‐basale ... 5
1.4 Les modules de régulation de la polarité apico-‐basale ... 6
1.5 La protéine Yrt ... 9
1.5.1 Structure de la protéine Yurt ... 9
1.5.2 Fonctions de Yrt dans la polarité apico-‐basale ... 10
1.5.3 Fonctions d’endocytose de Yrt ... 11
1.5.4 Implication de Yrt dans le cancer ... 12
1.6 La protéines Cérès ... 12
1.7 Présentation du modèle d’étude : Drosophila melanogaster ... 14
1.7.1 Cycle de vie ... 15
1.7.2 L’instestin ... 17
1.7.3 Le corps gras ... 21
1.7.4 Les ovaires ... 22
1.7.4.1 Ovogenèse ... 22
1.7.4.2 Point de contrôle de dégénérescence ... 24
1.8 Hypothèse et objectifs ... 25
2. Matériel et méthodes ... 27
2.1 Biologie moléculaire et lignées de drosophiles ... 27
2.1.1 Lignée transgénique de surexpression Cérès ... 27
2.1.2 Allèle mutant ceres8 et ceres3 ... 28
2.1.3 Autres lignées ... 29
2.1.4 Migration sur gel d’acrylamide ... 29
2.2 SDS PAGE et Western blot ... 30
2.3 Fixation d’embryons et immunofluorescence ... 31
2.4 Immunoprécipitation ... 33
2.5 Observation des lipides ... 33
2.6 Techniques de drosophiles ... 34
2.6.2 Test de viabilité embryonnaire ... 34
2.6.3 Mesure de la ponte d’embryons ... 35
2.6.4 Mesure de la prise alimentaire des adultes ... 35
2.6.5 Test de survie des adultes ... 35
2.6.6 Mesure de la taille des adultes ... 36
2.6.7 Préparation de cuticule d’embryons ... 36
2.6.8 Alimentation ... 36
2.7 Imagerie ... 37
2.8 Analyse statistique ... 37
2.9 Alignement de séquençage ... 37
3. Résultats ... 39
3.1 Analyse spatio-‐temporelle de l’expression de la protéine Cérès dans l’embryon de drosophile ... 39
3.1.1 Niveau d’expression protéique ... 39
3.1.2 Localisation protéique ... 40
3.2 Génération des outils moléculaires indispensables à l’étude fonctionnelle de Cérès ... 42
3.3 La protéine Cérès n’exerce pas de fonctions majeures dans l’établissement et le maintien de la polarité apico-‐basale ... 45
3.3.1 Développement des mutants ceres ... 46
3.3.2 Distribution des acteurs de le polarité apico-‐basale ... 48
3.3.3 Recherche d’interactions fonctionelles avec différents acteurs de la polarité apico-‐basale ... 49
3.4 Mise en évidence de nouvelles fonctions de Cérès dans le contrôle de l’équilibre énergétique ... 51
3.4.1 Perturbation progressive de la reproduction des mutants ceres ... 51
3.4.1.1 Défaut de la ponte et de la fécondation des œufs ceres ... 51
3.4.1.2 Altération de l’intégrité des ovaires ... 53
3.4.2 Les mutants ceres présentent des signes de stress métabolique ... 55
3.4.2.1 Activation des voies signalétiques de stress ... 55
3.4.2.2 Épuisement des réserves énergétiques ... 56
3.4.2.3 Retard de développement ... 57
3.4.3 Recherche des facteurs provoquant la carence nutritive des mutants ceres ... 59
3.4.3.1 Comportement alimentaire ... 59
3.4.3.2 Signalisation de la voie à l’insuline ... 60
3.4.3.3 Physiologie de l’intestin ... 62
4. Discussion et conclusion ... 65
4.1 Analyse spatio-‐temporelle de l’expression de la protéine Cérès ... 65
4.2 Les mutants ceres8 sont en carence nutritive ... 68
4.2.1 Arrêt précoce de la ponte ... 68
4.2.3 Sur-‐activation de l’AMPK ... 71
4.2.4 Diminution des réserves énergétiques ... 72
4.3 Fonctions de la protéine Cérès chez la drosophile ... 73
4.3.1 Polarité ... 73
4.3.2 Comportement alimentaire et voie à l’insuline ... 75
4.3.3 Maintien de la physiologie de l’épithélium intestinal ... 76
4.4 À la frontière entre polarité et métabolisme ... 78
4.5 Conclusion ... 80 5. Annexe ... 81 6. Bibliographie ... 83
Liste des tableaux
Tableau 1 : Conservation des protéines de polarité de l’humain à la drosophile ... 8 Tableau 2 : Anticorps et dilutions utilisés en Western blot et
immunofluorescence ... 32
Liste des figures
Figure 1 : Différents types d’épithéliums tapissent les cavités des organes
internes ... 1
Figure 2 : Schéma des structures jonctionnelles chez les Vertébrés et Drosophila ... 3
Figure 3 : Composition des jonctions adhérentes et septées chez la Drosophile ... 4
Figure 4 : Schéma d’un épithélium simple polarisé ... 5
Figure 5 : Schéma des modules de polarité présents dans les cellules épithéliales de drosophile ... 7
Figure 6 : Schéma représentant les domaines protéiques de Yrt ... 10
Figure 7 : Schéma représentant les fonctions de Cérès dans les cellules cancéreuses humaines ... 13
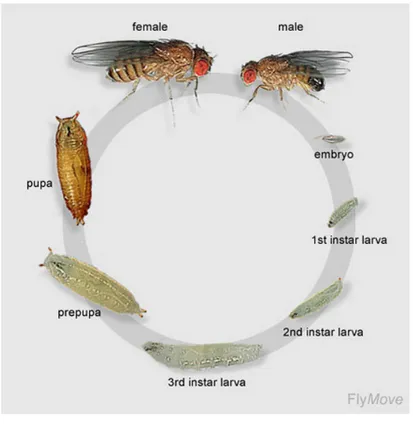
Figure 8 : Cycle de vie de Drosophila melanogaster ... 15
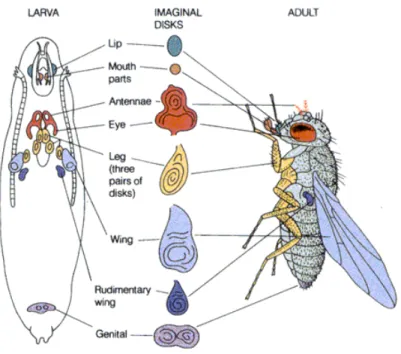
Figure 9 : Les disques imaginaux chez Drosophila melanogaster ... 17
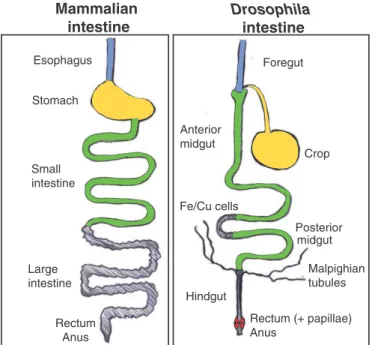
Figure 10 : Le système digestif de drosophile ... 18
Figure 11 : Formation du système digestif de drosophile ... 19
Figure 12 : Les types de cellules de l’intestin moyen chez l’adulte ... 20
Figure 13 : Stockage de gouttelettes lipidiques dans le corps gras ... 22
Figure 14 : Schéma représentant les structures ovariennes de drosophile ... 23
Figure 15 : Carte du plasmide pUASTattB ... 28
Figure 16 : Niveau d’expression de la protéine Cérès endogène au cours du développement de drosophile ... 39
Figure 17 : Localisation de la protéine Cérès endogène dans l’embryon de drosophile ... 41
Figure 18 : Caractérisation de la lignée transgénique de surexpression et des allèles mutants ceres dans l’embryon de drosophile ... 44
Figure 19 : Cérès interagit physiquement avec Yrt ... 45
Figure 20 : Les mutants ceres8 sont viables ... 47
Figure 21 : La modulation de l’expression de Cérès n’affecte pas l’architecture polarisée des tissus épithéliaux ... 49
Figure 22 : Absence d’interaction fonctionnelle entre Cérès et les protéines de polarité apico-‐basale Crb et Yrt ... 50
Figure 23 : Arrêt précoce de la ponte d’embryons des mutants ceres8 ... 52
Figure 24 : Dégénérescence des follicules ovariens matures des mutants ceres8 ... 54
Figure 25 : La phosphorylation de l’AMPK augmente dans les mutants ceres8 en réponse à une privation de nourriture ... 56
Figure 26 : Les réserves énergétiques des mutants ceres8 disparaissent au cours du temps ... 57
Figure 27 : Les mutants ceres8 présentent un retard de développement ... 58
Figure 28 : La prise alimentaire des mutants ceres8 est normale ... 60
Figure 29 : La voie à l’insuline est fonctionnelle dans les mutants ceres8 ... 61
Figure 30 : Perte de l’intégrité de l’épithélium intestinal des mutant ceres8 ... 62
Liste des abréviations et acronymes
α-‐Cat : Alpha catenin
AMG : Précurseurs de l’intestin moyen antérieur
AMPK : Adenosine monophosphate-‐activated protein kinase AP : Protéine adaptatrice
aPKC : Atypical protein kinase C Arm : Armadillo
Baz : Bazooka
BCA : Acide bicinchoninique CH4O : Méthanol
C2H3Cl3O2 : 2,2,2-‐trichloroéthane-‐1,1-‐diol (Hydrate de chloral)
C2H4O2 : Acide acétique
C2H5NO2 : Acide aminoacétique (glycine)
C2H6OS : 2-‐Hydroxy-‐1-‐éthanethiol (β-‐Mercaptoéthanol)
C3H5NO : 2-‐propénamide (acrylamide)
C3H6O2 : Acide propanoïque (Acide propionique)
C3H6O3 : Acide 2-‐hydroxypropanoïque (Acide lactique)
C3H8O3 : Propan-‐1,2,3-‐triol (glycérol)
C3H7O6PNa2.5H2O : Sodium beta-‐glycérophosphate pentahydraté
C4H11NO3 : 2-‐amino-‐2-‐hydroxyméthyl-‐1,3-‐propanediol (Tris)
C6H16N2 : Tétraméthyléthylénédiamine (TEMED)
C7H7FO2S : Fluorure de phénylméthylsulfonyle (PMSF)
C7H16 : Heptane
C8H7N3O2 : 5-‐amino-‐2,3-‐dihydro-‐1,4-‐phthalazinedione (luminol)
C8H8O3 : 4-‐hydroxybenzoate de méthyle (méthylparabène)
C9H8O3 : Acide coumarique
C10H16N2O8 : Acide éthylène diamine tétraacétique (EDTA)
C12H25NaO4S : Dodécylsulfate de sodium (SDS)
C18H14N2Na2O8S2 : 6-‐hydroxy-‐5-‐[(2-‐méthoxy-‐5-‐méthyl-‐4-‐sulfophényl)azo]-‐2-‐
naphtalénesulfonate de disodium (Rouge Allura)
C19H10Br4O5S : 3,3',5,5'-‐tétrabromophénol-‐sulfonephthaléine (Bleu de
bromophénol)
C20H12N2Na2O7S2 : Rouge ponceau
C20H12N2O4 : Acide 2,2’-‐biquinoline-‐4,4'-‐dicarboxylique (acide bicinchoninique,
BCA)
C20H38N6O4. 0.5 H2SO4 : N-‐Acetyl-‐L-‐leucyl-‐L-‐leucyl-‐L-‐argininal hemisulfate salt
(Leupeptine)
C21H20BrN3 : Bromure de 3,8-‐diamino-‐1-‐étyhl-‐6-‐phénylphénanthridinium (BET,
EtBr)
C33H60O10.5 : Triton X-‐100
C34H63N5O9 : I-‐Valeryl-‐L-‐Val-‐L-‐Val-‐AHMHA-‐L-‐Ala-‐AHMHA (Pepsatin A)
C58H114O26 : Polyoxyethylene (20) sorbitan monolaurate (Tween 20)
Cdc42 : Cell division cycle protein 42 Crb : Crumbs
Cora : Coracle
CuSO4,5H2O : Sulfate de cuivre pentahydraté
DAPI : 4',6'-‐diamidino-‐2-‐phénylindole DE-‐Cad : DE-‐Cadherin
Dlg : Discs large ECs : Enterocytes
EEs : Cellules enteroendocrines Eh : Eclosion homone
ETH : Ecdysis-‐triggering hormone
EMT : Transition épithélio-‐mésenchymateuse FA : FERM adjacent
FBM : Ferm binding motif
FERM : 4.1, Ezrin, Radixin and Moesin H2O2 : Peroxyde d’hydrogène
H3BO3 : Acide borique
Hh : Hedgehog Hpo : Hippo
ISCs : Cellules souches intestinales JA : Jonctions adhérentes
JS : Jonctions septées
K2HPO4 : Phosphate dipotassique
KCl : Chlorure de potassium Lgl : Lethal giant larvae
Na2HPO4 : Phosphate disodique
NaCl : Chlorure de sodium
NaClO : Hypochlorite de sodium (Eau de Javel) NaF : Fluorure de sodium
Na,K-‐ATPase : Sous unité alpha de la pompe sodium potassium Na3VO4 : Orthovanadate de sodium
(NH4)2S2O8 : Persulfate d’ammonium (APS)
Nrg : Neuroglian Nrx-‐IV : Neurexin IV
O/N : Une nuit de 16h (Overnight) Par-‐1 : Protease activated receptor 1 Par-‐3 : Partitioning defective 3 Par-‐6 : Partitioning defective 6 PBS : Tampon phosphate salin PBT : PBS Triton 100X
PDB : PDZ Binding Site
PDZ : Post synaptic density protein (PSD95), Drosophila Discs large tumor
suppressor (Dlg), and Zonula occludens-‐1 protein (Zo-‐1)
PMG : Précurseurs de l’intestin moyen postérieur Sav : Salvador
Std : Stardust
TAE : Tris, acétate, EDTA TBE : Tris, borate, EDTA
THPD : Domaine homologue à la trychohyalin et à la plectin Wg : Wingless WT : Wild type Wts : Warts Yki : Yorkie
« Everybody is a genius. But if you judge a fish by its ability to climb a tree,
it will live its whole life believing that it is stupid » -‐ Albert Einstein
Remerciements
Je remercie tout d’abord mon directeur de recherche, Dr Patrick Laprise, de m’avoir acceptée dans son laboratoire et soutenue dans la réalisation de cette maîtrise. Merci de m’avoir accordé ta confiance et d’avoir fait naître en moi la même passion que tu as pour la recherche. Merci pour ton enseignement et ton
encadrement de qualité, qui m'ont permis d'être la scientifique que je suis
aujourd’hui. Pour finir, merci de m’avoir appris à toujours donner le meilleur de soi et à viser l’excellence « If you work hard and it’s doesn’t work… Work harder ».
Merci aux membres du jury, Dr Martin Simard et Dr Mathieu Laplante, pour l’évaluation de mon mémoire et à la directrice du programme de Biologie Cellulaire et Moléculaire, Dr Josée Lavoie, pour ses recommandations et ses précieux conseils.
Je voudrais également remercier tous les membres de mon laboratoire, sans qui la vie de tous les jours n’aurait pas été la même.... Merci d’avoir assuré mes
arrières et d’avoir toujours été présent quand j’avais besoin de vous. Merci d’avoir eu la patience de répondre à mes questions, de m’aider dans mes manips et de partager des discussions scientifiques ou loufoques en salle à mouches. Je voudrais remercier particulièrement Helori et Elise pour leur amitié qui a largement dépassé le centre de recherche. Merci à Kevin d'avoir partagé avec moi sa vision du monde, et d'avoir passé des heures à débattre et à imaginer toutes sortes de théories. Merci à François de m'avoir tant appris lors de mes premiers pas au laboratoire, et d'avoir toujours continué à s’intéresser à mes projets, même après son départ du laboratoire. Merci à Lucile, Claire, Oussama, et Karine pour leur amitié et tous les bons moments passés ensemble. Merci à Rémi d'avoir été une source d’inspiration, un modèle à suivre et de m’avoir donné la chance de marcher dans la cour des grands…
Enfin, je souhaiterais remercier ma famille car sans elle je n'en serais pas là aujourd’hui. Merci à mes parents et à ma sœur de ne jamais avoir douté de mes capacités et d'être fiers de moi quoi qu’il en soit. Merci de m’avoir toujours soutenue dans la réalisation de mes projets, même quand ils m’ont menée à l’autre bout du monde… Merci à Julie pour son soutien inconditionnel et sa patience face à ce rythme de vie difficile. C’est avec toi que j’ai partagé mes réussites et que je me suis relevée de mes échecs. Merci de faire partie de ma vie et d’être quoi qu’il arrive à mes côtés.
1. Introduction
1.1 Les tissus épithéliaux
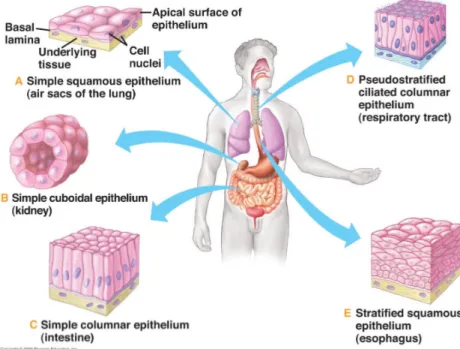
Les tissus épithéliaux sont l’un des quatre types de tissus retrouvés chez l’humain avec les tissus conjonctifs, musculaires et nerveux. Ils recouvrent la surface du corps et les cavités internes des organes, assurant ainsi une délimitation entre le milieu intérieur et extérieur. Ils sont caractérisés par la forme des cellules qui les composent soit pavimenteuses, cubiques ou cylindriques, et la capacité de ces cellules à se superposer formant ainsi des épithéliums simples, pseudo-‐stratifiés ou stratifiés (Fig 1). Les tissus épithéliaux sont également définis par la structure qu’ils adoptent telle que le feuillet, le tubule ou la glande.
Figure 1 : Différents types d’épithéliums tapissent les cavités des organes internes
Les cellules des épithéliums présentent une morphologie pavimenteuse (A, E), cubique (B) ou cylindrique (C, D). Elles s’organisent en feuillet de cellules (A, C, D E) ou en tubule (B) et se regroupent en monocouche (A à D) ou en multicouche (E). Tiré de Pearson Education Inc
Les tissus épithéliaux garantissent au milieu intérieur une protection contre les stress mécaniques, l’infiltration de liquides, et l’entrée de pathogènes. Ils servent également à transporter différentes molécules à travers les compartiments extérieur et intérieur de l’organisme. Cette fonction est possible grâce à leur capacité d’absorption et de sécrétion de part et d’autre de l’épithélium. Par exemple, l’épithélium intestinal permet l’absorption des nutriments, et l’épithélium pulmonaire sécrète du mucus.
1.2 Les jonctions cellules-‐cellules
Chez les Vertébrés, les jonctions adhérentes (JA) assurent la cohésion des tissus épithéliaux et la résistance face aux stress mécaniques (Fig 2). La perméabilité des tissus épithéliaux est garantie par les jonctions serrées situées au-‐dessus des jonctions adhérentes.
Chez la drosophile, les JA sont conservées mais les jonctions serrées n’existent pas (Fig 2). En revanche, une autre structure est capable d’assurer l’étanchéité du tissu, ce sont les jonctions septées (JS) (Tepass & Hartenstein 1994). Elles sont également capables de réguler le passage de solutés à travers l’espace intercellulaire (Skaer et al 1987). Les jonctions septées sont situées en dessous des jonctions adhérentes.
Figure 2 : Schéma des structures jonctionnelles chez les Vertébrés et Drosophila
Les jonctions adhérentes sont conservées des Vertébrés à l’espèce Drosophila, alors que les jonctions serrées sont retrouvées uniquement chez les Vertébrés. Chez la drosophile, les jonctions septées sont l’équivalent fonctionnel des jonctions serrées. Adapté de Tepass 2003
Chez la drosophile, les jonctions adhérentes attachent fermement deux cellules voisines grâces à l’interaction homotypique de la DE-‐Cadherin (DE-‐Cad, appelée également Shotgun (Shg) (Oda et al 1994 ; Shapiro et al 1995) (Fig 3). Le domaine cytoplasmique de DE-‐Cad est indirectement lié au cytosquelette d’actine via les protéines adaptatrices Armadillo (Arm) (Pfeifer et al 1993 ; Cox et al 1996) et alpha-‐ Catenin (α-‐Cat) (Oda et al 1993) (Fig 3). Cela permet de stabiliser les interactions homotypiques de la DE-‐Cad (Nagafuchi et al 1994) et de renforcer les jonctions adhérentes (Osawa et al 1990). Ces protéines sont structuralement et fonctionnellement conservées de la drosophile à l’humain (Tab 1).
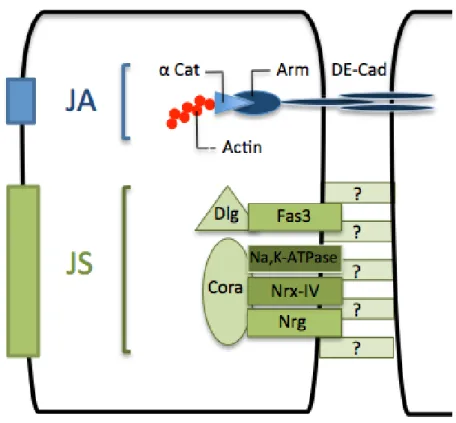
Les jonctions septées sont distinguables par microscopie électronique car elles forment une structure en échelle (Tepass & Hartenstein 1994). Les protéines transmembranaires Neurexin IV (Nrx-‐IV) (Baumgartner et al 1996), la sous-‐unité alpha de la pompe sodium potassium (Na,K-‐ATPase) (Paul et al 2003 ; Genova & Fehon 2003) et Neuroglian (Nrg) (Genova & Fehon 2003) forment la structure de base
Vertébrés Jonctions serrées Jonctions adhérentes Jonctions septées Drosophila
des jonctions septées (Fig 3). Ces protéines interagissent avec Coracle (Cora) (Fehon et al 1994 ; Lamb e al 1998) qui est indispensable à la stabilité du complexe (Ward et al 1998 ; Genova & Fehon 2003). On retrouve également la protéine Discs Large (Dlg) (Woods & Bryant 1991 ; Woods et al 1996) dont la présence est indispensable à la localisation de Nrg et d’une autre protéine transmembranaire enrichie aux jonctions septées, Fasciclin 3 (Fas3) ( Patel et al 1987 ; Snow et al 1989) (Fig 3).
Figure 3 : Composition des jonctions adhérentes et septées chez la Drosophile
Les jonctions adhérentes sont formées des interactions homotypiques de DE-‐cad reliées au cytosquelette grâce aux adaptateurs cytoplasmiques Arm et α-‐Cat. Les jonctions septées sont composées des protéines transmembranaires Na,K-‐ATPase, Nrx-‐IV, Nrg assemblées en complexe avec la protéine cytoplasmique Cora. On retrouve également la protéine transmembranaire Fas3 qui interagit avec Dlg.
1.3 La polarité apico-‐basale
Les épithéliums assurent des fonctions spécifiques selon qu’ils échangent avec le milieu intérieur ou extérieur. C’est pourquoi leurs composantes cellulaires sont distribuées de manière asymétrique selon deux domaines distincts. On retrouve le domaine apical qui fait face à l’extérieur, représenté le plus souvent par la lumière des organes (Fig 4). La région située directement au-‐dessus des jonctions adhérentes est appelée zone marginale. En dessous des jonctions adhérentes, il y a le domaine basolatéral. On distingue la partie latérale retrouvée à l’interface cellule-‐cellule et la partie basale qui est ancrée à la lame basale. Cette architecture polarisée est appelée polarité apico-‐basale ou polarité épithéliale.
Figure 4 : Schéma d’un épithélium simple polarisé
Les composantes cellulaires des tissus épithéliaux sont réparties selon un domaine apical et basolatéral séparés par les jonctions adhérentes. On peut diviser la membrane en 4 quatre portions : apicale (rouge), zone marginale (orange), latérale (vert) et basale (vert foncé).
1.4 Les modules de régulation de la polarité apico-‐basale
La polarité apico-‐basale repose sur l’action concertée de nombreux modules protéiques. Les modules d’un même domaine coopèrent fonctionnellement afin d’antagoniser l’activité des modules du domaine opposé (Laprise et al 2011) (Fig 5).
Au domaine apical, on retrouve le complexe Crumbs (Crb) composé des protéines Stardust (Sdt), Patj et Crb (Bulgakova et al 2009) ainsi que le complexe Par formé par les protéines Atypical protein kinase C (aPKC), Cell division cycle protein 42 (Cdc42) et Partitioning defective 6 (Par-‐6) (Joberty et al 2000) (Fig 5). Ces deux complexes interagissent positivement ensemble pour promouvoir la formation et la croissance du domaine apical (Hurd et al 2003 ; Sotillos et al 2004). Ils inhibent les complexes basolatéraux (Bilder et al 2003 ; Tanentzapf & Tepass 2002 ; Betschinger et al 2003 ;) et favorisent le positionnement des complexes jonctionnels (Grawe et al 1996 ; Hurd et al 2003). On les retrouve accumulés au niveau de la zone marginale.
Le domaine basolatéral possède également deux modules dont le groupe
Scribbled (Scrib) assemblé de Dlg, Lethal giant larvae (Lgl) et Scrib (Bilder &
Perrimon 2000) et le groupe Yrt/Cora (Yrt) constitué de Na,K-‐ATPase, Nrx-‐IV, Cora et Yrt (Laprise et al 2009) (Fig 5). Ces complexes favorisent l’identité de la membrane basolatérale et inhibent les complexes apicaux (Bilder et al 2002 ; Tanentzapf and Tepass 2002 ; Laprise et al 2006 ; Laprise et al 2013). Ils contribuent également au positionnement des jonctions adhérentes (Tanentzapf and Tepass 2002) et à la formation des jonctions septées (Baumgartner et al 1996 ; Lamb et al 1998 ; Paul et al 2003). Le groupe Scrib inhibe la machinerie apicale pendant la gastrulation de l’embryon, alors que le groupe Yrt/Cora le remplace fonctionnellement pendant l’organogenèse (Laprise et al 2009).
7 Crb Sdt Patj Cdc42 Par6 aPKC Baz Par1 LglDlg
Scrib Yurt Cora
Nrx-IV NaK-ATPase
AJ AJ
À l’interface des domaines apicaux et basolatéraux, Bazooka (Baz) est associée aux jonctions adhérentes. L’accumulation apicale de Bazooka dans les stades précoces de l’embryogenèse est le premier signal connu indispensable à la mise en place des jonctions adhérentes (Harris et al 2004) et de la polarité épithéliale (Harris et al 2005) (Fig 5). Baz favorise le recrutement apical de ses partenaires d’interaction aPKC (Wodarz et al 2000), Par-‐6 (Petronezki & Knoblich 2001), et Sdt (Krahn et al 2010), ce qui facilite, entre autres, la formation du complexe Crb. L’exclusion de Baz du domaine apical intervient suite à sa phosphorylation par aPKC et nécessite la présence de Crb (Morais-‐de-‐Sa et al 2010 ; Walther & Pichaud 2010), alors que son exclusion du domaine basolatéral est assurée par la kinase Protease activated receptor 1 (Par-‐1) (Benton & Johnston 2003). Cela va permettre la localisation de Baz aux jonctions adhérentes et favoriser la ségrégation entre les domaines apicaux et basolatéraux
Figure 5 : Schéma des modules de polarité présents dans les cellules épithéliales de drosophile.
Le domaine apical est composé des modules Crb (Crb-‐Stdt-‐Patj) Par (Par-‐6-‐aPKC-‐Cdc42) et de la protéine Baz, alors que le domaine basolatéral est formé des groupes Scrib (Scrib-‐Dlg-‐Lgl) et Yrt/Cora (Yrt-‐Cora-‐Nrx-‐IV-‐Na,K-‐ ATPase) ainsi que de la kinase Par-‐1. Tiré de Laprise et Tepass 2011
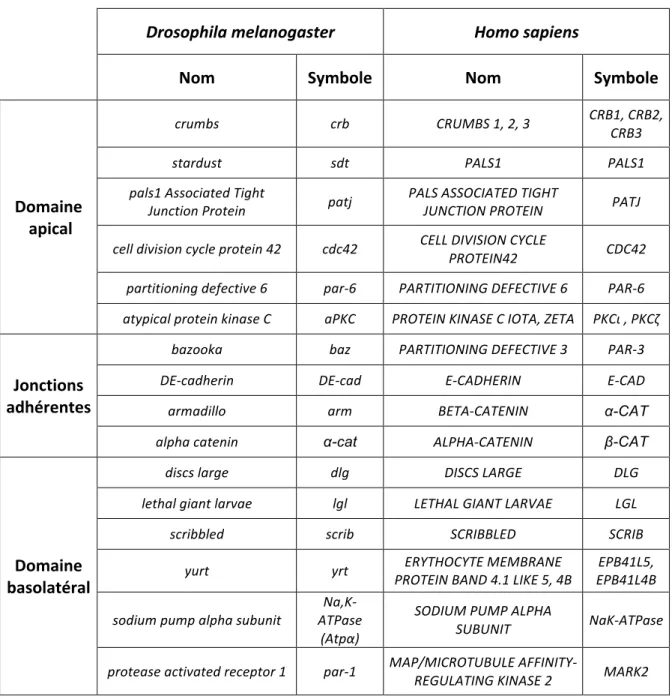
Les mécanismes orchestrant l’établissement et le maintien de la polarité épithéliale sont majoritairement conservés de la drosophile à l’humain. En effet, de nombreux gènes de polarité chez la drosophile ont des orthologues chez l’humain (Tab 1). De plus, on retrouve une forte conservation fonctionnelle et structurale de ces protéines de polarité (Ohno 2001 ; Roh et al 2003 ; Yamanaka et al 2003 ; Qin et al 2005).
Drosophila melanogaster Homo sapiens
Nom Symbole Nom Symbole
Domaine apical
crumbs crb CRUMBS 1, 2, 3 CRB1, CRB2, CRB3
stardust sdt PALS1 PALS1 pals1 Associated Tight
Junction Protein patj PALS ASSOCIATED TIGHT JUNCTION PROTEIN PATJ cell division cycle protein 42 cdc42 CELL DIVISION CYCLE PROTEIN42 CDC42
partitioning defective 6 par-‐6 PARTITIONING DEFECTIVE 6 PAR-‐6 atypical protein kinase C aPKC PROTEIN KINASE C IOTA, ZETA PKCι , PKCζ
Jonctions adhérentes
bazooka baz PARTITIONING DEFECTIVE 3 PAR-‐3 DE-‐cadherin DE-‐cad E-‐CADHERIN E-‐CAD armadillo arm BETA-‐CATENIN α-CAT alpha catenin α-cat ALPHA-‐CATENIN β-CAT
Domaine basolatéral
discs large dlg DISCS LARGE DLG lethal giant larvae lgl LETHAL GIANT LARVAE LGL scribbled scrib SCRIBBLED SCRIB
yurt yrt PROTEIN BAND 4.1 LIKE 5, 4B ERYTHOCYTE MEMBRANE EPB41L4B EPB41L5,
sodium pump alpha subunit ATPase Na,K-‐ (Atpα)
SODIUM PUMP ALPHA
SUBUNIT NaK-‐ATPase
protease activated receptor 1 par-‐1 MAP/MICROTUBULE AFFINITY-‐REGULATING KINASE 2 MARK2
Tableau 1 : Conservation des protéines de polarité de l’humain à la drosophile De nombreux gènes de polarité sont conservés structuralement et fonctionnellement de l’Homo sapiens à
1.5 La protéine Yrt
1.5.1 Structure de la protéine Yrt
Le gène yrt est conservé dans 131 espèces dont Drosophila melanogaster (Ensembl Genome Browser). La région codante est composée de 10 exons dont sont issus 3 transcrits. Le gène pleine longueur donne lieu au transcrit beta, alors que les transcrits gamma et delta sont issus respectivement de l’excision des exons 6 et 9, suite à des évènements d’épissage alternatif. La protéine Yrt possède quatre isoformes d’un poids moléculaire de 84 kDa (delta), 101 kDa (gamma), 106 kDa (beta) et 135 kDa. L’isoforme le plus haut n’a pas de transcrit connu. Cependant il apparaît lorsque l’on surexprime l’isoforme beta ce qui indique qu’il est une forme modifiée post-‐ traditionnellement de ce dernier. L’expression des quatre isoformes de Yrt est régulée différentiellement au cours de l’embryogenèse (Laprise et al 2006).
La protéine Yrt comporte dans sa portion N-‐terminale un domaine FERM (4.1, Ezrin, Radixin and Moesin) de 277 acides aminés (acide aminé 62 à 339) (Fig 6). Le domaine FERM est partagé par une famille de protéines qui porte le même nom et assurent des fonctions structurales, signalétiques, ou de transport intracellulaire. Elles permettent de relier des protéines cytoplasmiques à la membrane et au cytosquelette grâce à des interactions protéiques et protéines-‐lipides (Chisti et al 1998 ; Tepass 2009). Le domaine FERM de Yrt permet son interaction directe avec le FBM (FERM Binding Motif) de Crb (Laprise et al 2006).
En aval du domaine FERM, on retrouve un domaine FA (FERM-‐Adjacent) de 43 acides aminés (acide aminé 352 à 395) (Fig 6). Ce domaine est connu pour réguler l’activité des protéines FERM par sa phosphorylation. En effet, il présente de nombreux sites de phosphorylation par les protéines kinase A et C (Baines et al 2006). L’activité de la protéine Yrt est régulée par phosphorylation de son domaine FA par aPKC (Nakajima et al 2011 ; Gamblin et al 2013).
On retrouve également un domaine PDB (PDZ Binding site) à l’extrémité C-‐ terminale de la protéine Yrt. Ce domaine favorise les interactions protéiques via sa liaison aux domaines PDZ (Post synaptic density protein (PSD95), Drosophila Discs large tumor suppressor (Dlg), and Zonula occludens-‐1 protein (Zo-‐1), (Hoover et al 2002). Aucun interacteur de Yrt dépendant de ce domaine n’a été identifié à ce jour.
Figure 6 : Schéma représentant les domaines protéiques de Yrt.
La protéine Yrt contient un domaine FERM structuré en 3 lobes (F1, F2, F3), un domaine FA (FERM Adjacent) et un domaine PDB (PDZ Binding Site) Tiré de Tepass et al 2009
1.5.2 Fonctions de Yrt dans la polarité épithéliale
Yrt est un déterminant basolatéral appartenant au groupe Yrt/Cora (Laprise et al 2009). Lors de l’établissement de la polarité épithéliale, Yrt est localisée à la membrane basolatérale. Cette localisation est dépendante de son exclusion du domaine apical suite à sa phosphorylation par aPKC (Gamblin et al 2013). Yrt interagit fonctionnellement avec les membres de son groupe (Cora, Nrx-‐IV, Na,K-‐ATPase) pour réguler l’établissement de la polarité apico-‐basale (Laprise et al 2009). Yrt promeut l’identité de la membrane basolatérale par l’inhibition de la machinerie apicale. La perte de Yrt entraine une délocalisation des marqueurs de polarité dès le stade 11 de l’embryogenèse et provoque une létalité embryonnaire (Nüsslein-‐Volhard et al 1984 ; Hoover & Bryant 2002 ; Laprise et al 2006).
À partir du stade 14 de l’embryogenèse, Yrt est partiellement recrutée au domaine apical ou elle colocalise et interagit avec Crb pour réguler négativement son activité (Laprise et al 2006). Cela permet de limiter la croissance de la membrane apicale. La fraction basolatérale de Yrt restante est enrichie aux jonctions septées et favorise l’étanchéité des jonctions (Laprise et 2009). La présence de Yrt n’est pas indispensable à l’intégrité structurale des jonctions septées à l’inverse des autres membres du groupe Yrt/Cora (Baumgartner et al 1996 ; Lamb et al 1998 ; Paul et al 2003).
1.5.3 Fonctions d’endocytose de Yrt
Des résultats préliminaires du laboratoire soulèvent l’hypothèse que Yrt pourrait réguler négativement l’activité de Crb en favorisant son endocytose. Lorsque l’on surexprime une version de Yrt qui ne peut pas être exclue du domaine apical par aPKC, la taille du domaine apical est fortement diminuée. De plus, on retrouve une accumulation cytoplasmique d’agrégats de Crb dont le nombre augmente si on bloque la dégradation du lysosome.
En outre, plusieurs protéines associées à l’endocytose ont été identifiées comme étant des interacteurs physiques de Yrt par spectrométrie de masse. On retrouve quatre protéines adaptatrices (AP) et la Clathrin qui sont deux composantes majeures des vésicules d’endocytose (Mcmahon 2013).
Pour finir, il a été montré que le complexe Par inhibe l’endocytose de Crb et que ses fonctions sont dépendantes d’aPKC (Harris et al 2008). Cependant les cibles d’aPKC sont inconnues. Puisque qu’aPKC régule négativement Yrt (Gamblin et al 2013), il est possible d’émettre l’hypothèse qu’aPKC limite l’endocytose de Crb en inhibant les fonctions de Yrt.
1.5.4 Implication de Yrt dans le cancer
Yrt possède deux orthologues chez l’humain et la souris, les protéines EPBL41B appelée également EHM2, et EPB41L5 appelée également YMO1. Différentes études ont montré qu’EHM2 exerce des fonctions pro-‐métastatiques. L’expression d’EHM2 est associée à un fort potentiel métastatique des cellules issues de mélanome de souris (Hashimoto et al 1996 ; Shimizu et al 2000). La protéine humaine EHM2 est surexprimée dans les cancers de la prostate et diminue l’adhésion au collagène des cellules cancéreuses (Wang et al 2006). Cela favorise le détachement des cellules et accroit leur potentiel métastatique. La surexpression d’EHM2 a également été mis en évidence dans le cancer du sein, et associée à des cancers métastatiques avec un faible pronostique de survie. EHM2 favorise l’invasion des cellules cancéreuses du sein en facilitant la dégradation de la matrice extracellulaire, via l’augmentation de la transcription des métalloprotéases MMP9 (Yu et al 2010).
Chez la drosophile, Yrt pourrait favoriser la progression tumorale via sa capacité à réguler négativement Crb. En effet Crb est un potentiel suppresseur de tumeur (Karp et al 2008) dont la baisse de l’expression est indispensable à la poursuite de la transition épithélio-‐mésenchymateuse (EMT) (Campbell et al 2011).
1.6 La protéines Cérès
Chez l’humain, la protéine Cérès connue sous le nom d’ALBATROSS (ALB) a un poids moléculaire de 130 kDa et possède un domaine homologue à la trychohyalin et à la plectin (THPD) (Schnidt et al 2000). En association avec PARTIONING DEFECTIVE 3 (PAR-‐3), ALB favorise la mise en place des complexes jonctionnels apicaux (jonctions serrées et adhérentes), et maintien l’identité de la membrane basolatérale (Sugimoto et al 2008) (Fig 7). Lorsque l’expression d’ALB est diminuée par knockdown dans des cellules cancéreuses, PAR-‐3 et d’autres composantes jonctionnelles ne sont plus localisées aux jonctions. De plus, certains déterminants basolatéraux sont délocalisés
au domaine apical. Les fonctions d’ALB sont stabilisées par son interaction avec les filaments intermédiaires de kératine qu’elle lie via son domaine THPD.
Figure 7 : Schéma représentant les fonctions de Cérès dans les cellules cancéreuses humaines.
ALB favorise l’activité de PAR-‐3 dans la mise en place du complexe jonctionnel apical. En revanche, la perte d’ALB (Albatross KD) n’affecte pas les fonctions de PAR-‐3 dans la formation du domaine apical. Les fonctions d’ALB sont stabilisées par son interaction avec les filaments de kératine via son domaine THPD. Tiré de Sugimoto et al 2008
Chez la drosophile, la protéine CG5964 que nous avons renommée Cérès a été identifiée par criblage comme étant un régulateur de la sécrétion de Hedgehog (Hh, Aikin et al 2011). Des expériences réalisées en cellules de drosophile S2 suggèrent que Cérès participe au transport vésiculaire entre le réticulum endoplasmique et le golgi. Le knockdown de Cérès dans un tissu spécifique de la mouche diminue l’activité et délocalise la protéine Hh en plus de diminuer la sécrétion apicale de Wingless (Wg). Ces résultats suggèrent une implication de Cérès dans le transport vésiculaire et la sécrétion apicale.
1.7 Présentation du modèle d’étude : Drosophila melanogaster
Drosophila melanogaster, appelée communément mouche à fruit ou drosophile,
est un modèle génétique puissant largement utilisé à travers le monde depuis une centaine d’années. La drosophile ne possède que 4 chromosomes sur lesquels sont répartis environ 14 600 gènes. Le séquençage complet du génome de drosophile (Adams et al 2000) a mis en évidence un fort pourcentage de conservation avec le génome humain. Il est établit que 62% des gènes responsables de maladies chez l’humain sont retrouvés chez la drosophile (Fortini et al 2000).
Une des plus grandes forces de la drosophile est son utilisation pour les criblages génétiques (Saint Johnson 2002) qui a permis la découverte de nouveaux gènes impliqués dans différents processus cellulaires (Nüsslein-‐Volhard & Wieschaus 1980). C’est ainsi que de nombreux mécanismes ont été découverts chez la drosophile avant d’être confirmés chez l’humain (Holley et al 1995).
Plusieurs techniques développées chez la drosophile permettent d’induire facilement la perte ou la surexpression d’un gène et d’en caractériser les conséquences physiologiques dans un tissu spécifique ou l’ensemble de la drosophile. Ces manipulations génétiques sont facilement traçables au travers des générations grâce à l’observation de traits phénotypiques (couleur des yeux, taille des ailes, forme des poils). De plus, les drosophiles sont capables d’assurer une large progéniture en seulement 10 jours et ne coûtent par cher à entretenir, ce qui en fait un modèle très pratique à utiliser.