Bioaérosols viraux dans les usines de traitement des eaux usées :
détection moléculaire et métagénomique
Mémoire Evelyne Brisebois Maîtrise en microbiologie Maîtrise ès sciences (M. Sc.) Québec, Canada © Evelyne Brisebois, 2016
Bioaérosols viraux dans les usines de traitement des eaux usées :
détection moléculaire et métagénomique
Mémoire
Evelyne Brisebois
Sous la direction de :
Caroline Duchaine, directrice de recherche Alexander Culley, codirecteur de recherche
iii
Résumé
Les travailleurs des centres de traitement des eaux usées sont souvent atteints de maladies reliées à l’exposition virale telles que le rhume, la grippe et les infections gastro-intestinales. Dans la littérature, plusieurs études ont caractérisé les bactéries présentes dans les eaux usées ainsi que les bactéries dans les aérosols de ces centres de traitement. Les virus dans les eaux usées ont aussi été dans la mire de certaines études. Cependant, malgré le fait que les maladies affectant les travailleurs soient majoritairement de cause virale et que certains virus pathogènes aient été démontrés pour être capables de se transmettre par l’air, seule une minime quantité d’études a décrit jusqu’à présent les aérosols viraux de ces environnements. Qu’en est-il des virus aérosolisés qui peuvent provoquer ces maladies chez les travailleurs? Auxquels virus potentiellement pathogènes les travailleurs sont-ils exposés chaque jour dans ces environnements de travail? Cette étude avait pour but d’échantillonner l’air de quatre centres de traitement des eaux usées à l’aide de différents échantillonneurs pour décrire la communauté virale dans les aérosols et, ultimement, d’identifier les virus aérosolisés qui peuvent mettre en danger la santé des travailleurs. Onze virus ont été sélectionnés en fonction de leur présence dans les eaux usées et quantifiés de manière spécifique par une technique de biologie moléculaire appelée qPCR. De plus, une étude métagénomique a été effectuée sur des échantillons d’air de très grands volumes dans l’une des usines à l’étude afin de caractériser les communautés virales totales présentes dans l’air des centres de traitement. Les échantillons d’air ont été pris à deux reprises dans chaque centre, une fois durant la saison d’été et une fois durant la saison d’hiver afin de déterminer l’effet de la température de l’eau sur le type de virus aérosolisés.
iv
Abstract
Wastewater treatment center (WTCs) workers may be vulnerable to diseases caused by viruses, such as the common cold, influenza and gastro-intestinal infections. Although there is a substantial body of literature characterizing the microbial community found in wastewater, only a few studies have characterized the viral component of WTC aerosols, despite the fact that most diseases affecting WTC workers are of viral origin and that some of these viruses are transmitted through the air. In this study, we evaluated in four WTCs the presence of 11 viral pathogens of particular concern in this milieu and used a metagenomic approach to characterize the total viral community in the air of one of those WTCs. The presence of viruses in aerosols in different locations of individual WTCs was evaluated and the results obtained with four commonly used air samplers were compared. We detected four of the eleven viruses tested, including human adenovirus (hAdV), rotavirus, hepatitis A virus (HAV) and Herpes Simplex virus type 1 (HSV1). The results of the metagenomic assay uncovered very few viral RNA sequences in WTC aerosols, however sequences from human DNA viruses were in much greater relative abundance.
v
Table des matières
Résumé ... iii
Abstract ... iv
Table des matières ... v
Liste des tableaux ... vii
Liste des figures ... viii
Liste des abréviations et des sigles ... ix
Remerciements ... x
Avant-propos... xii
Chapitre 1 : Introduction ... 1
Les aérosols ... 3
La formation des aérosols et leur évolution dans le temps ... 3
Le comportement des particules aérosolisées et les forces agissant sur ces particules ... 5
Les bioaérosols ... 7
L’aérovirologie ... 9
Les centres de traitement des eaux usées ... 10
Les eaux usées et les processus d’épuration ... 10
Les contaminations dans l’eau et les bioaérosols des usines ... 11
La description des centres de traitement au Québec ... 12
Le syndrome de l’égoutier et autres maladies associées ... 13
Les virus à l’étude ... 14
La métagénomique ... 14
Principe ... 14
La métagénomique virale ... 15
Les applications pour l’air jusqu’à maintenant ... 16
Les adaptations pour les échantillons d’air ... 17
La problématique, les objectifs et les hypothèses des travaux de recherche... 18
La problématique ... 18
Les objectifs ... 19
vi
Chapitre 2 : Exposition professionnelle aux bioaérosols viraux dans les usines de traitement
des eaux usées : détection moléculaire et métagénomique ... 22
2.1. Article: Human viral pathogens are pervasive in wastewater treatment centers aerosols ... 23 Résumé ... 24 Abstract ... 25 Introduction... 26 Methods ... 28 Results ... 32
Discussion and conclusion ... 37
Conflict of interests ... 41 Acknowledgements ... 42 Appendix ... 43 References ... 44 Figure Legend ... 48 Chapitre 3 : Discussion ... 50 3.1. qPCR ... 52 3.2. Métagénomique ... 54
Chapitre 4 : Conclusion et perspectives ... 56
Bibliographie ... 60
Annexe ... 70
vii
Liste des tableaux
Tableau 1-1. Vitesse de déposition des aérosols dépendamment de leur diamètre aérodynamique.
Tableau 1-2. Concentration de micro-organismes mesurés dans différents environnements de travail.
Tableau 1-3. Catégories de contamination environnementale de bactéries et d’endotoxines dans différents environnements.
Tableau 1-4. Usines et sites à l’étude pour ce projet. Table 2-1. Description of WTCs and sites of the study.
Table 2-2. Primers and probes used for qPCR detection of selected viruses.
Table 2-3. Positive viral amplifications (in gene copies/m3 of air) from air samples of Coriolis, SASS 2300 and SASS 3100 in WTCs.
Table 2-4. Assay quantification limits.
Table 2-5. Detection and quantification of rotaviruses with the Marple sampler. Table 2-6. Sequencing and assembly output details.
Table 2-7. Families represented in the group “other” of graph B with their respective percentage.
viii
Liste des figures
Figure 1-1. Processus d’assèchement des aérosols et l’obtention des noyaux de gouttelettes.
Figure 1-2. Émission de particules de diamètres aérodynamiques variés d’une personne toussant.
Figure 1-3. Schéma des étapes de traitement et d’assainissement des eaux usées.
Figure 2-1. Air samples from WTC 4 (A) Classification of assigned sequences at a domain taxonomic level, (B) Classification of virus-assigned sequences by Family, (C) Hosts of viruses based on the Figure 2-1B assignments.
Figure 2-2. Distribution of human viruses in samples from sampling locations 1 (A), 2 (B) and 3 (C) from WTC 4.
Figure 2-3. Heat map comparing metagenomic results from site 1, 2 and 3 of WTC 4 with samples from wastewater and source water in North America (public databases). The red scale represents low abundance cases, as the green one represents high abundance cases.
ix
Liste des abréviations et des sigles
ADN : Acide désoxyribonucléique
ADNc : ADN complémentaire synthétisé à partir d’une molécule d’ARN AdV : Adénovirus
ARN : Acide ribonucléique
CTEU : Centre de traitement des eaux usées
ELPI®+ : Impacteur électrique à basse pression (Electrical Low Pressure Impactor)
EV : Entérovirus
HAV : virus de l'hépatite A HRV : Rhinovirus humain
HSV-1 : Virus de l’herpès simplex (Herpes Simplex Virus) de type 1 HSV-2 : Virus de l’herpès simplex (Herpes Simplex Virus) de type 2 InfA : Virus de l’Influenza A
InfB : Virus de l’Influenza B
IUCPQ : Institut universitaire de cardiologie et de pneumologie de Québec nm : nanomètre (10-9 m)
NoV GI : Norovirus génogroupe I NoV GII : Norovirus génogroupe II
qPCR : Réaction en chaîne par polymérase (quantitative)
qRT-PCR : Transcription inverse suivie d’une réaction en chaîne par polymérase (quantitative) µm : micromètre (10-6 m)
x
Remerciements
C’est avec le cœur gros que j’écris cette page aujourd’hui. Caroline, ma mentore, mon amie, je ne trouve pas les mots pour exprimer mon niveau de gratitude envers toi. On ne rencontre pas souvent des personnes marquantes comme toi dans une vie. Tu as une incroyable force positive autour de toi et tu as toujours su trouver les mots pour nous encourager et nous surpasser et à donner le meilleur de nous-mêmes. Je te remercie du fond du cœur pour tous ces beaux moments de complicité passés ensemble, ces fous-rires, ces discussions profondes et toutes les choses que tu m’as apprises, autant au niveau professionnel que personnel. C’est triste que mon parcours professionnel se termine ici avec toi, mais je sais que notre amitié ne se termine certainement pas là. Merci mille fois et je te dis à bientôt pour une autre discussion de chats!
Marc, Nathalie et Valérie, vous êtes extraordinaires. À vous trois, vous complétez à merveille le « team » Bioaérosols. Merci pour votre aide et tout le précieux temps que vous avez passé à me transmettre vos connaissances. J’en suis très reconnaissante.
Merci à Léa d’exister tout simplement. Tu as été le rayon de soleil de ma première année de maîtrise, toujours altruiste et souriante, toujours à voir le côté positif des choses, tu me fais du bien. J’apprécie tous les moments qu’on passe ensemble, que ce soit à parler de sciences ou de recettes! Tu es une amie en or, une âme sœur.
Merci à Gillina et Philippa, deux femmes d’exception que j’ai eu la chance d’avoir comme premières modèles en laboratoire. Merci à mes grandes amies et collègues Laetitia et Marie-Ève, avec qui j’ai aussi partagé des peines et de nombreuses joies. Nos discussions de chaussures, de vêtements, de garçons, de thés vont me manquer énormément. Un merci spécial à tous les autres membres du laboratoire : Vanessa, ma partenaire de sorties terrain et mon successeur de projet, Hamza, mon indien à la mode préféré, Jonathan pour toutes les photos de cochons et qui je sais au fond va s’ennuyer de moi au plus au point même s’il fait son « tough ». Ne pleure pas, je reviendrai vous voir.
Merci à mes parents et ma sœur, qui m’ont toujours encouragé et qui me prennent pour une star parce que j’ai fait une maîtrise, merci! Plus sérieusement, ils ont toujours été fiers de ce que j’accomplis et ç’a été une motivation de plus à réussir et donner mon maximum jusqu’au bout. Vous êtes une famille formidable et même si on est loin les uns des autres, on réussit à se parler presque tous les jours. Je vous aime.
Je tiens à remercier tous les membres de mon comité aviseur, Caroline Duchaine, Alexander Culley, Jacques Corbeil et Yann Gilbert, pour leur temps et leurs précieux conseils. Un énorme merci à l’Institut Robert-Sauvé en santé et sécurité du travail (IRSST) aussi qui m’a octroyé une bourse de maîtrise pour ce projet.
xii
Avant-propos
Le présent mémoire a été divisé en quatre sections. Tout d’abord, une introduction aborde l’avancement des connaissances actuelles sur l’aérobiologie, les centres de traitement des eaux usées et la métagénomique. Le chapitre deux représente les sections de méthodologie et de résultats présentées sous forme d’article scientifique ayant été soumis le 20 février 2017 dans le journal Journal of Environmental Sciences (numéro de manuscrit JES_2017_455). Finalement, une section de discussion et de conclusions générales revient sur l’ensemble des résultats présentés dans l’article. En annexe se retrouve une partie de l’introduction portant sur l’échantillonnage et la détection des virus aérosolisés, ayant été écrite en anglais par moi-même pour un chapitre de livre soumis dans le livre « Microbiology of aerosols », Anne-Marie Delort and Pierre Amato, eds. (en préparation), Le tableau ne sera pas publié, et le texte sera fractionné pour faire partie de trois chapitres distincts.
1
Chapitre 1 : Introduction
3
Les aérosols
La formation des aérosols et leur évolution dans le temps
Les aérosols représentent toutes les particules pouvant se retrouver en suspension dans l’air pour un certain temps. Il peut s’agir de particules de nature différente, par exemple de poussières, de pollens, de micro-organismes vivants ou morts et même de fragments de ceux-ci. Les aérosols vont souvent être un mélange de particules solides et liquides, tout dépendamment de la source d’aérosolisation de laquelle ils proviennent.
Les particules aérosolisées peuvent aussi être de différentes grosseurs, ou plus précisément de différents diamètres aérodynamiques. Lors de l’aérosolisation, les particules et micro-organismes présents à la source risquent d’être mis en suspension en gouttelettes, créant au départ de gros aérosols. Par la suite, si les aérosols créés sont assez petits pour demeurer en suspension dans l’air un certain temps, l’eau comprise entre les particules s’évaporera rapidement pour mener à une particule complètement sèche et de diamètre aérodynamique plus petit, appelée noyau de gouttelette. Les noyaux de gouttelettes peuvent alors rester en suspension dans l’air très longtemps et se disséminer sur de très longues distances.
Figure 1-1. Processus d’assèchement des aérosols et l’obtention des noyaux de gouttelettes. Figure tirée de [1].
4
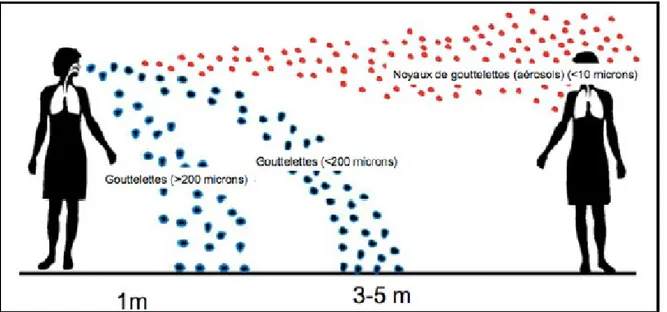
Figure 1-2. Émission de particules de diamètres aérodynamiques variés d’une personne toussant. Figure tirée de [2].
Dans cette figure, on peut observer la production d’aérosols par la toux d’une personne. D’abord, les grosses gouttelettes de diamètre aérodynamique plus élevé que 200 µm sédimenteront assez rapidement dans un rayon d’environ un mètre de la source. Les petites gouttelettes, plus petites que 200 µm, resteront en suspension dans l’air un peu plus longtemps pour aller sédimenter dans un rayon entre trois et cinq mètres de la personne. Finalement, les particules possédant un diamètre aérodynamique assez petit pour rester en suspension dans l’air assez longtemps pour s’assécher complètement et devenir des noyaux de gouttelettes resteront aérosolisées très longtemps. Ils pourront se disséminer sur des distances allant parfois jusqu’à plusieurs dizaines de kilomètres [3, 4], et infecter un nouvel hôte, si micro-organismes infectieux il y a.
L’humidité relative ambiante où se trouvent les aérosols aura un impact sur la grosseur de particules aérosolisées. Plus l’humidité relative est élevée, moins les aérosols se dessècheront rapidement, bien que ce processus soit toujours extrêmement rapide [5]. Le processus d’aérosolisation aura aussi un impact sur la nature et la quantité de particules émises. Il est donc possible que des aérosols de grosseurs et de compositions différentes proviennent d’une même source, mais que ceux-ci aient été aérosolisés à partir d’un processus différent. Par exemple, une personne toussant créera plus d’aérosols et de diamètres aérodynamiques plus gros que cette même personne qui respire normalement, car le processus d’aérosolisation, l’éternuement, est plus violent que la respiration normale [6].
5
Les sources d’aérosols peuvent être quasi infinies. Les processus d’aérosolisation peuvent être d’origine naturelle ou anthropogénique. De l’humain à l’industrie et en passant par les volcans, tout mouvement de matière est une éventuelle source d’aérosols. Bien entendu, les aérosols produits seront de la nature des molécules présentes à la source. Par exemple, l’épandage de fumier dans les champs émettra des particules et des micro-organismes présents dans la matière épandue et dans le sol, tandis qu’une personne atteinte d’une infection gastro-intestinale est plus susceptible d’émettre des particules virales desquelles elle est contaminée.
Le comportement des particules aérosolisées et les forces
agissant sur ces particules
Le comportement des aérosols dépend de plusieurs facteurs, dont l’un des plus importants est le diamètre aérodynamique. Le temps de résidence dans l’air des particules est variable. Comme mentionné précédemment, le diamètre aérodynamique d’une particule sera déterminé entre autres par le processus d’aérosolisation et l’humidité relative ambiante. Par la suite, il déterminera à son tour le comportement des aérosols quant aux mécanismes de capture auxquels ils seront soumis. Les particules de plus d’un µm de diamètre sédimenteront, tandis que les plus petits aérosols ne seront pas ou que très peu soumis à la force gravitationnelle.
Tableau 1-1. Vitesse de déposition des aérosols dépendamment de leur diamètre aérodynamique.a
6
Les particules de diamètre aérodynamique de moins de 1 µm se déposent très lentement, c’est-à-dire à moins de 8,8 x 10-7 m/s. Quatre autres mécanismes peuvent donc
intervenir sur les particules de diamètres aérodynamiques plus petits et qui ne sédimentent pas : l’impaction par inertie, l’interception, la diffusion et la précipitation électrostatique.
L’impaction inertielle est le processus par lequel les particules sont éliminées à partir d'un courant d'air en forçant les gaz à faire un virage. Une particule présente dans un courant d’air est capturée par impaction sur une surface alors que le gaz la transportant (air) change de trajectoire. Ainsi, la particule aérosolisée continue sa trajectoire initiale ou sera déviée seulement un peu et ira quand même s’impacter sur une surface. Une fois impactée, la particule sera retenue à la surface par des forces électrostatiques et de van der Waals [7]. Ce mécanisme est directement proportionnel à la masse des particules. Plus une particule est massive, plus elle aura tendance à conserver sa vitesse et direction initiales.
L’interception est le mécanisme impliqué lorsqu’une particule, suivant le courant d’air auquel elle est soumise, va s’impacter sur une surface ou un objet, car celui-ci se trouve dans sa trajectoire. Elle ne sera pas déviée du courant d’air comme par l’impaction par inertie; elle se déposera simplement sur l’objet en raison de sa grosseur et de la proximité entre les deux.
La diffusion représente quant à elle l’effet de l’agitation aléatoire des particules, mieux connue sous le nom de mouvement brownien. Ce phénomène se produit surtout avec les particules de diamètre aérodynamique de moins de 0,5 µm, car il s’agit du bombardement constant des molécules de gaz entourant l’aérosol. Ainsi, les particules aérosolisées ne sont que très peu soumises au courant d’air les entourant et se déplacent de façon aléatoire selon le bombardement qu’elles subissent. Ces bombardements peuvent pousser les aérosols à s’impacter sur une surface ou un objet quelconque. Lorsque soumis à ce phénomène, les aérosols auront tendance à se déplacer d’un environnement où la concentration en particules est élevée vers un environnement où elle est moindre.
Finalement, la précipitation électrostatique est dépendante de la charge des particules et des surfaces avoisinantes. Les charges opposées s’attirent et les charges semblables se repoussent. Si une molécule est chargée négativement, elle sera donc attirée par une surface chargée positivement et ira se déposer sur celle-ci, peu importe le courant
7
d’air auquel elle est soumise. Inversement, une particule chargée sera repoussée d’une surface partageant la même charge.
Les bioaérosols
Les bioaérosols représentent tous les aérosols d’origine biologique. Par exemple, il peut s’agir de virus, de bactéries, de moisissures, mais aussi de fragments de ceux-ci comme des membranes, des acides nucléiques et des toxines. Les bioaérosols peuvent être de différents diamètres aérodynamiques tout comme les aérosols, et sont régis par les mêmes lois de déposition et de capture. Dans le cas où les micro-organismes retrouvés sur certaines particules soient encore infectieux, il est intéressant de tirer profit des mécanismes de déposition des particules en forçant leur capture sur des surfaces.
En effet, plusieurs études ont rapporté la survie des bactéries et l’infectivité des virus après leur récupération sous forme d’aérosols [8-11]. Les virus ont aussi été démontrés pour demeurer infectieux sur des particules de différents diamètres aérodynamiques, signifiant que toutes les grosseurs de bioaérosols peuvent avoir un effet sur la santé des êtres susceptibles de les respirer [12-14]. Les bioaérosols représentent une menace implacable pour la transmission des maladies; il est difficile de se protéger d’une menace qu’on ne peut percevoir. Récemment, plusieurs études se sont intéressées à l’exposition professionnelle aux bioaérosols [15-17], car différentes maladies souvent retrouvées chez certains groupes de travailleurs pourraient être causées par la transmission des bioaérosols.
8
Tableau 1-2. Concentration de micro-organismes mesurés dans différents environnements de travail.†
Milieu de travail Bactéries totales (UFC/m³)a Bactéries à Gram négatif (UFC/m³) Actinomycètes thermophiles (UFC/m³) Moisissures (UFC/m³) Extérieur 102 101 101 103 Agriculture (normal) 107 103 103 103-4 Agriculture (foins moisis) 109 103 109 109 Boulangerie 102-3 Centre de compostage 105 102 104 104 Centre d’épuration
des eaux usées 10
4 104 100 103 Champignons (compost) 106 -b 107 104 Champignons (culture) 103 - 102 102 Déchets domestiques (collecte) 104 103 103 104 Édifice à bureaux 102 101 101 102-3 Effluents des papetières 104 103 101 104 Fluides de coupe 106 104 - 105 Humidificateur 103 103 - 102-3 Moulin à coton 105 104 105 103 Papetière 106 102-3 - 103 Porcherie 106 103-4 - 104 Scierie 104 103-4 103 106 Tourbière - - - 108 Transformation du sucre 105 103 102 103 Tri de déchets domestiques 104 103 100 104 Usine de tabac 103 102 - 104
a UFC/m3 = unité formatrice de colonie par mètre cube d’air b - = non documenté
† Tableau tiré de http://www.irsst.qc.ca/media/documents/pubirsst/T-23.PDF (Page
consultée le 31 mars 2016).
Le précédent tableau rassemble la concentration de quatre groupes de micro-organismes dans 20 différents environnements de travail, ainsi qu’à l’extérieur. Il est rapidement possible d’observer que le nombre de micro-organismes retrouvés d’un environnement à l’autre varie grandement; les édifices à bureaux présentent en moyenne 102 UFC/m3 d’air, alors qu’en agriculture la concentration peut monter jusqu’à 109 UFC/m3
9
des eaux usées, les travailleurs sont susceptibles d’être exposés à des concentrations importantes de bactéries et de moisissures, pouvant causer diverses maladies comme le syndrome toxique d’exposition aux poussières organiques (STEPO) [18, 19], une obstruction chronique des voies respiratoires [20], ainsi que des alvéolites allergiques extrinsèques [21, 22].
L’aérovirologie
La revue de la littérature portant sur l’aérovirologie a été incluse en annexe (annexe 1). Il s’agit d’un chapitre de livre dont je suis l’auteure principale, écrit en anglais et portant sur les présentes connaissances des aérosols viraux.
10
Les centres de traitement des eaux usées
Les eaux usées et les processus d’épuration
Les eaux arrivant dans les centres de traitement des eaux usées, ou les CTEU, sont des eaux très contaminées. Il s’agit des eaux d’usées résidentielles et industrielles qui sont amenées dans ces centres afin d’en retirer les contaminants et de les remettre en circulation en toute sécurité. Le traitement de ces eaux est divisé en plusieurs étapes ou procédés.
Premièrement, l’eau entre dans l’usine et arrive au dégrillage où les gros déchets comme les papiers, le bois et les plastiques sont retenus par des grilles. Le dessablage va ensuite retirer les matières abrasives comme le sable et la terre qui risquent de sédimenter et de boucher des pompes dans les étapes subséquentes. En même temps se fait le dégraissage qui, par injection de bulles d’air dans l’eau, va faire flotter les graisses à la surface sous forme d’écumes et qui sera enlevé à l’aide d’une écumoire. L’eau entre ensuite dans les bassins de décantation primaire où le temps de résidence est plus long et la matière fine n’ayant pas été retirée aux étapes précédentes va sédimenter et être retirée par pompage. Par la suite, le traitement secondaire commence et certaines étapes peuvent différer d’une usine à l’autre. Dans la première situation, le traitement biologique ou aussi appelé la biofiltration, l’eau arrive dans un bassin d'aération où des tapis de micro-organismes se nourrissent de la matière organique résiduelle dans l’eau. La clarification va ensuite recueillir sous forme de boues les matières polluantes agglomérées et les micro-organismes. Dans certaines usines, ces deux dernières étapes sont remplacées par des bassins de décantation secondaire où le temps de résidence est très long. Au lieu d’être digérées, les matières organiques restantes dans l’eau stagnante vont sédimenter selon le même principe que la décantation primaire. Le temps de résidence beaucoup plus long fait en sorte que plus de particules, et celles-ci plus petites, pourront se retrouver sous forme de boues au fond des bassins. Toutes les boues récoltées lors des différents traitements vont par la suite être incinérées ou réutilisées à des fins de compostage, par exemple.
11 Figure 1-3.
Schéma des étapes de traitement et d’assainissement des eaux usées. Figure adaptée de [23].
Plusieurs des étapes décrites incluent un brassage ou un bullage constant de l’eau, ce qui peut créer beaucoup d’aérosols. Comme l’eau est très contaminée en micro-organismes, ceux-ci risquent donc de se retrouver dans l’air. Dans les années passées, les installations des CTEU étaient plus souvent extérieures, afin de limiter le confinement des travailleurs et de laisser l’aération naturelle se charger des odeurs. Cependant, la création d’odeurs dans les quartiers avoisinants a mené à la construction de plus en plus de sites de traitement intérieurs. Le confinement des travailleurs est alors grandement augmenté et ceux-ci sont plus susceptibles d’être en contact avec des bioaérosols créés lors des étapes d’assainissement de l’eau.
Les contaminations dans l’eau et les bioaérosols des usines
Plusieurs ont tenté de déterminer la nature des contaminants biologiques dans les eaux usées, plus précisément les micro-organismes. Beaucoup d’études ont démontré une forte contamination en bactéries et en virus dans les eaux pénétrant dans les usines [24-29]. Des études ont aussi démontré que des bactéries se retrouvaient aérosolisées lors des différentes étapes du brassage des eaux et qu’elles pouvaient se retrouver en suspension dans l’air pendant plusieurs heures, les rendant encore plus susceptibles d’être inhalées
12
[30-33]. L’analyse des bioaérosols bactériens est beaucoup plus simple que celle des virus. La culture et l’identification des bactéries sont relativement faciles ; celles-ci croissent sur des milieux de culture et peuvent être par la suite identifiées par observation au microscope et par séquençage. Elles peuvent aussi être identifiées par qPCR spécifique sur des échantillons liquides sortis directement d’un échantillonneur, ou encore par séquençage de la région conservée 16S. Avec cette méthode, les résultats obtenus donnent de l’information sur plusieurs bactéries à la fois, sauvant beaucoup de temps, d’argent et de travail.
Pour ce qui est des virus, et surtout les virus humains, la culture est beaucoup plus fastidieuse. Comme les virus sont des parasites obligatoires, ils doivent absolument être à l’intérieur de leurs cellules hôtes pour se développer. De la culture cellulaire doit donc être entreprise pour être en mesure de cultiver les virus pathogènes humains d’intérêt. Des analyses qPCR spécifiques peuvent aussi être effectuées sur des échantillons liquides. Cependant, peu importe la méthode conventionnelle utilisée, un seul virus ne peut être identifié à la fois. Avec l’arrivée de la métagénomique virale, des analyses de masse peuvent maintenant être obtenues, mais aucune étude de ce type n’a été menée sur l’air des CTEU à ce jour. La lourdeur des analyses virales pourrait expliquer le manque de littérature sur les virus aérosolisés dans les CTEU. Quatre études métagénomiques ont néanmoins porté sur les virus des eaux usées jusqu’à présent [34-37], démontrant que la contamination virale de l’eau est extrêmement élevée.
En effet, très peu d’études se sont intéressées aux virus pouvant être aérosolisés de l’eau contaminée entrant dans les usines. En 1985, Fannin et al. ont détecté de très faibles concentrations d’entérovirus dans les sorties d’air d’un centre de traitement aux États-Unis [38]. Carducci et al. ont aussi démontré la présence d’entérovirus dans l’air de ces environnements en 1995 [39]. En 1999, Romano et al. ont isolé deux souches de réovirus et deux souches d’entérovirus [40]. Quant à eux, Masclaux et al. ont quantifié des adénovirus et des norovirus à partir d’échantillons d’air aussi en 2014 [41].
La description des centres de traitement au Québec
Au Québec, les CTEU sont très diversifiés en terme de procédés de traitement secondaire. Les quatre usines sélectionnées pour cette étude possédaient plusieurs caractéristiques différentes. Étant donné que le but de l’étude était avant tout de documenter l’exposition professionnelle aux bioaérosols viraux, seuls les sites intérieurs où des
13
travailleurs sont amenés à effectuer des tâches quotidiennement ont été sélectionnés pour l’analyse de l’air.
Tableau 1-4. Usines et sites à l’étude pour ce projet.
CTEU 1 Dégrillage Dessablage/dégraissage Biofiltration CTEU 2 Dégrillage Dessablage/dégraissage Biofiltration
CTEU 3 Dégrillage primaire
Dégrillage secondaire
CTEU 4 Dégrillage
Dessablage/dégraissage Décantation secondaire
Les CTEU 1 et 2 présentaient les mêmes sites intérieurs, tandis que les CTEU 3 et 4 possédaient des caractéristiques différentes des trois autres centres. Le CTEU 3 est un centre différent de ceux normalement rencontrés au Québec. Il ne possédait que deux sites intérieurs, soient les dégrillages primaire et secondaire.
Le syndrome de l’égoutier et autres maladies associées
Les travailleurs de ces usines font partie d’une catégorie de travailleurs plus à risque de contracter des infections respiratoires et gastro-intestinales que le reste de la population. Les quelques mesures de biosécurité déjà en place semblent être insuffisantes pour les protéger des risques biologiques auxquels ils sont exposés chaque jour. Seul le lavage des mains est proposé comme moyen de protection de la santé des travailleurs. Les maladies recensées dans cette population sont la majorité du temps reliées à une cause virale. En effet, ceux-ci ont 37% plus de chance de présenter des symptômes aigus de gastro-entériques, en plus de maux de têtes, de fièvres, de rhumes et de grippes plus fréquents [42-44]. Le terme de syndrome de l’égoutier a été utilisé pour décrire l’ensemble des symptômes ressentis par ces travailleurs [45].
14
Les virus à l’étude
La sélection des virus pour la partie sur la quantification moléculaire a été faite selon les critères suivants. Certains virus sont parfois retrouvés en grande concentration dans les eaux usées et les matières fécales, comme les norovirus de génotypes I (NoroGI) et II (NoroGII) [26, 28, 29], les adénovirus (AdV) [26, 28, 29], les entérovirus (EV) [26, 28, 29], les virus de l’herpès simplex de sérotypes 1 (HSV-1) et 2 (HSV-2) [34], les virus de l’hépatite A (HAV) [46, 47] et les rotavirus [48, 49]. De plus, ces virus sont connus comme cause de maladies respiratoires et entériques [50-63]. Étant donné leur importance dans la littérature en lien avec le genre de maladies rencontrées chez le syndrome de l’égoutier, il est probable que ces virus soient reliés au syndrome de l’égoutier. Les autres virus sélectionnés sont des virus dont l’incidence dans le domaine des maladies respiratoires et entériques est connue, comme les virus de l’influenza A et B [64-67] et les rhinovirus [68-72]. Les rhinovirus ont aussi été retrouvés dans des échantillons d’eaux usées [34]. Le virus de l’hépatite A avait aussi comme caractéristique une séropositivité élevée contre des antigènes de ce virus chez les travailleurs des CTEU [73-76]. Les travailleurs ont développé des anticorps contre ce virus ; ils ont donc été en contact d’une manière ou d’une autre avec celui-ci ou avec des fragments de ce virus.
La métagénomique
Principe
La métagénomique est une méthode développée il y a quelques années dans le but de séquencer des milliers de séquences d’ADN en même temps, provenant toutes d’un même environnement. Ces séquences sont obtenues des fragments de génomes de tous les organismes présents dans un échantillon et l’ensemble de ces séquences est appelé le métagénome [77]. Il est estimé que seulement 1% des bactéries environnementales sont cultivables par les méthodes de laboratoire, sous-estimant ainsi la diversité et l’abondance [78]. La métagénomique gagne de plus en plus en popularité. Elle a pour avantage de permettre la description de la fraction non-cultivable des microorganismes (tout en n’excluant pas la cultivable), puisqu’elle vise la détection du matériel génétique et non la capacité à croître en laboratoire. C’est une discipline qui fait appel à plusieurs domaines scientifiques. Les microbiologistes sont souvent appelés pour la partie pratique, c’est-à-dire
15
l’échantillonnage et le traitement des échantillons (préparation, extraction, etc.). Par la suite, le séquençage de l’ADN génère une quantité phénoménale de données pouvant aller jusqu’à des milliards de séquences [79]. Une expertise en bio-informatique est donc requise pour le tri et l’analyse de ces données dans l’ultime but de relier toutes les séquences obtenues à leur organisme de provenance. Ainsi, la métagénomique dresse le portrait des micro-organismes présents dans un échantillon et fournit aussi des renseignements quant à leur abondance relative. La métagénomique offre aussi le portrait complet du potentiel génétique d’un environnement.
La métagénomique virale
Grâce à leur petite taille, les virus peuvent être concentrés à partir d’un échantillon complexe. Lorsque l’on s’intéresse à la diversité des phages et des virus dans un échantillon, il est impossible de prédire l’ensemble des souches hôtes, rendant impossible la caractérisation complète par culture. La métagénomique virale permet donc d’outrepasser cette limite de la culture et de détecter un plus grand nombre de phages et de virus d’un échantillon. Par exemple, les dix-sept premiers génomes de phages de bactéries aquatiques séquencés l’ont été grâce à la métagénomique [80]. Au début de ce projet, l’hypothèse avait été émise qu’il soit possible de traiter les échantillons avant l’extraction des acides nucléiques dans le but d’enrichir la partie virale des échantillons d’air des usines d’épuration des eaux usées. De cette façon, toutes les séquences d’acides nucléiques générées proviendront presque exclusivement de virus, facilitant par la suite la tâche de bio-informatique. En effet, en diminuant la diversité d’un échantillon (en concentrant les virus par exemple), on s’assure que le séquençage s’effectue plus en profondeur. Comme l’eau usée est extrêmement contaminée en bactéries, il y aura une quantité faramineuse de bactériophages aussi, car ces virus se répliquent de façon exponentielle dans les bactéries. Les virus dont les hôtes ne sont pas retrouvés dans les eaux usées seront aussi présents, mais en quantité beaucoup moindre puisqu’il leur est impossible de se dupliquer. Il est donc logique de penser que la majorité des séquences virales qui seront retrouvées proviendront de bactériophages. Il est donc primordial de réduire au maximum la diversité microbienne des échantillons pour s’assurer de bien aller chercher les séquences de virus humains susceptibles de se retrouver dans la flore virale majoritairement composée de bactériophages. Il sera discuté plus loin que la séparation physique des virus afin de les concentrer dans un échantillon d’air ne peut pas être abordée comme dans le cas d’autres
16
types d’échantillons (eau par exemple) puisque les virus sont présents à l’intérieur de particules plus grosses dans l’air, rendant moins utile leur taille comme facteur d’enrichissement.
Les applications pour l’air jusqu’à maintenant
La métagénomique virale de l’air est une science nouvellement explorée. À la lumière de nos connaissances, seulement trois études se sont intéressées à caractériser la flore virale de l’air par séquençage et analyses métagénomiques jusqu’à présent. La première a été menée en 2012 par Whon et al. sur des échantillons d’air de trois milieux différents : un quartier résidentiel, un complexe industriel et une forêt. Les échantillons ont été pris à l’aide d’un cyclone liquide positionné à une hauteur d’un mètre par rapport au sol [81]. Seule la flore virale à ADN a été étudiée, et une majorité de phages et de virus de plantes a pu être observée. Les méthodes d’analyse consistaient à la microscopie électronique et le pyroséquençage. La deuxième, menée par Hall et al., s’est intéressée à la métagénomique des virus à ADN de l’air d’abattoirs de bovins et d’ovins en 2013 [82].Des échantillonneurs personnels à filtres furent utilisés. Encore une fois, seuls les virus à ADN ont été étudiés. Ceux-ci représentaient au plus 0,0051% des domaines identifiés. Des homologies entre des séquences retrouvées et des séquences de certains virus pathogènes humains ont pu être observées, notamment avec les rétrovirus, les papillomavirus, et les polyomavirus. Finalement, Be et al. ont aussi caractérisé en 2014 la flore virale à ADN dans l’air d’espaces urbains [83]. Des unités d’échantillonnage portables furent obtenues de la région de Washington de 2009. Ils représentent une semaine d’échantillonnage à chaque jour, pour chacune des quatre saisons. Une purification de l’ADN seulement a été exécutée sur le liquide d’élution des filtres avant de procéder aux analyses métagénomiques. Après les vertébrés, les séquences virales (incluant les séquences de phages) sont celles les plus sous-représentées dans les analyses de cette étude. Aucune identification à des virus ou des familles de virus n’a été faite. Étant donné le manque de littérature sur la matière, aucun protocole standardisé n’a pu être développé pour l’échantillonnage de l’air adapté pour des analyses de métagénomique virale.
17
Les adaptations pour les échantillons d’air
Pour ce projet, certaines modifications ont dû être apportées aux protocoles de métagénomique choisis. Étant donné que les bioaérosols ont été récoltés dans des liquides de collection, des protocoles développés pour des échantillons marins avaient tout d’abord été sélectionnés. Une fois récoltés et retournés au laboratoire, les échantillons ont subi de longues agitations mécaniques afin de désagréger les agrégats de molécules qui constituent les gros aérosols. Toutefois, aucun contrôle n’est possible afin de valider l’efficacité de cette méthode de relargage des virus à partir des particules d’aérosols. Par la suite, des étapes de filtration et d’isolement des virus selon leur petite taille et densité ont pu être effectuées dans le but d’enrichir la fraction virale et d’obtenir des résultats de séquençage plus profonds.
18
La problématique, les objectifs et les hypothèses
des travaux de recherche
La problématique
Les CTEU sont très contaminés en micro-organismes à cause des eaux qu’ils traitent. Les bactéries et les virus contenus dans ces eaux ont été bien documentés dans la littérature. Les bactéries dans l’air de ces centres a aussi été dans la mire de plusieurs études et ont prouvé la réelle exposition à ces micro-organismes. Malgré la proéminence de maladies majoritairement de causes virales chez les travailleurs et la présence de plusieurs virus pathogènes dans les eaux usées, un manque flagrant dans la littérature sur l’exposition professionnelle aux bioaérosols viraux dans les CTEU empêche d’établir des liens avec les problèmes de santé des travailleurs et, ultimement, de proposer des moyens de protection adéquats pour la santé respiratoire. Ainsi, la flore virale des CTEU doit être étudiée plus en profondeur et de manière plus exhaustive que les études existantes afin de contribuer à l’avancement des connaissances en la matière.
19
Les objectifs
Dans cette étude, l’air de quatre usines du Québec a été échantillonné dans le but d’obtenir une description approfondie de la flore virale de l’air. De plus, les usines ont été visitées à deux reprises, une fois pendant l’été et une fois pendant l’hiver, afin de caractériser l’effet de la température sur les virus aérosolisés. Les objectifs généraux de cette étude étaient donc :
• D’étudier les bioaérosols des onze virus sélectionnés à l’aide de la quantification moléculaire spécifique;
• D’évaluer le contenu génétique viral total retrouvé dans les échantillons de l’air des usines d’épuration des eaux usées grâce à la métagénomique virale;
20
Les hypothèses
Selon les connaissances actuelles, trois hypothèses ont aussi été élaborées en fonction des objectifs de recherche :
1. Les virus les plus retrouvés dans les eaux usées dans la littérature sont aussi retrouvés dans l’air des usines;
22
Chapitre 2 : Exposition professionnelle aux
bioaérosols viraux dans les usines de traitement
des eaux usées : détection moléculaire et
métagénomique
23
2.1.
Article: Human viral pathogens are pervasive in
wastewater treatment centers aerosols
Article soumis le 20 février 2017 dans le journal Journal of Environmental Sciences (numéro de manuscrit JES_2017_455)
Evelyne Briseboisa,b, Marc Veilletteb, Vanessa Dion-Duponta,b, Jacques Lavoiec, Jacques
Corbeild, Alexander Culleya and Caroline Duchainea,b,*.
aUniversité Laval, Pavillon Alexandre Vachon, 1045, ave de la Médecine, Québec, Qc,
Canada, G1V0A6
bCRIUCPQ, 2725, chemin Sainte-Foy, Québec, Qc, Canada, G1V 4G5 cIRSST, 505 Boulevard de Maisonneuve O, Montréal, QC, Canada, H3A 3C2 dCRCHU, 2705 boulevard Laurier, RC-709, Québec, Qc, Canada, G1V 4G2
Keywords: Airborne viruses; air sampling; wastewater treatment plants; viral metagenomics.
24
Résumé
Les travailleurs des centres de traitement des eaux usées (CTEU) peuvent être vulnérables aux maladies causées par les virus comme le rhume, la grippe et les infections gastro-intestinales. Bien que la communauté microbienne dans les eaux usées soit bien décrite dans la littérature, seules quelques études ont caractérisé la fraction virale des aérosols des CTEU malgré le fait que la plupart des maladies affectant les travailleurs des CTEU soient d'origine virale et que certains de ces virus soient transmis par l’air. Dans cette étude, la présence de 11 agents pathogènes viraux particulièrement préoccupants dans ce milieu a été évaluée dans quatre CTEU et une approche métagénomique a été employée pour caractériser la communauté virale totale dans l'air d'un de ces CTEU. La présence de virus dans les aérosols dans différents sites des CTEU a été évaluée et les résultats obtenus avec quatre échantillonneurs d'air couramment utilisés ont été comparés. Qquatre des onze virus testés ont été détectés, soit l'adénovirus humain (hAdV), le rotavirus, le virus de l'hépatite A (HAV) et le virus Herpès Simplex de type 1 (HSV-1). Les résultats de l'essai métagénomique ont révélé très peu de séquences d'ARN virales dans les échantillons d’aérosols, mais les séquences d'ADN provenant de virus humains présentaient une abondance relative beaucoup plus grande.
25
Abstract
Wastewater treatment center (WTCs) workers may be vulnerable to diseases caused by viruses, such as the common cold, influenza and gastro-intestinal infections. Although there is a substantial body of literature characterizing the microbial community found in wastewater, only a few studies have characterized the viral component of WTC aerosols, despite the fact that most diseases affecting WTC workers are of viral origin and that some of these viruses are transmitted through the air. In this study, we evaluated in four WTCs the presence of 11 viral pathogens of particular concern in this milieu and used a metagenomic approach to characterize the total viral community in the air of one of those WTCs. The presence of viruses in aerosols in different locations of individual WTCs was evaluated and the results obtained with four commonly used air samplers were compared. We detected four of the eleven viruses tested, including human adenovirus (hAdV), rotavirus, hepatitis A virus (HAV) and Herpes Simplex virus type 1 (HSV1). The results of the metagenomic assay uncovered very few viral RNA sequences in WTC aerosols, however sequences from human DNA viruses were in much greater relative abundance.
26
Introduction
WTCs (wastewater treatment centers) are, unsurprisingly, highly contaminated environments. Concentrations of viruses in effluent waters can be extremely high, sometimes reaching 1011 viruses/mL (La Rosa, Pourshaban et al. 2010). Several studies
have reported higher symptom and disease rates among this group of workers (Khuder, Arthur et al. 1998). They experience respiratory symptoms, fevers, gastrointestinal symptoms, and headaches more often than the non-exposed population (Khuder, Arthur et al. 1998, Smit, Spaan et al. 2005, Van Hooste, Charlier et al. 2010). The term “sewage worker’s syndrome” was used for the first time in 1973 to describe the assemblage of these symptoms (Rylander, Andersson et al. 1976).
Despite the fact that most WTC occupational symptoms can be associated with viral infections, only a few studies have investigated viruses as an occupational risk in these environments. Previous studies have demonstrated the presence of human pathogenic viruses in influent water, including noroviruses (Pouillot, Van Doren et al. 2015, Qiu, Lee et al. 2015) and rotaviruses (Baggi, Demarta et al. 2001, Qiu, Lee et al. 2015) that cause gastroenteritis; adenoviruses (Osuolale and Okoh 2015, Qiu, Lee et al. 2015), rhinoviruses and enteroviruses (Baggi, Demarta et al. 2001, Qiu, Lee et al. 2015) that are responsible for the common cold, and even herpes simplex viruses, which can cause oral and genital sores and blisters (Bibby and Peccia 2013). Among the viruses detected at WTCs, many of these pathogens are transmitted by aerosols (Tseng, Chang et al. 2010, Bonifait, Charlebois et al. 2015). Despite the importance of this route of transmission, surprisingly few studies have examined the potential exposure of WTC workers to pathogenic viruses through the air. Moreover, in these studies, only one or two viruses were analyzed (Romano, Montesi et al. 1999, Uhrbrand, Schultz et al. 2011, Masclaux, Hotz et al. 2014). In Denmark WTCs, noroviruses (Noro) GI and GII were detected for the first time in air samples using personal samplers (Uhrbrand, Schultz et al. 2011). Adenovirus (AdV), hepatitis E virus (HEV) and norovirus were also investigated in air samples from 31 Swiss WTCs. The researchers found AdV, noroviruses and HEV in 100%, 2% and 0%, respectively of the WTC aerosols tested (Masclaux, Hotz et al. 2014). Enterovirus and reovirus were identified in 3.4% of the air samples from an aerosol study of WTCs in Italy (Romano, Montesi et al. 1999).
To date, metagenomics has been used to characterize viral communities in aerosols in only three studies (Whon, Kim et al. 2012, Hall, Leblanc-Maridor et al. 2013, Be, Thissen
27
et al. 2015). In all of these studies, RNA viruses were excluded, despite the fact that most human respiratory viruses have RNA genomes such as coronavirus, influenza virus, and metapneumovirus. To the best of our knowledge, this is the first time that a metagenomic approach has been used to characterize viral communities from WTC aerosols.
In Eastern Canada, WTC secondary treatment can differ from one plant to another. For example, some centers perform biological removal of residual matter (also called biofiltration) as the water exits the early stages of the treatment pipeline, while others conduct a secondary decantation as an alternative to biofiltration. Every day, WTC employees work in rooms or locations where different processing steps are taking place, some of which increase the risk of exposure to bioaerosols. In this study, we evaluated the presence of viruses in aerosols in different locations of WTCs, determined the abundance of several human viral pathogens in WTC aerosols, and compared the performance of four commonly used air samplers.
28
Methods
WTCs and site selection
Since very few reports exist on occupational airborne viral exposure, there is no consensus sampling strategy for the collection and purification of viral nucleic acids from aerosols. We therefore compared the efficiency of low and high flow rate filtration-based sampling approaches, as well as liquid cyclonic sampling and nanometric and micrometric fractionating samplers.
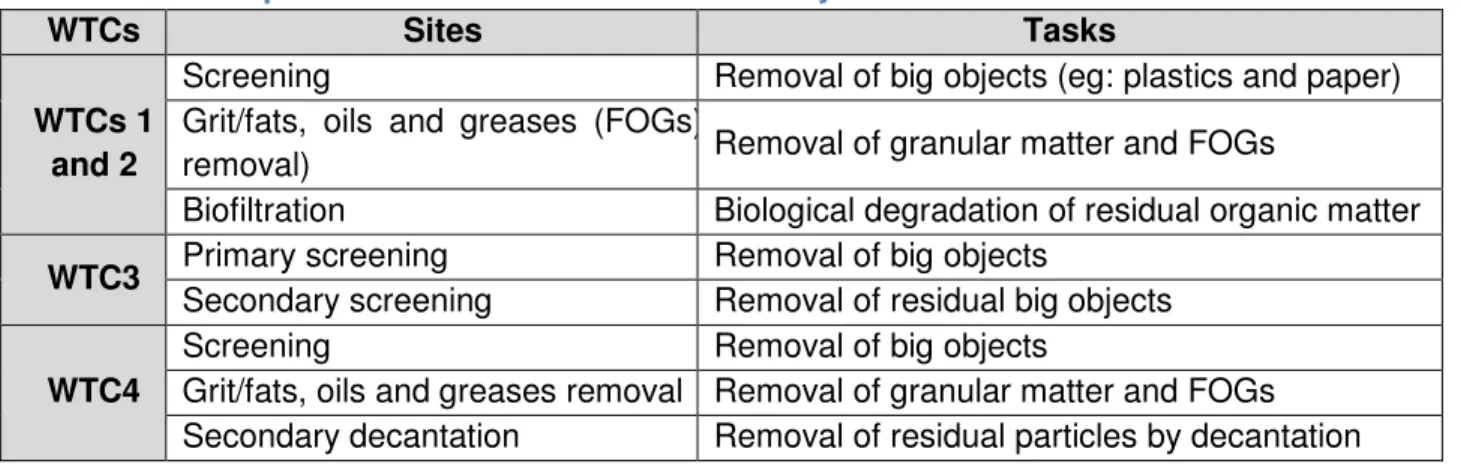
Air samples were collected from four different WTCs from Eastern Canada. As the main purpose of this study was to identify the putative etiological agents of some occupational diseases to WTC workers, only indoor sites were sampled, where the workers were confined and therefore, more exposed to bioaerosols and where daily tasks had to be performed. Based on these criteria, 11 sites (or sampling locations) distributed among four WTCs were eligible for this study. Pertinent details for each site are presented in Table 2-1.
Table 2-1. Description of WTCs and sites of the study.
WTCs Sites Tasks
WTCs 1 and 2
Screening Removal of big objects (eg: plastics and paper) Grit/fats, oils and greases (FOGs)
removal) Removal of granular matter and FOGs
Biofiltration Biological degradation of residual organic matter
WTC3 Primary screening Removal of big objects
Secondary screening Removal of residual big objects WTC4
Screening Removal of big objects
Grit/fats, oils and greases removal Removal of granular matter and FOGs Secondary decantation Removal of residual particles by decantation
Sampling methods
Four different samplers were used in this study. All four were used to collect samples for qPCR while only one of the four was used for both molecular quantification and metagenomics (SASS 2300). The samplers were positioned at the same location at each water treatment site. Although the sampling duration was dependent on the sampler type, sampling was always performed during the same day over the same 6 hour shift.
29
The Coriolis®µ (Bertin Technologies, Montigny-le-Bretonneux, FRANCE) is a liquid cyclone that collects particles in 15 mL of Phosphate Buffer Saline (PBS). The flowrate was 200 L/min for 10 min, for a total of 2 m3 of air/sample. The volume of the recipient was
readjusted to 15 mL after sampling to compensate for evaporation. Five mL of the total were used to conduct viral qPCR analyses (0.67 m3 of air).
The second sampler was a fractionating collector, the Marple Personal Cascade Impactor (Thermo Fisher Scientific, Waltham, USA). A maximum of two sampling locations per WTC were selected due to limitations on the duration of sampling at each site. In this machine, air is accelerated by going through six radial slots of a first impactor stage in which each of the 8 stages impacts a subfraction of particles ranging from 0.5 to 21 µm of aerodynamic diameters that are ultimately collected on filters. After the 8th stage, another filter collects the remaining fine particles. The flowrate of the Marple was of 2 L/min for 5 hours, for a total of 0.6 m3 filtered. Each of the 9 filters was eluted in 5 mL of PBS and 500
µl were used for qPCR.
The third sampler was a high flowrate device called the SASS® 3100 (Research International, Washington, USA). Airborne particles are collected on a charged electret filter. Air passes through the filter at a flowrate of 300 L/min for a total of 20 m3 of air filtered by
sample (sampling time of 67 min). Filters were eluted afterward in 5 mL of the elution buffer with the help of the SASS 3010 Particle Extractor.
The final sampling device, the SASS 2300 (Research International, Washington, USA), is a liquid cyclone that collects particles in 5 mL of buffer. The flowrate was 300 L/min for a total of 30 m3 of air filtered by sample (sampling time of 100 min). One half of the final
sample of WTC 4 was used for qPCR analyses and the other half for metagenomics.
All samples were kept frozen at -80°C from the time they arrived in the laboratory to when nucleic acids were extracted. Samples from the Coriolis®µ were concentrated via 100 kDa Amicon tangential flow filtration devices (Millipore, Darmstadt, GERMANY) to a final volume of 200 µl before freezing.
30
All nucleic acids were extracted with the QIAamp MinElute Spin Virus kit (Qiagen, Hilden, GERMANY) that co-purifies DNA and RNA as per instructions. Reverse transcription was performed on samples used for quantification of RNA viruses with the iScript™ cDNA Synthesis kit according to the manufacturers’ instructions.
qPCR analyses
We selected viral primers based on two criteria: 1. whether they amplified human pathogens that can cause symptoms often encountered by WTC workers and 2. whether their presence in wastewaters had been demonstrated. Eleven viruses fit these criteria: influenza viruses (Inf) A and B, noroviruses (Noro) GI and GII, herpes simplex viruses (HSV) 1 and 2, human rhinovirus, enterovirus, human adenovirus, rotavirus (rota) and hepatitis A virus.
The same thermocycler protocol was performed for all eleven viruses: an initial denaturation step of 3 min at 94°C, followed by 40 cycles of a denaturation step at 94°C for 15 sec followed by an annealing/extension step at 60°C for 1 min. All standard curves had an efficiency between 90% and 110% and an R2 above 0.98. Table 2-2 lists the sequences
31
Table 2-2. Primers and probes used for qPCR detection of selected viruses.
Viruses RNA/
DNA Forward primers Reverse primers Probes Reference
Inf A RNA GACCRATCCTGTCACCTCTGAC AGGGCATTYTGGACAAAKCGTCTA TGCAGTCCTCGCTCACTGGGCACG (Selvaraju and
Selvarangan 2010)
Inf B RNA TCCTCAACTCACTCTTCGAGCG CGGTGCTCTTGACCAAATTGG CCAATTCGAGCAGCTGAAACTGCGGTG (Selvaraju and
Selvarangan 2010)
Noro GI RNA CGYTGGATGCGNTTYCATGA CTTAGACGCCATCATCATTYAC AGATYGCGATCYCCTGTCCA (Kageyama, Kojima et
al. 2003)
Noro GII RNA CARGARBCNATGTTYAGRTGGATGAG TCGACGCCATCTTCATTCACA TGGGAGGGCGATCGCAATCT (Kageyama, Kojima et
al. 2003)
HSV-1 DNA CGCATCAAGACCACCTCCTC GCTCGCACCACGCGA TGGCAACGCGGCCCAAC (Corey, Huang et al.
2005)
HSV-2 DNA CGCATCAAGACCACCTCCTC GCTCGCACCACGCGA CGGCGATGCGCCCCAG (Corey, Huang et al.
2005) HRV RNA GTGAAGAGCCSCRTGTGCT GCTSCAGGGTTAAGGTTAGCC TGAGTCCTCCGGCCCCTGAATG (Hayden, Herrington et
al. 2003) EV RNA GGCCCCTGAATGCGGCTAAT CAATTGTCACCATAAGCAGCCA CGGACACCCAAAGTAGTCGGTTCCG
(Donaldson, Griffin et al. 2002, Meijer, van der
Sanden et al. 2012)
hAdV DNA GCCACGGTGGGGTTTCTAAACTT GCCCCAGTGGTCTTACATGCACAT TGCACCAGACCCGGGCTCAGGTACTCCGA (Heim, Ebnet et al.
2003)
Rotavirus RNA ACCATCTWCACRTRACCCCTCTATGAG GGTCACATAACGCCCCTATAGC AGTTAAAAGCTAACACTGTCAAA (Zeng, Halkosalo et al.
2008) HAV RNA GGTAGGCTACGGGTGAAAC CCTCCGGCGTTGAATGGTTT ACAGCGGCGGATATTGGTGAGTTGTTAAGA (Qiu, Cao et al. 2014)
W= A/T, S= C/G, K= G/T, R= A/G, Y= C/T and N= A, T, C or G.
Metagenomics study
Samples intended for metagenomics analyses were thawed from the -80°C freezer and concentrated in Amicon 100 kDa tangential flow filtration devices (Millipore) to a final volume of 200 µl. Nucleic acids were subsequently extracted with the QIAamp MinElute Spin Virus kit (Qiagen, Hilden, GERMANY) as per instructions. Concentrated nucleic acids were eluted in a final volume of 87.5 µl to directly undergo the digestion of DNA with the RNase-Free DNase Set from Qiagen, followed by RNA purification with the RNeasy MinElute Cleanup kit from Qiagen. RNA samples were processed for library construction with the Kapa Stranded mRNA-Seq Kit (Kapa Biosystems, San Diego, USA) and then sequenced on the MiSeq sequencing platform (Illumina, Inc., paired-end 300 nucleotides reads) at the sequencing facility of the Centre hospitalier de l’Université Laval (CHUL). The reads were assembled into contigs by Ray Meta (Boisvert, Raymond et al. 2012). The Ray Meta software uses a bloom filter to remove k-mers that are potentially background noise and sequencing errors. Table 2-6 provides details for the sequencing and assembly output. Data were subsequently analyzed via the web-based MG-RAST (Meyer, Paarmann et al. 2008) and MetaVir platforms (Roux, Faubladier et al. 2011).
32
Results
qPCR
Four viruses were detected above quantification limits; HSV1, HAV, hAdV and rotavirus. Table 2-3 shows where they were detected and at what concentration.
Table 2-3. Positive viral amplifications (in gene copies/m3 of air) from air samples of
Coriolis, SASS 2300 and SASS 3100 in WTCs.
WTCs Sites Coriolis SASS 2300 SASS 3100
WTC1
Site 1 Rota (4.7 x 104) hAdV, HSV 1 hAdV, HSV 1
Site 2 Rota (1.9 x 105) hAdV, HSV 1 hAdV, HSV 1
Site 3 Rota (4.5 x 10 4), HAV (4.7 x 103) hAdV (4.9 x 104), HSV 1 hAdV (5.2 x 103), HSV 1 WTC2
Site 1 Rota (2.8 x 104) hAdV, HSV 1 hAdV
Site 2 Rota (1.1 x 105) hAdV, HSV 1 hAdV
Site 3 Rota (9.8 x 104) hAdV (5.2 x 10 4),
HSV 1 hAdV (3.7 x 10
4)
WTC3 Site 1 - hAdV -
Site 2 - hAdV hAdV (2.5 x 104)
WTC4 Site 1 - hAdV -
Site 2 - hAdV hAdV
No concentration in parenthesis means detection below assay quantification limit.
Table 2-4. Assay quantification limits.
Detection limits (copies/m3) ADV HAV HSV1 Rotavirus
Coriolis 7.5 x 102 8.0 x 102 7.5 x 102 9.2 x 102
Marple - - - 7.5 x 102
SASS 2300 4.2 x 102 - 4.2 x 102 -
33
Table 2-5. Detection and quantification of rotaviruses with the Marple sampler.
WTC 1 WTC 2 WTC 3 WTC 4
Cut-Off
(µm) Stages Site 2 Site 3 Site 2 Site 3 Site 1 Site 2 Site 2 Site 3
21.3 1 - 9.6 x 104 8.2 x 104 2.9 x 105 - - - - 14.8 2 5.0 x 104 - - 9.6 x 104 - 2.6 x 104 - - 9.8 3 2.4 x 104 1.6 x 104 9.0 x 104 1.4 x 106 - 3.6 x 104 1.2 x 105 - 6 4 6.4 x 104 - 8.7 x 104 6.3 x 104 - 1.4 x 104 - - 3.5 5 1.5 x 105 3.9 x 104 7.1 x 104 1.6 x 105 - 5.0 x 104 2.3 x 104 - 1.55 6 - 1.4 x 104 - 5.2 x 104 - 8.3 x 105 - - 0.93 7 - 1.9 x 104 7.2 x 104 7.6 x 105 - 5.5 x 104 - 2.4 x 104 0.52 8 - - - - - 4.1 x 104 1.1 x 105 - 0 F - 2.0 x 104 - 1.2 x 105 - 4.2 x 104 - 3.4 x 104 Metagenomics
Viral metagenomes were produced from three sites from WTC 4. The dataset for the three locations were as shown in Table 2-6.
Table 2-6. Sequencing and assembly output details.
Sites Number of reads Number of contigs Total size of assembly Average contig length N50 of assembly Median contig length Largest contig Site 1 18741055 45130 101595404 2251 8800 772 1635750 Site 2 764755 602 536721 891 811 588 15090 Site 3 604133 268 240403 897 849 622 8222
34
Figure 2-1. Air samples from WTC 4 (A) Classification of assigned sequences at a domain taxonomic level, (B) Classification of virus-assigned sequences by Family, (C) Hosts of viruses based on the Figure 2-1B assignments.
In Figure 2-1A, the classification of assigned sequences is at a domain taxonomic level. In Figure 2-1B, the classification of virus-assigned sequences (n=9,996) was divided by Family: Families with less than 1% representation were classified in the group “other”. Figure 2-1C represents the hosts of viruses based on the Figure 2-1B assignments. Bacteria represented the most abundant organisms in all three samples with an average of almost 93% of all sequences. Among viral sequences, more than 62% were from phages in the order Caudovirales. 1.2% of the viruses’ hosts were human (see Figure 2-1C). The
92,88% 5,32% 1,49% 0,16% 0,11% 0,04% A. Domains distribution Bacteria Eukaroyta Unassigned Viruses Archaea Unclassified 29,5% 26,2% 19,9% 16,4% 4,5% 3,5% B. Virus families Baculoviridae Siphoviridae Podoviridae Myoviridae Unclassified Other Phages 67,15% 1,19% 30,03% 0,06% 0,47% 0,28% 0,18% 0,63% 0,01% C. Hosts of viruses Bacteria Humans Insects Fishes Mammals Amoeba Plants Algae Birds
35
classification of these sequences to the lowest taxonomic level possible is shown in Figure 2-2. Table 2-7 (in appendix) shows the taxonomy of the viruses in the group “other” to the family level.
Figure 2-2. Distribution of human viruses in samples from sampling locations 1 (A), 2 (B) and 3 (C) from WTC 4.
Human endogenous retroviruses were recovered from all three samples. Sequences from HSV1 and HSV5, two types of herpes simplex viruses causing latent infections, were also found in all three sites. Chlamydiamicrovirus sequences were identified in both site 2 and site 3 samples, and were the most highly represented human virus in site 3. Human papillomavirus, a virus that causes latent sexually transmissible infections, was the most
8,9% 8,9% 7,1% 3,6% 71,4% A. Site 1 HSV1 HSV5 HAdV HIV Human endogenous retrovirus 3,4% 3,4% 53,9% 14,6% 24,7% B. Site 2 HSV1 HSV5 Human papillomavirus Chlamydiamicrovirus Human endogenous retrovirus 12,1% 12,1% 36,4% 6,1% 33,3% C. Site 3 HSV1 HSV5 Chlamydiamicrovirus Variola virus Human endogenous retrovirus
36
common human virus detected in site 2. Variola virus sequences were found to be in the air sample from site 3 only.
Figure 2-3. Heat map comparing metagenomic results from site 1, 2 and 3 of WTC 4 with samples from wastewater and source water in North America (public databases). The red scale represents low abundance cases, as the green one represents high abundance cases.
37
As this was the first time that viral metagenomics had been applied to air samples from WTCs, we supposed that viruses, and also all organisms found in the air, would originate from wastewaters. To verify this hypothesis, a heat map was generated with MetaVir featuring our results and data from a public database of samples of wastewaters and source waters in North America.
In Figure 2-3, samples are clustered based on similarity. For example, site 1 sequences are more similar to sequences from site 2 than the sequences from site 3, while the sequences from site 3 are more similar to wastewater sequences than to those from the source water sample.
Discussion and conclusion
Different viruses were detected with all four sampling devices which suggest that the device is a source of bias. For example, rotavirus was detectable only with the Marple and the Coriolis, and not with the SASS high flow rate samplers. Rotavirus is a double-stranded RNA virus. As RNA is extremely fragile and rapidly degraded by RNases found ubiquitously in nature, there is a chance that samplers with a flow rate higher than 200 L/min remove less robust RNA virus species during collection (Cao, Noti et al. 2011, Toulouse M.-J. 2014). This may also explain why only DNA viruses were detected with the SASS 2300 and the SASS 3100 samplers. The Marple and the Coriolis are based on two completely different capture mechanisms; filtration and liquid impaction. Both of these mechanisms may induce particle loss (bouncing and re-aerosolization). The different sizes and characteristics of the eleven selected viruses likely also plays a role in the capture efficiency of each sampler, as some enveloped or non-enveloped viruses are more or less resistant to desiccation caused by the sampling times and rates.
Overall, rotaviruses seem to be abundant in the air of WTCs based on qPCR detection. The sites that showed the highest concentrations of rotaviruses were also the sights that had the highest concentration of total viruses (e.g. biofiltration sites of WTCs 1 and 2). Inhalation of pathogenic viruses causing respiratory infections has been studied over the years since it represents a preferential route of transmission for some viruses. Rotavirus is known to cause gastroenteritis. The infectious dose of this virus through the oral route is below 100 viral