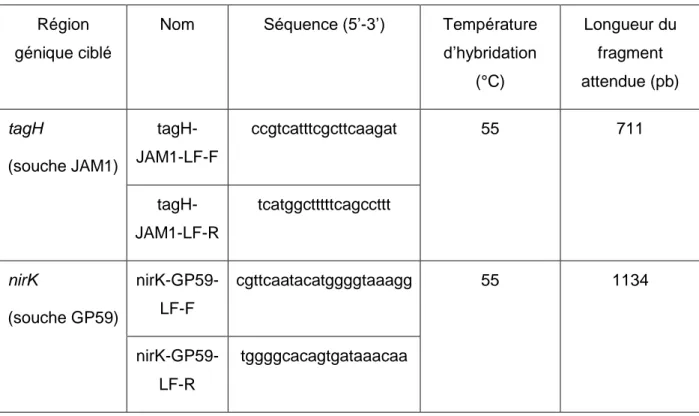
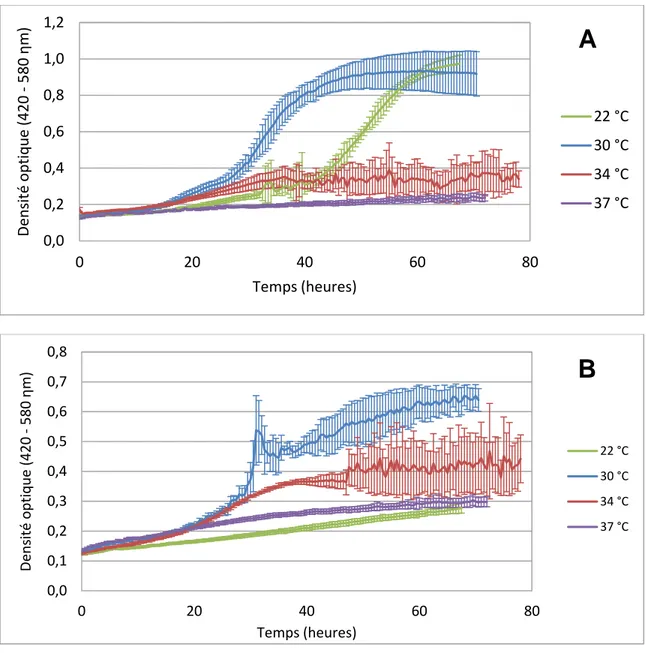
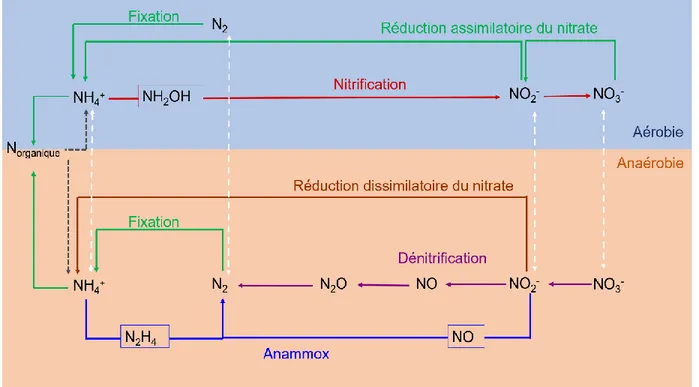
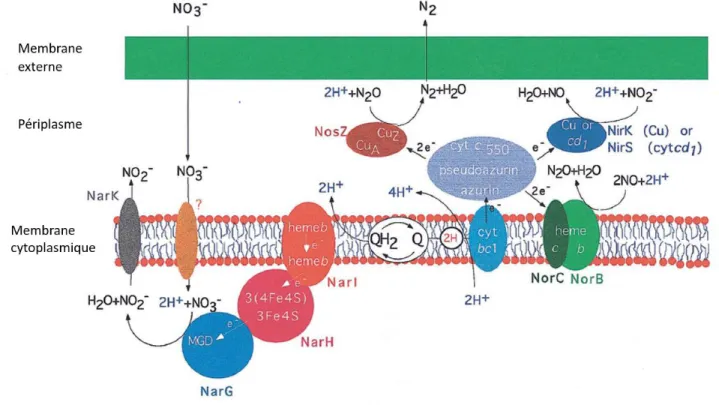
Caractérisation de la souche methylophaga SP. GP59, une nouvelle bactérie dénitrifiante
Texte intégral
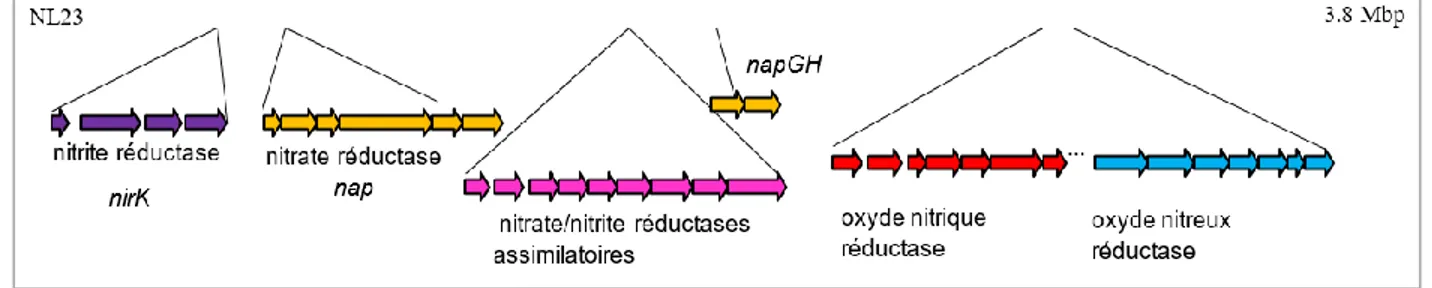
Figure

Outline
Documents relatifs
Par séquençage Illumina ® et grâce au site d’analyse PubMLST nous avons réalisé l’analyse génomique de six souches appartenant au clone B:14:P1.7,16 ST32 (quatre isolées
Aucun des 32 animaux testés n'est mort à la suite d'une épreuve hétérologue (Tableau 1). Les expériences d'immunité croisée entre la souche Gardel d'une part et
L'examen de ce matériel coloré au carmin chlorhydrique et monté au baume du Canada, nous permet de confirmer un certrnn nombre de caractères morphologiques propres
On leur demande les bons souvenirs qu’ils avaient de la personne qui est morte et les mauvais souvenirs aussi qu’ils ont de cette personne pour les aider à intégrer que,
Dans ces conditions, on peut inverser les priorités auctoriales et comprendre la préface des Cahiers comme un récit premier, sur lequel les aventures de Le Golif se
Comparing the relative intensities of the [M-NCO – -PO 3 – - H 2 O] signals of the different investigated ONs, it is apparent that the charge level dependence of cyanate loss
Marine Le Goas, Aurélie Paquirissamy, Béatrice Cambien, Thierry Pourcher, Geraldine Carrot, Jean- Philippe Renault, Serge Palacin.. Nanotechnologies are being widely studied
Par rapport aux patients avec évolution délétère, les patients présentant une évolution favorable ont, à leur admission, le même nombre total de cellules progénitrices mais