THESE
THESE
En vue de l'obtention du
DOCTORAT DE L’UNIVERSITÉ DE TOULOUSE
DOCTORAT DE L’UNIVERSITÉ DE TOULOUSE
Délivré par l'Université Toulouse III - Paul Sabatier Discipline ou spécialité : Biologie moléculaire
JURY
Mr. Pierre-Emmanuel Gleizes, Professeur d'université, Toulouse - Président Dr. Christine Allmang-Cura, Chargée de recherche CNRS, Strasbourg - Rapporteur
Dr. Hervé Le Hir, Chargé de recherche CNRS, Gif-sur-Yvette - Rapporteur Dr. Rémi Bordonné, Directeur de recherche CNRS, Montpellier - Examinateur
Dr. Philipp Milkereit, PI, Regensburg (Allemagne) - Examinateur Dr. Yves Henry, Directeur de recherche CNRS, Toulouse- Directeur de Thèse
Ecole doctorale : Biologie-Santé-Biotechnologie de Toulouse Unité de recherche : Laboratoire de Biologie Moléculaire Eucaryote
Directeur(s) de Thèse : M. Yves HENRY Rapporteurs : Dr Allmang-Cura, Dr Le Hir
Présentée et soutenue par Simon LEBARON Le 1er Octobre 2008
À M. Tirrolloy Philippe, Mon grand-père, disparu trop tôt .
Il m’a appris à conjuguer observation et
raisonnement afin de mieux comprendre
le monde dans lequel je vis …
1
2
Table des Matières... 1
Introduction ... 6
I. Les Ribosomes... 7
A. Composition des ribosomes ...7
B. Principe de la traduction par les ribosomes ...8
1. L’initiation de la traduction ... 8
2. L’élongation de la traduction ... 9
3. La terminaison de la traduction... 9
C. Structure des ribosomes ...10
1. Structure de la petite sous-unité procaryote... 10
2. Structure de la grande sous-unité procaryote... 11
3. Structure du ribosome 70S... 11
4. Structure du ribosome 80S des eucaryotes ... 12
D. Bases moléculaires des activités du ribosome...13
1. Le décodage de l’information génétique... 13
2. La synthèse de la liaison peptidique ... 13
II. La biogenèse des ribosomes ... 16
A. Présentation du processus ...16
B. Organisation de l’ADN ribosomique chez Saccharomyces cerevisiae...17
C. Le Nucléole ...18
1. Structure du nucléole ... 18
2. Dynamique structurale du nucléole ... 19
3. Composition du nucléole ... 20
4. Le nucléole : un compartiment multifonctionnel... 21
D. La transcription de l’ARN ribosomique 5S...23
E. La transcription par l’ARN polymérase I ...24
1. Composition de l’ARN polymérase I ... 25
2. Formation du complexe d’initiation... 25
3. Elongation de la transcription Pol I dépendante ... 26
4. Terminaison de la transcription Pol I... 27
5. La transcription Pol I : un processus hautement régulé ... 27
3
F. Les événements post-transcriptionnels de la biogenèse des ribosomes ...30
III. Les Hélicases... 32
A. Classification des hélicases ...32
1. Superfamille I (SFI) ... 33
2. Superfamille II (SFII) ... 33
B. Les motifs conservés...34
1. Motif Q (G-F-X-X-P-X-P-I-Q):... 34
2. Motif I (A/G-X-X-G-X-G-K-T) ... 35
3. Motif Ia (P-T-R-E-L-A) et Ib (TPGR)... 35
4. Motif II (X-X-D-E-X-X)... 35
5. Motif III (S-A-T)... 36
6. Motif IV ... 36
7. Motif V... 36
8. Motif VI (X-R-X-G-R-X-G-R)... 37
C. Structure tertiaire des hélicases à ARN de la famille SFI et SFII ...37
D. Mode d’action des hélicases à ARN de la famille SFI et SFII...40
1. Mécanisme « Stepping » ... 41
2. Mécanisme du mouvement Brownien ... 41
E. Spécificité de reconnaissance du substrat des hélicases à ARN. ...43
F. Régulation de l’activité des hélicases par des partenaires protéiques ...44
G. Modifications post-traductionnelles des hélicases ...46
IV. Fonctions des Hélicases à ARN... 47
A. Fonctions moléculaires des hélicases à ARN ...47
1. Les hélicase à ARN permettent la séparation de duplexes d’ARN ... 47
2. Le réarrangement de particules ribonuléoprotéiques (RNP) par les hélicases à ARN ... 47
3. Les hélicases à ARN peuvent perturber des associations protéines/protéines... 50
4. Les hélicases à ARN aident à la formation de duplexes d’ARN ... 50
B. Processus biologiques impliquant des hélicases à ARN...51
1. Les hélicases à ARN et la transcription ... 51
2. Les hélicases à ARN et l’épissage des ARNm ... 52
3. Les hélicases à ARN et l’export nucléaire ... 53
4
5. Les hélicases à ARN dans la biogenèse des ribosomes ... 54
6. Les hélicases à ARN et la dégradation de l’ARN... 56
V. Prp43p une hélicase à ARN multifonctionnelle ... 58
A. Introduction...58
B. Description de la protéine Prp43p ...58
C. Données préliminaires suggérant un rôle de Prp43p dans la biogenèse des ribosomes ....59
Résultats... 60
I. Prp43p une hélicase à ARN, impliquée dans l’épissage des pré-ARNm agît aussi dans la biogenèse des ribosomes... 61
A. Discussion ...63
II. Prp43p une hélicase à ARN qui doit être régulée ... 64
A. Introduction ...64
B. Recherche des partenaires directs de Prp43p...64
C. Présentation des partenaires à domaine G-patch de Prp43p ...65
1. Pfa1p/Sqs1p ... 65
2. Gno1p/Pxr1p ... 65
3. Ntr1p/Spp382p ... 66
D. Étude de l’implication des partenaires à G-patch de Prp43p dans la biogenèse des ribosomes ...67
1. Rôle de Pfa1p dans la biogenèse des ribosomes ... 67
2. Rôle de Gno1p dans la biogenèse des ribosomes. ... 68
3. Rôle de Ntr1p dans la biogenèse des ribosomes. ... 68
E. Gno1p et Pfa1p sont des partenaires directs de Prp43p...70
1. Pfa1p est capable de se lier directement à Prp43p in vitro ... 70
2. Gno1p est capable de se lier directement à Prp43p in vitro... 70
3. Gno1p et Pfa1p n’interagissent pas ensemble in vitro... 71
F. Gno1p et Pfa1p ne sont pas requises pour l’association de Prp43p avec les particules pré-ribosomiques ...71
1. Pfa1p n’est pas nécessaire à l’association de Prp43p avec les particules pré-ribosomiques ... 72
2. Gno1p n’est pas requise pour l’association de Prp43p avec les particules pré-ribosomiques... 73
G. Il existe un lien entre Gno1p et Pfa1p in vivo ...74
1. L’absence de Pfa1p n’a pas d’effets sur Gno1p in vivo... 74
5
H. Gno1p et Pfa1p régulent les activités enzymatiques de Prp43p in vitro ...76
1. Gno1p et Pfa1p sont capables de moduler l’activité ATPase de Prp43p in vitro ... 76
2. Seule Pfa1p stimule l’activité hélicase de Prp43p in vitro ... 78
I. Discussion ...79
J. Perspectives ...81
Conclusion Générale ... 83
I. Etude sur la fonction moléculaire des facteurs intervenant dans la biogenèse des ribosomes. ... 84
II. La biogenèse des ribosomes est intégrée aux autres processus cellulaires... 85
III. Les hélicases à ARN sont des enzymes qui nécessitent d’être régulées... 86
Matériel et Méthodes ... 88
Annexe 1 ... 95
La caractérisation de Npa2p révèle l’existence d’un complexe, formé en association avec les protéines Dbp6p, Npa1p, Nop8 et Rsa3p, impliqué dans les étapes précoces de maturation des particules ribosomiques 60S chez S. cerevisiae ... 95
6
Tableau 1 : Caractéristiques des sous-unités ribosomiques
Figure 1 : Représentation d’un ribosome en cours de traduction
Coefficientsédimentation Taille Nombre d’ARN
Nombre de protéines Grande sous unité 50S 1.5 MDa
2 L’ARNr 23S (2904 nt) L’ARNr 5S (122nt) 34 Ribosome procaryote : Escherichia Coli
Petite sous unité 30S 0.8 MDa 1
L’ARNr 16S (1542 nt) 21
Grande sous unité 60S 2.9 MDa
3 L’ARNr 25S (3392 nt) L’ARNr 5S (121 nt) L’ARNr 5.8S (158 nt) 46 Ribosome eucaryote : Saccharomyces cerevisae
Petite sous unité 40S 1.4 MDa 1
7
I. Les Ribosomes
Le ribosome est la particule RiboNucléoProtéique (RNP) cellulaire la plus complexe qu’il soit. De plus, c’est un élément clé de l’expression génique. En effet, l’information génique est conservée sous la forme d’acide désoxyribonucléique (ADN). Pour que l’information soit exploitable par la cellule, il est nécessaire que l’ADN soit transcrit en acide ribonucléique (ARN). À l’issue de ce processus, deux possibilités existent : Soit les ARN sont directement exploitables par la cellule (ARN dits non codants). Soit les ARN sont dits messagers (ARNm) : ils doivent être traduits en protéines par les ribosomes. Dans ce processus, les ribosomes assument une double fonction de décodage des ARNm et de synthèse de polypeptides.
A. Composition des ribosomes
Les ribosomes sont des RNP résultant de l’association de molécules d’ARN ribosomiques (ARNr) avec des protéines ribosomiques. L’ARNr est un constituant important dans ces complexes car il représente 60% de la masse totale des ribosomes. Bien qu’il existe des différences dans la composition en ARNr et en protéines, les ribosomes procaryotes et eucaryotes sont structurellement bien conservés.
Les ribosomes sont organisés en deux sous-unités de tailles différentes. On désigne couramment la taille des sous-unités et de leurs molécules d’ARN en unité Svedberg (S) qui correspond à la vitesse de sédimentation des particules en suspension. On parle ainsi de petite sous-unité de 40S et de grande sous-unité de 60S chez les eucaryotes (30S et 50S chez les procaryotes et les archae). L’association entre ces deux éléments forme la particule ribosomique mature de 80S (70S chez les procaryotes et les archaebactéries) (figure 1).
La petite sous-unité ribosomique est responsable de l’activité de décodage de l’information génétique contenue dans les ARN messagers. Elle permet l’établissement des interactions entre les codons de l’ARNm et les anticodons des ARN de transfert (ARNt). Elle est donc directement impliquée dans la fidélité de la traduction. La petite sous-unité ribosomique est constituée d’une molécule d’ARNr 18S et de 32 protéines (Planta and Mager, 1998) chez la levure S. cerevisiae (ARNr 16S et 21 protéines chez E. coli) (tableau 1).
La grande sous-unité ribosomique contient le centre peptidyl transférase (PTC) qui catalyse la formation des liaisons peptidiques entre les acides-aminés incorporés. Cette activité est donc nécessaire à l’élongation de la chaîne peptidique. La grande sous-unité est
8
composée des ARNr 25/28S, 5.8S et 5S ainsi que de 46 protéines chez les eucaryotes et des ARNr 23S et 5S associés à 34 protéines chez les procaryotes (tableau 1).
B. Principe de la traduction par les ribosomes
Les deux sous-unités ribosomiques contiennent, à leur interface, trois sites de liaison aux ARNt. Ces différents sites sont :
Le site A (Aminoacyl) dans lequel se positionne l’ARNt amino-acylé qui entre dans le ribosome.
Le site P (Peptidyl) dans lequel se trouve l’ARNt portant la chaîne polypeptidique en cours de synthèse.
Le site E (Exit) dans lequel se trouve l’ARNt déacylé juste avant qu’il ne se dissocie de l’ARNm et du ribosome.
Les mécanismes détaillés du décodage et de synthèse des liaisons peptidiques seront précisés plus loin grâce aux résultats obtenus par les analyses de structure. La traduction est une réaction qui se déroule en trois étapes : l’initiation, l’élongation et la terminaison (pour revue voir : (Ramakrishnan, 2002; Steitz, 2008)).
1. L’initiation de la traduction
L’initiation de la traduction débute par la mise en présence de trois éléments : la petite sous-unité ribosomique, l’ARNm et l’ARNt associé à la methionine. Cette étape nécessite des facteurs d’initiation de la traduction : IF chez les procaryotes et eIF chez les eucaryotes. L’association entre la petite sous-unité et l’ARNm, ainsi que la reconnaissance du codon initiateur diffèrent entre les procaryotes et les eucaryotes :
Chez les Procaryotes, l’association entre la petite sous-unité et l’ARNm se fait directement via un appariement entre l’ARNr 16S et l’ARNm. Pour cela l’ARNr 16S porte une séquence complémentaire à une séquence nommée Shine-Dalgarno présente sur l’ARNm, à environ 10 nucléotides (nt) en amont du codon initiateur. La petite sous-unité est alors correctement positionnée sur le codon AUG.
Chez les Eucaryotes, l’initiation de la traduction débute par l’association de la petite sous-unité au niveau de la coiffe 5’ de l’ARNm. Cette association nécessite l’intervention de facteurs eIF mais aussi de la queue polyA et de ses facteurs associés. La petite sous-unité va
9
ensuite scanner l’ARNm en se déplaçant dans le sens 5’- 3’ à la recherche du codon initiateur AUG situé dans le bon contexte, c’est-à-dire au sein d’une séquence nommée « Kozak ».
L’ARNt portant la méthionine possède une conformation qui lui permet d’être logé immédiatement au sein du site P, en vis-à-vis du codon AUG, formant ainsi le complexe ternaire dit « d’amorçage ». La grande sous-unité peut alors s’associer avec ce complexe, ce qui correspond à la fin de l’étape d’initiation.
2. L’élongation de la traduction
Le processus d’élongation, à la différence de l’initiation de la traduction à été conservé au cours de l’évolution et ne diffère que très peu entre les procaryotes et les eucaryotes. Cette étape est composée de plusieurs cycles permettant l’assemblage des acides aminés un à un, chaque cycle comportant trois étapes qui se répètent tout au long du processus d’élongation.
La première étape de ce cycle est le postionnement d’un nouvel ARNt amynoacylé au site A. Cette ARNt est apporté par le facteur d’élongation EF-Tu lié à une molécule de GTP. Différentes étapes, mettant en jeu un appariement entre le codon de l’ARNm et l’anticodon de l’ARNt ainsi que différents changements de conformation du centre de décodage de la petite sous-unité, assurent le contrôle de la fidélité de la traduction (Ogle et al., 2001). Si l’ARNt entrant dans le site A est correct, alors un changement de conformation du facteur EF-Tu intervient, suite à l’hydrolyse du GTP en GDP, ce qui induit une baisse de son affinité pour l’ARNt aminacylé et son détachement du ribosome.
La deuxième étape consiste en la formation de la liaison peptidique, entre la chaine polypeptidique naissante (au site P) et l’acide aminé porté par l’ARNt (au site A), par le PTC. Un modèle moléculaire de cette réaction sera présenté ultérieurement.
La troisième étape nécessite l’intervention d’un autre facteur d’élongation, EF-G qui s’associe au ribosome. L’hydrolyse du GTP lié à ce facteur induit la translocation du ribosome sur l’ARNm. Ce décalage d’un codon exactement (3 nt) libère le site A et positionne l’ARNt portant la chaîne polypeptidique en P et l’ARNt déacylé dans le site E. Suite à l’hydrolyse de sa molécule de GTP, EF-G se dissocie du ribosome et un nouveau cycle d’élongation peut commencer.
3. La terminaison de la traduction
La terminaison de la traduction intervient lorsqu’un codon STOP (UAA, UAG ou UGA) au sein de l’ARNm se positionne au niveau du site A du ribosome suite à la
Elongation de la traduction
Terminaison de la traduction
Figure 2 : Les étapes d’élongation et de terminaison de la traduction
L’étape d’initiation n’est pas représentée ici. Adaptée de Steitz., 2008
10
translocation de celui-ci. Il n’y a pas d’ARNt ayant un anti-codon complémentaire à ces codons. Les facteurs RF1/2 se positionnent alors dans le site A et induisent l’hydrolyse par le ribosome de la liaison ester unissant la chaîne polypeptidique à l’ARNt dans le site P. Ceci permet la libération du polypeptide. Ensuite, la GTPase RF3 interagit avec les facteurs RF1/2 et suite à l’hydrolyse d’une molécule de GTP, ces trois facteurs sont éjectés du ribosome.
Les étapes d’élongation et de terminaison de la traduction des ARNm sont résumées
(figure 2).
A la fin de ce processus, une étape de recyclage intervient. Elle permet la dissociation des complexes ribosome/ARNt/ARNm et l’utilisation des sous-unités ribosomiques pour un nouveau cycle de traduction. Ce processus de recyclage nécessite l’intervention de facteurs RRF (ribosome release factor) assistés par les facteur IF3 et EF-G.
C. Structure des ribosomes
La compréhension des mécanismes moléculaires de la traduction a beaucoup avancé grâce à la détermination de la structure des ribosomes. La première observation des ribosomes par microscopie électronique a été réalisée par George Palade en 1955 (Palade, 1955). Depuis de nombreuses autres observations de la structure des ribosomes ont été effectuées avec différentes techniques telles que la cryomicroscopie électronique et l’analyse de structures cristallographiques par diffraction de rayons X. Ces techniques ont été dans un premier temps appliquées à des sous-unités ribosomiques isolées : structures de la petite sous-unité (Schluenzen et al., 2000; Wimberly et al., 2000) et structures de la grande sous-unité (Ban et al., 1998; Ban et al., 1999; Ban et al., 2000; Harms et al., 2001). Dans un deuxième temps, ces techniques ont permis d’étudier des ribosomes complets associés aux ARNt et à une molécule d’ARNm. Les ribosomes procaryotes ont fait l’objet de la majorité de ces études (Korostelev et al., 2006; Schuwirth et al., 2005; Selmer et al., 2006; Yusupov et al., 2001).
Par convention, lors de la description des structures tertiaires de la grande ou de la petite unité la face dite « avant » correspond à la face en interaction avec l’autre sous-unité.
1. Structure de la petite sous-unité procaryote
La petite sous-unité ribosomique des bactéries est composée de l’ARNr 16S et d’une vingtaine de protéines. L’ARNr de cette sous-unité comme tous les ARNr présente une
Figure 3 : Structure de la petite sous-unité procaryote
A- Structure secondaire de l’ARNr 16S B- Modélisation 3D de l’ARNr 23S C- Structure
cristallographique de la petite sous-unité (l’ARNr est en gris et les protéines associées en violet).
Adaptée de Wimberly et al., 2000.
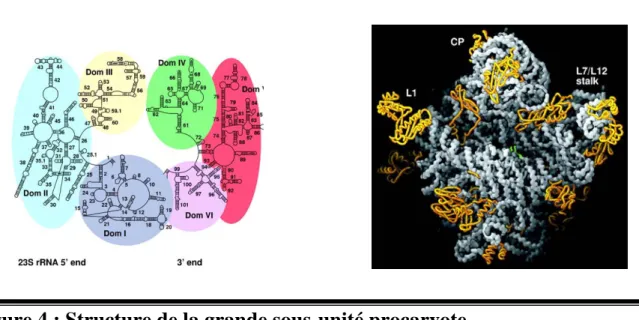
Figure 4 : Structure de la grande sous-unité procaryote
Sont représentées la structure secondaire du 23S et la structure cristallographie de la particule 50S.
11
structure secondaire complexe (figure 3). La structure cristallographique de la petite sous-unité a été résolue par plusieurs équipes (Schluenzen et al., 2000; Wimberly et al., 2000). La première des observations est que la modélisation de la structure de l’ARN 16S correspond à la structure tridimensionnelle de la petite sous-unité composée de trois domaines (figure 3). C’est l’ARNr 16S qui donne la forme globale de la sous-unité alors que les protéines semblent servir à consolider cette structure. En effet, ces protéines ribosomiques sont associées fortement à l’ARNr par l’intermédiaire de plusieurs zones d’interaction. De plus, ces protéines interagissent entre elles ce qui rigidifie la structure. La région impliquée dans le contact avec l’ARNm ainsi qu’avec la boucle de anticodon des ARNt situés dans le site A et le site P est dépourvue de protéine. Il semble donc que seul l’ARNr 16S assure le décodage de l’information et la fidélité du processus de traduction.
2. Structure de la grande sous-unité procaryote
La grande sous-unité est composée de deux ARNr, le 23S et le 5S, associés à plus d’une trentaine de protéines. La structure cristallographique de cette sous-unité révèle une organisation monolithique à laquelle s’ajoute deux protubérances latérales portant les protéines ribosomiques L1 et L7/L12 (Ban et al., 2000; Harms et al., 2001) (figure 4). Cette structure est constituée d’un unique domaine, au lieu de trois pour la petite sous-unité, ce qui indique que la petite sous-unité a besoin d’une plus grande flexibilité. L’ARNr 5S et 23S interagissent indirectement par l’intermédiaire des protéines ribosomiques. Comme pour la petite sous-unité, ce sont les composants ARN qui donnent la forme générale de la particule et les protéines maintiennent cette conformation.
3. Structure du ribosome 70S
Plusieurs études ont permis de caractériser la structure d’un ribosome complet parfois associé à des ARNm et à des ARNt (Beer et al., 1960; Korostelev et al., 2006; Schuwirth et al., 2005; Selmer et al., 2006; Yusupov et al., 2001). Ces structures ont permis d’étudier les interactions existant entre les deux sous-unités ainsi que la conformation et la position des ARNt et ARNm au sein du ribosome.
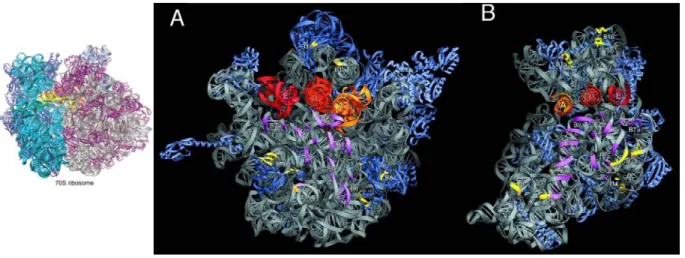
Le groupe de Noller en 2001 a résolu une structure cristallographique d’un ribosome et en a déduit les structures de la grande et de la petite sous-unité (Yusupov et al., 2001)
(figure 5). L’observation des zones de contact de ces deux structures permet de mieux
Figure 5 : Représentation de la structure du ribosome 70S et de ses
sous-unités
Les interactions entre les deux sous-unités sont indiquées :ARN-ARN en violet, protéine/protéine et protéine/ARN en jaune. Les ARNt sont repérés au site A, P et E.
Adaptée de Yusupov et al., 2001 et de Korostelev et al., 2006
12
ARNt et en particulier l’ARNt situé dans le site P constituent une large zone d’interaction. À cela s’ajoutent des interactions entre les composants même des ribosomes. Certaines protéines interagissent entre elles et les ARNr constituent la plus grande surface d’interaction (figure
5). On peut noter que les zones de contact impliquant les ARNr se trouvent plutôt dans la
région centrale, très proche des sites fonctionnels, alors que celles impliquant les protéines se situent en périphérie. L’analyse de la structure d’un ribosome 70S associé à une molécule d’ARNm et à plusieurs ARNt a permis aussi d’avancer dans la compréhension des mécanismes moléculaires du décodage de l’information génétique et de la synthèse protéique. Ainsi, la comparaison des différentes structures de ces ribosomes, en présence ou en absence d’ARNt (Korostelev et al., 2006), a mis en évidence que le déplacement du ribosome et l’enchaînement des différentes étapes reposent sur des changements de conformation de cette RNP.
Les structures les plus récentes de ces ribosomes ont révélé que les ARNt se situent dans une zone du ribosome composée majoritairement d’ARNr. Les trois ARNt sont globalement orientés de la même façon par rapport aux deux sous-unités. À savoir, la tige boucle portant l’anticodon interagit avec la petite sous-unité alors que le reste de cette molécule lie la grande sous-unité (figure 6). La portion amino-acylée des ARNt se trouve dans la grande sous-unité au niveau du centre peptidyltranférase, afin de permettre la synthèse de la liaison peptidique. Enfin, l’ARNm associé au ribosome se courbe au niveau des sites A et P, ce qui garantit le positionnement correct des codons et anticodons.
4. Structure du ribosome 80S des eucaryotes
La majorité des études sur la structure des ribosomes a été réalisée à partir de ribosomes issus des bactéries. Les quelques données disponibles sur la structure des ribosomes eucaryotes semblent indiquer que ces structures sont globalement conservées. Très récemment, Chandramouli et ses collaborateurs ont résolu une structure par cryomicroscopie électronique d’un ribosome de chien à 8.7 Å (Chandramouli et al., 2008). La première observation est que l’architecture globale des ribosomes est conservée entre la bactérie et les mammifères, malgré la présence d’un plus grand nombre de protéines ribosomiques. l’implication de protéines ribosomiques plus nombreuses. Il semble exister aussi des interactions supplémentaires entre les deux sous-unités.
Figure 7 : Mécanisme de décodage de l’information génétique
Adaptée de Rodnina et al., 2002
13
D. Bases moléculaires des activités du ribosome
1. Le décodage de l’information génétiqueCette fonction consiste au recrutement d’un ARNt aminoacylé présentant un anticodon parfaitement complémentaire au codon de l’ARNm au sein du site A du ribosome. C’est lors de cette étape que des erreurs dites de traduction se produisent. En effet, l’insertion d’un mauvais acide aminé peut avoir des répercutions très importantes sur la fonction de la protéine nouvellement synthétisée. Il existe plusieurs revues détaillant les étapes nécessaires à la reconnaissance des ARNt corrects (Daviter et al., 2006; Rodnina et al., 2002). Dans un premier temps, le complexe ARNt aminoacylé/ EF-Tu(GTP) est recruté au sein du site A. Puis, l’appariement correct entre le codon et l’anticodon entraîne un changement de conformation du ribosome aboutissant à la stabilisation du complexe ARNt-ARNm et à l’hydrolyse du GTP. Le facteur EF-Tu(GDP) se dissocie alors du ribosome et se produit l’étape d’accommodation qui consiste au basculement de l’ARNt vers le centre peptidyltransférase (figure 7). Il semble que le processus de sélection de l’ARNt se fasse en deux étapes : tout d’abord lors de la première interaction entre le codon et l’anticodon puis après l’hydrolyse du GTP. Le mécanisme de décodage a lieu au sein de la petite sous-unité dans une région principalement composée de l’ARNr 16S dont les hélices 18, 27, 34 et 44. Ces hélices sont en contact avec le complexe ARNt-ARNm et vont être capables de stabiliser uniquement les bons appariements et ainsi participer à la sélection de l’ARNt.
2. La synthèse de la liaison peptidique
La formation de la liaison peptique est essentielle à la production des protéines. En effet, elle permet d’établir la liaison entre deux acides aminés. Lors de la synthèse peptidique, le centre peptidyl transférase opère le transfert de la chaîne peptidique portée par l’ARNt positionné dans le site P sur l’ARNt aminoacylé positionné dans le site A. Les nombreuses études réalisées sur le fonctionnement du ribosome ont rapidement permis de conclure que l’activité peptidyltransférase est portée par la grande sous-unité.
Le processus chimique permettant la formation de la liaison peptidique est bien connu. L’atome d’azote du groupement amine de l’aminoacyl-ARNt présent au site A va opérer une attaque nucléophile sur le carbone portant la fonction ester du peptidyl-ARNt (méthionine-ARNt lors du premier cycle) présent au site P du ribosome. De cette réaction résulte la formation d’une liaison peptidique entre le groupement C-terminal de la chaîne peptidique en
Figure 8 : Formation de la liaison peptidique
Adaptée de Rodnina et al., 2006
Figure 9 : Le PTC est uniquement constitué d’ARN
A- Localisation du PTC au niveau de la grande sous-unité ribosomique, les ARNr sont en
orange (23S) et poupre (5S), les protéines en violet. B- Distance entre le PTC et les protéines les plus proches en Å.
Adaptée de Cech et al., 2000 et de Nissen et al., 2000
14
cours de synthèse et le groupement amine de l’acide aminé entrant. Ce qui entraîne le transfert de la chaîne peptidique (méthionine seule lors du premier cycle) sur l’ARN de transfert aminoacylé du site A. L’ARNt du site P se retrouve alors désacylé (figure 8).
Le groupe de Steitz a publié en 2000 la strucutre cristallographique de la grande sous-unité de Haloarcula marismortui en présence de molécules analogues aux ARNt (Ban et al., 2000). Dans cette étude, il a été montré que le ribosome est un ribozyme, c’est-à-dire que l’activité catalytique est portée par l’ARNr de 23S et non pas par une protéine ribosomique. En effet, les ARNt sont positionnés dans la grande sous-unité au niveau d’une région uniquement constituée par l’ARNr 23S. Les protéines les plus proches se situent à 18.4 Å de l’ARNt, ce qui ne leur permet pas de participer à la formation de la liaison peptidique (Cech, 2000) (figure 9). Néanmoins, plusieurs études ont mis en évidence qu’aucune mutation des nucléotides de l’ARNr 23S de cette région n’avait d’effet remarquable sur la catalyse de la liaison peptidique. Il a donc été proposé que les nucléotides du site actif du ribosome permettraient de rapprocher les groupements réactifs des ARNt et de les positionner correctement les uns par rapport aux autres. De plus, le ribosome, et principalement l’ARNr 23S, formerait un environnement électrostatique favorable à la réaction chimique sans toute fois y participer directement (Rodnina et al., 2007) .
Dans l’étude du groupe de Steitz en 2000, on trouve aussi une description détaillée du tunnel de sortie du polypeptide au sein de la grande sous-unité. Ce tunnel débute au PTC pour déboucher à la surface de la grande sous-unité (figure 10). Il est constitué d’ARNr 23S et de protéines, et son diamètre varie entre 10 et 28 Å. Plusieurs études ont tenté de montrer une implication de ce tunnel dans le repliement des protéines. Mais il semble que ce tunnel ait pour unique fonction de libérer la chaîne polypeptidique en cours de traduction (Steitz, 2008).
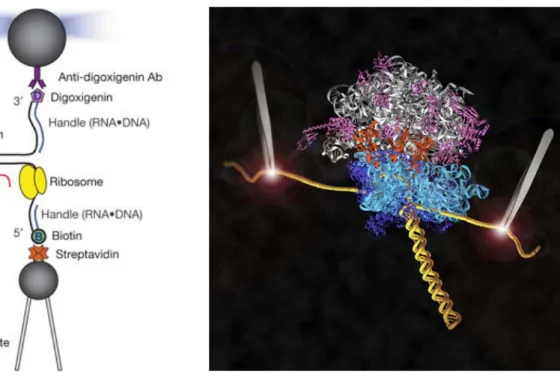
Comme nous avons pu le voir durant ce premier chapitre, les ribosomes sont des particules complexes dont le mode d’action est de mieux en mieux compris grâce à la cristallographie. Très récemment, une nouvelle technique est apparue et semble pouvoir permettre d’avancer dans la compréhension des paramètres des réactions nécessaires à la traduction : c’est l’étude à l’échelle de la molécule unique des ribosomes. Ainsi Jin-Der Wen et ses collaborateurs (Wen et al., 2008) ont fixé un ARNm en tige-boucle entre deux pinces magnétiques. Puis ils ont suivi la traduction de cet ARNm par un ribosome (figure 11). Leur première conclusion est qu’en dépit de la présence ou de l’absence de structure secondaire la traduction des ARNm par les ribosomes est continue mais passe par des étapes successives de
Figure 10 : Coupe transversale de la grande sous-unité 50S
15
Tranlocation-Pause-Translocation. Il est à parier que l’utilisation d’une telle technique permettra de tester de nombreux paramètres de la traduction telles que la séquence et la structure de l’ARNm, la composition en protéines ribosomiques, la nature des ARNt …
Figure 11 : Observation à l’échelle de la molécule unique de l’activité
traductionnelle d’un ribosome
16
II. La biogenèse des ribosomes
A. Présentation du processus
La biogenèse des ribosomes est un processus des plus importants et des plus coûteux en énergie de la cellule. Chez les eucaryotes, il nécessite l’action coordonnée des trois ARN polymérases, mobilisant ainsi 60% des efforts transcriptionnels de la cellule (Warner, 1999) . L’ARN polymérase II (pol II) est requise pour la synthèse des protéines ribosomiques (RP, présentes dans les sous-unités matures) et des autres facteurs impliqués dans la synthèse des ribosomes, dont la transcription d’une partie est co-régulée : on appel l’ensemble de ces gènes le régulon Ribi (Jorgensen et al., 2004; Wade et al., 2001). L’ARN polymérase III (pol III) produit le précurseur de l’ARNr 5S et l’ARN polymérase I (pol I) permet la transcription du précurseur commun aux ARNr 5.8S, 18S et 25S (levure)/28S (mammifères). Ces précurseurs sont dits pré-ARNr et vont devoir subir de nombreuses étapes de modifications chimiques, impliquant de petits ARN nucléolaires (snoARN), et des clivages endo- et exonucléolytiques permettant la suppression de séquences additionnelles appelées « espaceurs ». Dans tous les organismes, la production des ARN ribosomiques matures ne résulte pas directement de la transcription mais requiert une série d’étapes post-transcriptionnelles. Cette maturation à lieu de concert avec l’assemblage de la soixantaine de protéines ribosomiques. Le processus, permettant de délivrer les sous-unités ribosomiques matures, est très régulé dans le temps et dans l’espace. Il débute avec la transcription des pré-ARNr dans un compartiment nucléaire spécifique : le nucléole. Les particules pré-ribosomiques transitent alors par le nucléoplasme puis au travers des pores nucléaires pour être exportées dans le cytoplasme, lieu des dernières étapes de maturation.
La majorité des études permettant de mieux comprendre ce processus, ont été réalisées chez la levure Saccharomyces cerevisiae, car cet organisme permet l’utilisation de beaucoup d’outils génétiques, biochimiques, moléculaires et cellulaires. Depuis quelques années des études sont menées sur des cellules de mammifères et la première des constatations est qu’hormis quelques variations, la biogenèse des ribosomes est un processus dont les mécanismes et les facteurs impliqués sont bien conservés. La plupart des informations de ce chapitre viendront donc d’études réalisées chez la levure mais sont souvent transposables chez l’homme. J’essaierai de souligner les différences majeures quand elles existent (Petes, 1979).
A
B
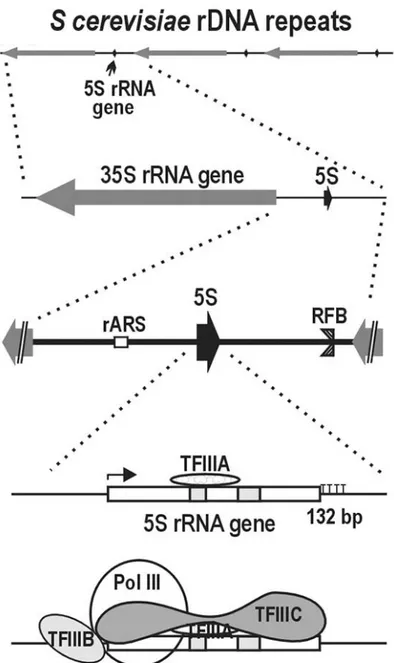
Figure 12 : Organisation génomique de l’ADN ribosomique chez
Saccharomyces cerevisiae
A- Représentation d’une unité ribosomique (Pr, promoteur ; Tr, terminateur ; En, activateur). B- Agrandissement d’une région intergénique.
17
B. Organisation de l’ADN ribosomique chez Saccharomyces
cerevisiae
Les gènes codant les ARNr chez S. cerevisiae sont contenus dans une portion génomique d’une longueur de 9,1 kb répétée en tandem nommée unité ribosomique (figure
12a). Cette unité contient les séquences des ARN ribosomiques de 18S, 5.8S, 25S et 5S ainsi
qu’une origine de réplication. Une unité d’ADN ribosomique contient deux unités de transcription indépendantes et orientées en sens contraire. La première, transcrite par l’ARN polymérase I, code le pré-ARNr de 35S contenant les séquences des ARN ribosomiques de 18S, 5.8S et 25S. La seconde, transcrite par l’ARN polymérase III, code l’ARNr de 5S. Ces deux gènes sont séparés par des régions intergéniques IGS1(NTS1) et IGS2(NTS2). Cette unité d’ADN ribosomique est répétée en tandem sur le bras droit du chromosome XII environ 100 à 200 fois, selon l’âge et le fond génétique de la souche (Petes and Smolik-Utlaut, 1979). Ce cluster est retrouvé dans la littérature sous l’appellation de « régions organisatrices du nucléole (NORs) ». Chez les eucaryotes supérieurs ces NORs sont répartis sur plusieurs chromosomes. Il a été raporté que seulement 50% des gènes des ADNr sont transcrits (Warner, 1999), même pour les cellules en phase exponentielle de croissance. Dans l’IGS2, on retrouve une origine de réplication autonome ribosomique (rARS) et une séquence d’association à la cohésine (CAR) (Laloraya et al., 2000). D’autre part, l’IGS1 renferme une barrière pour stopper les fourches de réplication : « replication fork barrier (RFB) » (Voelkel-Meiman et al., 1987), à laquelle s’ajoute une séquence de 400nt (EXP) qui semble importante pour le contrôle du nombre de copies (Kobayashi et al., 1992) (figure 12b).
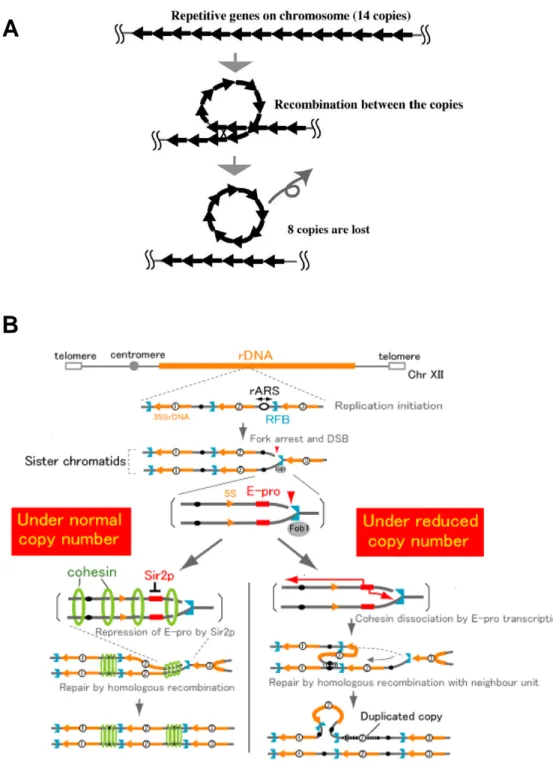
Alors que la répétition de ces gènes suggère que de nombreux événements de recombinaison (figure 13a) peuvent exister, générant des entités toxiques pour la cellules, les ADNr circulaires extra-chromosomiques (ERCs), il est remarquable que le nombre de copies dans les organismes soit maintenu. Cependant, il semble que, dans certaines conditions, ces organismes soient capables de réguler le nombre de copies afin d’assurer une production suffisante d’ARNr (Oakes et al., 1999; Vu et al., 1999).
Les mécanismes moléculaires permettant ces variations sont de plus en plus étudiés. Ils semblent reposer sur des événements de recombinaison inégaux, qui sont normalement proscrits par la cohésine associée aux séquences CAR. Effectivement, la cohésine assure qu’en cas de recombinaison, celle-ci se réalise avec la répétition alignée sur la chromatide sœur, ce qui n’a aucun effet sur le nombre de copies d’ADNr. Il est donc nécessaire pour générer une variation du nombre de répétitions de l’ADNr que la cohésine soit déplacée de
A
B
Figure 13 : Modèle de duplication d’unités ribosomiques par recombinaison
A- Principe de recombinaison dans une région répétée classique. B- Modèle de duplicationd’unité ribosomique dans un organisme qui ne possède pas assez de ces unités. Adaptée de Kobayashi 2006
18
ces loci. La perte d’expression de l’histone déacétylase Sir2p augmente le nombre d’événements de recombinaison des ADNr (Gottlieb et al., 1989) et de transcription des régions IGS1 et IGS2 par l’ARN polymérase II (Li et al., 2006a). Les ARN non codants de ces régions semblent être capables de déplacer la cohésine (Kobayashi and Ganley, 2005). Ensuite, il semble que les protéines Rad52p et Mre11p, impliquées dans la réparation des cassures à ADN double brins (DSB) soient nécessaires à l’amplification du nombre de répétitions des unités ribosomiques (Yano et al., 2004). D’autre part, la protéine Fob1 qui permet de bloquer la réplication de l’ADN dans un seul sens en se fixant à la séquence RFB, semble aussi impliquée (Kobayashi et al., 1998). L’ensemble de ces informations ont permis d’établir un modèle expliquant comment la combinaison de la réplication et d’une cassure double brin peut aboutir à la production d’une chromatide sœur ayant un nombre de répétitions plus élevé à la fin de la phase S du cycle cellulaire (figure 13b) (Kobayashi et al., 2004). Il est à noter que ce modèle est aussi valable pour la diminution du nombre de répétitions. Enfin, très récemment il a été montré que la protéine Trf4p protéine du complexe TRAMP, qui stimule la dégradation d’ARN nucléaires par l’exosome, est impliquée dans la régulation du nombre de répétition d’ADNr (Houseley et al., 2007). Cette régulation semble passer par le contrôle de la stabilité d’un ARN non codant, transcrit à partir de l’IGS1 : IGS1-R.
C. Le Nucléole
Au départ il a été décrit comme étant une usine à ribosome, c’est-à-dire totalement dévolu à la synthèse de ces RNP (Warner, 1990). Nous allons voir qu’il est maintenant admis que ce compartiment est aussi essentiel pour d’autres processus cellulaires. La majorité des études sur ce compartiment proviennent de quatre organismes : La levure, le xénope, la drosophile et l’homme. Alors que plusieurs propriétés du nucléole sont conservées entre ces organismes, certaines différences existent. La divergence la plus importante est que le nucléole des mammifères se désassemble durant la mitose contrairement à celui de la levure qui reste intact tout au long du cycle cellulaire. Je vais ici me limiter à exposer les propriétés conservées ou extrapolables entre les différents organismes.
1. Structure du nucléole
L’analyse de la structure des nucléoles repose majoritairement sur les avancées en microscopie électronique. La première des informations est que le nucléole ne possède pas de
Figure 14 : Observation par microscopie électronique du nucléole de S.
cerevisiae
A-Observation de la cellule entière (RE : Réticulum Endoplasmique ; CH : Nucléoplasme ;
Nu : Nucléole). B-Observation du nucléole (FC : centre fibrillaire ; DFC : centre fibrillaire dense ; GC : composant granulaire).
19
membrane, il correspond donc au regroupement de différents facteurs au sein du noyau. Couramment, le nucléole est séparé en trois sous-compartiments : Le centre fibrillaire (F ou FC), il est entouré du composant fibrillaire dense (D ou DFC) et à la périphérie on trouve le composant granulaire (G ou GC) (figure 14). Selon l’organisme, la répartition du volume nucléolaire entre ces trois compartiments peut varier. A côté de ces différents compartiments, des brins plus ou moins condensés de chromatine peuvent pénétrer à l’intérieur du nucléole complexifiant ainsi son organisation. De plus, chez les plantes on trouve des cavités ou des vacuoles. La transition entre ces différents domaines n’est pas toujours claire. Il faut donc plutôt imaginer le nucléole comme un compartiment nucléaire réparti en plusieurs zones dont les séparations ne sont pas franches. Il est plus juste d’utiliser la notion de territoires, comme nous allons le voir par la suite. L’existence même de ces trois compartiments est sujette à caution pour la levure. En effet, il existe une hypothèse selon laquelle la levure ne possèderait que deux compartiments (F et G) au lieu de trois (Thiry and Lafontaine, 2005).
Enfin, des études avec différents polymères de glucose montrent que des macromolécules jusqu’à 2000 KDa peuvent pénétrer dans le nucléole (Handwerger et al., 2005). Le nucléole peut donc être comparé à une éponge au sein du noyau qui retiendrait certains facteurs, ce qui est cohérent avec l’aspect très dynamique de la structure des nucléoles.
2. Dynamique structurale du nucléole
La structure globale du nucléole est largement tributaire de son activité de maturation des ribosomes. Ainsi des souches de levure ne possédant plus d’ADNr chromosomique mais transformées par des plasmides comportant ces ADNr, renferment de multiples petits nucléoles à la place de l’unique nucléole normalement observé (Oakes et al., 1998). Le fait que ces mutants soient viables démontre que la structuration nucléolaire classique n’est pas nécessaire à la fonction du nucléole. De plus, si le promoteur pol I de l’ADNr est remplacé par un promoteur pol II sur ces plasmides, on observe un nucléole compact qui ne possède plus de contacts avec l’enveloppe nucléaire (Oakes et al., 1998). Ceci suggère que l’ARN pol I n’est pas strictement nécessaire à la biogenèse des ribosomes mais que la nature de la polymérase qui transcrit les ARNr influe sur la structure de nucléole. Ensuite, il a été montré que les transcrits ribosomiques eux-mêmes, plus que la pol I, sont requis pour l’organisation du nucléole (Leger-Silvestre and Gas, 2004). De plus, beaucoup de facteurs intervenant dans la biogenèse des ribosomes ont un effet sur la structure du nucléole chez la levure
Figure 15 : Les protéines nucléolaires
Répartition des 726 protéines purifiées à partir des nucléoles chez l’homme en fonction de leur rôle dans la vie cellulaire.
20
ribosomes possèdent un unique petit nucléole appelé « nucléole en anneau » qui contient juste un FC (Raska et al., 1983).
Enfin, chez les mammifères lors de l’entrée de la cellule en mitose, la transcription s’arrête et le nucléole se désagrège. Les mécanismes moléculaires impliqués dans ce processus sont de mieux en mieux étudiés (pour revue voir (Boisvert et al., 2007)). Ces mécanismes reposent principalement sur l’inhibition de la transcription par l’ARN pol I via la phosphorylation du facteur de transcription SL1 (Heix et al., 1998). Lors de la mitose subsistent des dérivés nucléolaires (NDF) renfermant notamment des snoRNP (Dundr and Olson, 1998). La reformation du nucléole est très rapide après la mitose ou après des traitements visant à le déstabiliser comme le photo-blanchiement . Cette faculté du nucléole est en corrélation avec sa structure sans membrane reposant sur l’association de différents facteurs.
3. Composition du nucléole
Afin de déterminer la composition de ce compartiment nucléaire plusieurs approches ont été tentées. Des études immunologiques ont révélé que la composition protéique de ce compartiment était différente de celle du nucléoplasme, et que donc certaines protéines pouvaient êtres spécifiques du nucléole, comme la nucléoline et la fibrillarine chez les eucaryotes supérieures, ou comme certaines protéines de S. cerevisiae décrites comme nucléolaires « NucleOlar Proteins » (NOP). Il apparaît pourtant que ces protéines sont aussi retrouvées dans le nucléoplasme et c’est une accumulation nucléolaire plutôt qu’un adressage total qu’il faut envisager lorsqu’on parle de protéines nucléolaires.
Ensuite, le nucléole des cellules de mammifères a été purifié et sa composition protéique a été analysée par spectrométrie de masse (Andersen et al., 2002). De même, les résultats d’analyse de la composition en protéines du nucléole de plusieurs organismes ont été croisés avec les données issues d’études à grandes échelles sur des interactions entre les protéines de la levure S. cerevisiae. Ceci a permis de définir la composition protéique du nucléole de levure (Staub et al., 2006). Les informations principales issues de ces études sont d’une part que 90% des protéines nucléolaires humaines ont un homologue chez S. cerevisae (Andersen et al., 2005). D’autre part, les fonctions connues de ces différentes protéines ne sont pas uniquement reliées à la biogenèse des ribosomes (30%) mais aussi à d’autres processus cellulaires (figure 15), ce qui a encouragé différentes équipes à étudier les autres fonctions du nucléole. Enfin, l’identifiaction des protéines du nucléole de cellules Hela après
21
différents traitements indique que la composition protéique est dynamique et peut varier selon les besoins cellulaires (Andersen et al., 2005).
4. Le nucléole : un compartiment multifonctionnel
Depuis une dizaine d’années, plusieurs études sont menées afin d’identifier les différentes fonctions du nucléole. Le principal processus qui se déroule dans ce compartiment est la transcription de l’ADNr par la pol I (voir chapitre : La trancription par l’ARN Polymérase I). Mais il semble que nucléole soit un compartiment nucléaire permettant de réguler d’autres processus cellulaires, peut-être en lien avec la régulation de la biogenèse des ribosomes :
La régulation du cycle cellulaire est une autre des fonctions du nucléole. Les
modifications post-traductionnelles sont les changements dynamiques les plus observés durant la progression du cycle cellulaire. Plusieurs de ces modifications, comme la sumoylation et la phosphorylation, sont régulées par le nucléole. Ainsi SENP5, une protéase qui reconnaît spécifiquement les groupements SUMO des protéines, est majoritairement localisée dans le nucléole. Or, l’inhibition de SENP5 par interférence à l’ARN engendre des défauts de division (Di Bacco et al., 2006) .
Chez la levure, Cdc14p est une phosphatase essentielle à la sortie de mitose. Elle permet d’abolir les phosphorylations dépendantes de Cdh1 qui régulent la mitose. Or, Cdc14p est séquestrée dans le nucléole, par la protéine Net1p, dans un état inactif durant l’interphase pour éviter une sortie de mitose non coordonnée (Shou et al., 1999). La libération de Cdc14p (FEAR) nécessite la phosphorylation de Net1p.
La séquestration de protéines spécifiques dans le nucléole semble être une propriété couramment utilisée par la cellule pour réguler des activités spécifiques durant le cycle cellulaire. Ainsi, la reverse transcriptase de la télomérase est retenue dans le nucléole jusqu’à ce que les télomères soient répliqués en fin de phase S (Wong et al., 2002). Cette séquestration semble être réalisée par la liaison de la reverse transcriptase à la nucléoline (Khurts et al., 2004).
Ainsi le nucléole permet de séparer certaines enzymes, intervenant au cours du cycle cellulaire, du reste du nucléoplasme pour les relâcher uniquement lorsque leur action est requise.
22
La réponse au Stress est un autre processus cellulaire dans lequel le nucléole est
impliqué. En effet, l’exposition de la cellule à différents stress du type dommages à l’ADN ou hypoxie, provoque une stabilisation de p53, ce qui induit un arrêt du cycle cellulaire et la mise en place de l’apoptose. La stabilité de p53 est régulée par l’association des protéines HDM2 et p14ARF chez les humains. HDM2 est une ubiquitine ligase spécifique de p53 qui permet la dégradation normale de p53 par le protéasome. Or, suite à l’exposition de la cellule à différents stress, l’expression de p14ARF est augmentée. Cette protéine étant majoritairement nucléolaire, il a été proposé que l’augmentation de son expression provoquait la séquestration d’HDM2 dans le nucléole. Ceci entraîne la stabilisation de p53 qui n’est alors plus dégradée par le protéasome (Wsierska-Gadek and Horky, 2003). De même en réponse au stress, le facteur de transcription TIF-IA est exclu du nucléole, ce qui a pour effet d’arrêter la transcription par l’ARN polymérase I.
Enfin, le nucléole est capable de réguler la biogenèse d’autres RNP que les
particules ribosomiques. Alors que la fonction principale du nucléole est la transcription des
ADNr et la maturation des ARNr, il est actuellement clair qu’il est aussi impliqué dans la maturation d’autres ARN cellulaires.
La particule de reconnaissance du signal (SRP) résulte de l’association de six protéines avec un ARN. Sa fonction est de bloquer la traduction de certaines protéines afin qu’elle se poursuive dans le réticulum endoplasmique. Il a été montré que les composants de ce complexe transitaient par le nucléole avant d’être exporté dans le cytoplasme où il exerce sa fonction chez les mammifères (Jacobson and Pederson, 1998). Il est possible que le nucléole aide à l’assemblage de ce complexe, mais on peut aussi envisager qu’il permette l’association de la particule SRP avec les ribosomes en cours de formation.
La RNase P est une RNP requise pour la maturation des pré-ARNt, dont le composant ARN est présent dans le nucléoplasme mais aussi dans le nucléole (Jacobson et al., 1997). Il est possible que le nucléole permette l’assemblage de la particule ou que certains pré-ARNt soient maturés dans le nucléole. De même la snRNP U6 est retrouvée dans le nucléole. Il est possible que l’ARN de cette particule nécessite un passage dans ce compartiment afin de subir quelques modifications chimiques (Gerbi et al., 2003). Les pré-ARNt, l’ARN de la RNase P et l’ARN U6 sont synthétisés par la pol III en dehors du nucléole comme l’ARNr 5S (chez les mamifères). Il doit donc exister une voie permettant de coupler des transcriptions par l’ARN pol III à l’importation dans le nucléole des ARN néo-synthétisés.
Figure 16 : Initiation de la transcription Pol III chez S. cerevisiae
23
Le nucléole semble aussi être le siège d’un autre mécanisme moléculaire : l’édition de certains ARN par l’enzyme ADAR2 (Desterro et al., 2003; Vitali et al., 2005). Il est aussi un lieu de stockage et/ou de maturation de la machinerie d’interférence à ARN que ce soit les petits ARN d’interférence (siARN) (Pontes et al., 2006) ou les micro ARN (miRNA) (Politz et al., 2006).
Le nucléole est donc un compartiment nucléaire, conservé chez les eucaryotes, dont la principale fonction est de permettre la biogenèse des ribosomes. Cependant de nouvelles données ont démontré que ce compartiment pouvait jouer d’autres rôles dans la survie cellulaire. Ces différentes fonctions annexes pouvant toutes être reliées à la biogenèse des ribosomes ou à la fonction des ribosomes à l’exception de la régulation des siARN. En effet, le contrôle du cycle cellulaire, la réponse au stress et la formation des snoRNP sont des processus reliés à la biogenèse des ribosomes (voir revue à la fin de l’introduction). Alors que l’assemblage de la snRNP U6, le contrôle de la formation de la RNase P, la formation de la particule SRP, l’édition des pré-ARNm et de l’activité des miARN sont reliés à la fonction des ribosomes. Il est donc possible d’envisager deux hypothèses pour expliquer le caractère multifonctionnel du nucléole : La cellule utilise ce compartiment nucléaire pour concentrer en un seul lieu les événements nécessaires à la formation de ribosomes fonctionnels, ce qui signifie que la régulation des siARN a un lien encore méconnu avec la fonction des ribosomes. Ou alors, la cellule utilise ce compartiment nucléaire comme lieu de stockage, car il permet de séparer certains composants du reste du nucléoplasme, pour différents processus.
Les dernières études sur la dynamique de la composition de ce nucléole indiquent que d’autres fonctions peuvent encore être envisagées pour ce compartiment telles que l’assemblage de la télomérase ou de « exon junction complex »(EJC). Reste à savoir si elle seront reliées à la formation de ribosomes fonctionnels. Dans tous les cas, il est certains que ce compartiment joue un rôle clé dans la régulation des événements nucléaires en général et dans la biogenèse des ribosomes en particulier.
D. La transcription de l’ARN ribosomique 5S
La transcription du pré-ARNr 5S est assurée par l’ARN pol III, à la différence des autres ARN ribosomiques transcrits par l’ARN pol I. Il existe une différence entre les mammifères pour lesquels la transcription du pré-ARNr 5S se fait dans une région
péri-Figure 17 : Observation des ADNr en cours de transcription
On observe par microscopie électronique les arbres de Miller, dont chaque branche correspond à un pré-ARNr en cours de transcription. Les flèches indiquent les structures appelées « terminal knobs » correspondant à l’association co-transcriptionnelle de certains facteurs intervenant dans la maturation des pré-ARNr.
24
nucléolaire, et la levure S. cerevisiae qui possède l’ADNr 5S au milieu de l’ADNr transcrit par la pol I. En effet, chez la levure, il est supposé que cette transcription a lieu dans le nucléole même si pour le moment aucune évidence expérimentale ne permet de l’affirmer. Dans tous les organismes eucaryotes cette transcription nécessite trois facteurs : TFIIIA qui se lie spécifiquement au promoteur interne du gène codant l’ARNr 5S ; TFIIIB et TFIIIC qui sont requis pour toutes les transcriptions réalisées par l’ARN pol III (Braun et al., 1992; Kassavetis et al., 1990) (figure 16). Récemment la transcription de l’ARNr 5S a pu être observée sur des arbres de Miller, ce qui a permis d’étudier l’impact de facteurs environnementaux tels que des stress nutritionnels sur l’activité de l’ARN pol III (French et al., 2008). L’extrémité 5’ du 5S semble correspondre au point +1 de la transcription et ne requiert donc pas de maturation. Alors que l’extrémité 3’ du pré-ARNr 5S est étendue de 10 à 15 nucléotides par rapport à l’extrémité 3’ de l’ARNr 5S (Piper et al., 1983). L’élimination de cette extrémité nécessite l’action de l’exonucléase Rex1p (van Hoof et al., 2000).
Les détails concernant l’incorporation de cet ARNr aux particules pré-ribosomiques sont relatés dans la revue présente à la fin de cette introduction.
E. La transcription par l’ARN polymérase I
La transcription de l’ADN ribosomique codant les ARNr 18S, 25S et 5.8S est assurée par l’ARN polymérase I. L’ARN polymérase I (pol I) est entièrement dévouée à la transcription des ADNr qui représente à elle seule 50% des efforts transcriptionnels de la cellule. Ce mécanisme peut être observé en analysant par microscopie électronique des ADNr, issus de la purification du nucléole, en cours de transcription. Ainsi Miller a observé ces structures qu’il a appelées « arbres de Noël » (McKnight and Miller, 1976). L’observation de ces structures lui a permis, ainsi qu’à d’autres équipes, de montrer que la transcription des ADNr n’était pas homogène (certaines unités sont actives, d’autres non). De plus, l’assemblage de protéines sur l’ARNr pouvait être co-transcriptionnel, formant des structures dites « terminal knobs » (Dragon et al., 2002; French et al., 2003; Osheim et al., 2004; Osheim et al., 1996) (figure 17).
Ce mécanisme semble fondamentalement conservé chez les eucaryotes. Les différences remarquables entre les différents organismes eucaryotes doivent être le reflet du manque d’informations sur les mécanismes moléculaires de ce processus dans certains organismes. En effet, certaines différences constatées lors de la comparaison des études effectuées sur plusieurs organismes il y a quelques années, ne semblent plus valides
Tableau 2 : Composition protéique des différentes ARN polymérases
*Protéines formant le noyau des ARN polymérases ARN polymérase
Ensemble protéique
Pol I Pol II Pol III
Protéines communes (Rpb5, Rbp6, Rpb8, Rpb10, Rpb12)* Protéines paralogues Rpa190* Rpa135* Rpc40* Rpc19* Rpb1* Rpb2* Rpb3* Rpb11* Rpc160* Rpc128* Rpc40* Rpc19* Protéines spécifiques Rpa12* Rpa14* Rpa43* Rpa34 Rpa49 Rpb7* Rpb9* Rpb4* Rpc17* Rpc11* Rpc25* Rpc31 Rpc34 Rpc37 Rpc53 Rpc82
25
maintenant. Je vais donc tenter de présenter un mécanisme général de la transcription par la Pol I. Je m’attacherai à présenter en priorité les connaissances issues des études sur la levure
S. cerevisiae, organisme dans lequel j’ai effectué mes travaux de recherche. Puis je
présenterai les différentes régulations qui peuvent affecter ce processus.
1. Composition de l’ARN polymérase I
La Pol I est un complexe enzymatique constitué de 14 unités protéiques. 12 de ces unités forment un coeur protéique retrouvé dans les trois ARN polymérases. Ce coeur est composé de cinq protéines qui sont communes au trois polymérases : Rpb5, Rpb6, Rpb8, Rpb10 et Rpb12 ; et de sept paralogues présents dans les différentes ARN polymérases. Deux autres protéines sont spécifiques de la Pol I (Tableau 2). Plusieurs de ces protéines possèdent des orthologues chez les bactéries et les archées (Kwapisz et al., 2008).
2. Formation du complexe d’initiation
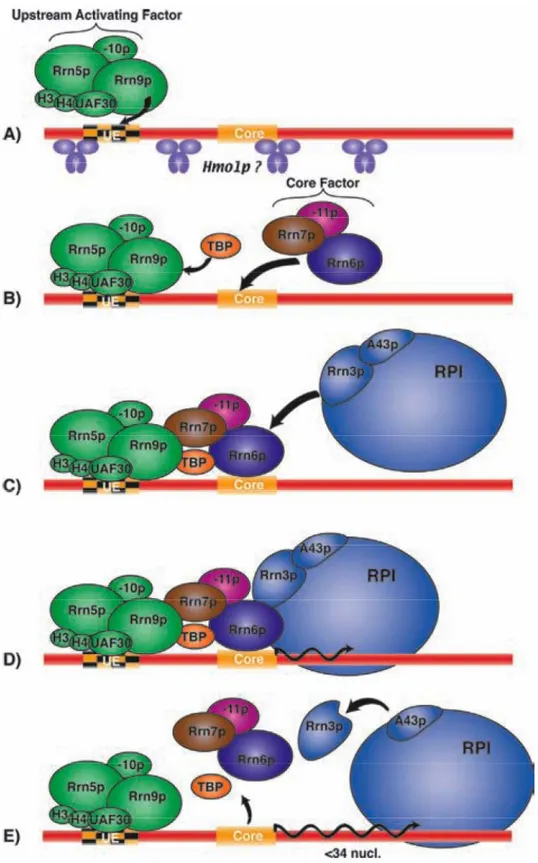
Chez les vertébrés et la levure, le promoteur Pol I est une région d’environ 150pb. Cependant, la conservation de la séquence entre les espèces est très faible, elle correspond à la spécificité de la pol I à une espèce et à l’évolution rapide des séquences intergéniques des ARNr. Néanmoins, la plupart des promoteurs pol I contiennent deux éléments distincts que sont le promoteur central (Core) et une séquence située en amont de ce promoteur (UPE ou UE). Il semble que la distance entre ces deux régions soit cruciale pour la transcription Pol I
in vivo, mais que la région Core soit suffisante in vitro (Keener et al., 1998; Verbeet et al.,
1984). Il est admis que la formation d’un complexe d’initiation avec la Pol I requiert une protéine qui est capable de reconnaître la boîte TATA (TBP) et plusieurs protéines spécifiques de la pol I capables de s’associer avec la TBP. L’ensemble de ces facteurs forme un complexe capable de s’associer spécifiquement aux promoteurs pol I.
Chez S. cerevisiae, deux facteurs sont requis pour l’initiation de la transcription, le facteur UAF, composé de plusieurs sous-unités protéiques, qui reconnaît l’UE et le facteur « core » (CF) qui reconnaît la région centrale du promoteur. Dans un premier temps, le facteur UAF se fixe spécifiquement sur la région UE puis la TBP s’associe à ce complexe (de manière indépendante ou directement associée au CF) (Lin et al., 1996; Steffan et al., 1996; Steffan et al., 1998). Ensuite, le CF s’associe à la région Core et l’ensemble des protéines fixées sur ce promoteur permet de recruter la Pol I par l’intermédiaire de la protéine Rrn3p. Il apparaît que la fixation de la Pol I au niveau du promoteur grâce au CF est stabilisée par la
Figure 18 : Etapes de l’initiation de la transcription par l’ARN Pol I
26
TBP. Enfin, UAF est strictement essentielle pour la transcription Pol I in vivo même si son absence peut-être compensée par un phénomène appelé « polymerase switching » qui permet la transcription de l’ADNr par la Pol II (Oakes et al., 1999). Les différentes étapes qui aboutissent à la formation du complexe d’initiation sont schématisées (figure 18).
3. Elongation de la transcription Pol I dépendante
Le recrutement de la Pol I par le CF permet de la positionner directement au point +1 de la transcription. Il est généralement admis qu’à la différence de la Pol II, la transcription Pol I ne nécessite pas d’hydrolyse de nucléotide triphosphate (NTP). L’initiation de la transcription nécessite l’association de la Pol I avec Rrn3 chez les mammifères et la levure (Yamamoto et al., 1996). Juste après l’initiation de la transcription, la protéine Rrn3p, la TBP et le CF se dissocient de la Pol I, alors que l’UAF semble rester associé à l’UE, ce qui pourrait permettre un deuxième cycle de transcription plus rapide (Aprikian et al., 2001). La dissociation de Rrn3p n’est pas nécessaire pour la poursuite de l’élongation dans la mesure où la fusion traductionnelle de Rrn3p avec la sous-unité A43 de la pol I n’affecte en rien la viabilité ou la croissance de la levure (Laferte et al., 2006).
Spt4p et Spt5p sont deux facteurs importants pour l’élongation de la transcription de la Pol II mais aussi de la Pol I (Schneider et al., 2006). Dans les deux cas ces facteurs semblent affecter la processivité de la polymérase.
Enfin un autre facteur semble nécessaire à la transcription Pol I chez la levure : la protéine Hmo1p. Cette protéine fait partie des protéines à boîtes HMG et devient essentielle en absence des protéines spécifiques de la Pol I, Rpa34p et Rpa49p (Gadal et al., 2002). Cette protéine pourrait être l’équivalent du facteur UBF des mammifères, ce qui serait un élément supplémentaire permettant de rapprocher les mécanismes de transcription par la Pol I chez les mammifères et la levure. Cette protéine s’associe à l’ADNr au niveau du promoteur des gènes ribosomiques mais aussi le long de ceux-ci (Hall et al., 2006; Kasahara et al., 2007). Très récemment, il a été montré qu’Hmo1 était spécifiquement associée aux unités ribosomiques actives (Merz et al., 2008). Il reste à définir si Hmo1 est recrutée par la Pol I ou si elle reconnaît les unités transcrites par la Pol I. Il est donc probable que comme la Pol II, l’activité de la Pol I soit modulée par des structures chromatiniennes spécifiques.
Figure 19 : Terminaison de la transcription Pol I
Schématisation de la terminaison de la transcription par l’ARN pol I en fonction des différents facteurs associés à l’ADN ribosomique.
27
4. Terminaison de la transcription Pol I
Comme nous avons pu le voir précédemment, les unités d’ADN ribosomiques sont très structurées avec des séparations entre les gènes dites IGS1 et IGS2. La RFB de l’IGS1 permet de fixer la protéine Fob1p, ce qui établit une structure qui bloque les fourches de réplication afin d’éviter qu’elles n’entrent en collision avec la machinerie de transcription Pol I (Huang et al., 2006). Les analyses de la transcription Pol I chez la levure ont montré que 90% des transcrits se terminent au niveau d’un premier terminateur (T1) et 10% au niveau d’un autre (T2). Le T1 correspond à une séquence riche en T située à 10nt d’un site de fixation de la protéine Reb1p. Une région identique est retrouvée en amont du T2 alors qu’aucune protéine n’a été identifiée comme interagissant spécifiquement avec cette région (Reeder et al., 1999; Reeder and Lang, 1997).
La terminaison de la transcription par la Pol II requiert la protéine Rat1p, une exonucléase 5’-3’. Après un clivage co-transcriptionnel réalisé par la machinerie de polyadénylation, Rat1p est capable de digérer l’ARN par son extrémité 5’ et ce de manière très efficace. Ce qui conduit à l’arrêt de la transcription. Ainsi, Rat1p est aussi appelée l’exonucléase « torpille ». Alors que les autres fonctions de cette exonucléase nucléaire peuvent être assumées par l’exonucléase Xrn1p en absence de Rat1p, la terminaison de la transcription n’est réalisée que par Rat1p (Henry et al., 1994; Luo et al., 2006). Or, il a été montré qu’un clivage co-transcriptionnel du pré-ARNr existe aussi. En effet, Rnt1p coupe le pré-ARNr dans sa région 3’ (Henras et al., 2004; Kufel et al., 1999). Il a été montré qu’en absence de Rnt1p ou avec l’expression d’une forme thermosensible de Rnt1p, la transcription se termine majoritairement en T2 et certaines transcriptions continuent même après ce terminateur (Catala et al., 2008). Enfin, il a été récemment montré que les extrémités 5’ générées par Rnt1p sont digérées par Rat1p. Ce qui permet la terminaison de la transcription de la Pol I de la même façon que pour la terminaison de la transcription Pol II (El Hage et al., 2008; Kawauchi et al., 2008) (figure 19).
5. La transcription Pol I : un processus hautement régulé
La croissance cellulaire est directement corrélée à la synthèse des ARNr. Le contrôle de la croissance cellulaire en fonction des nutriments disponibles et/ou des stress environnementaux s’établit par l’action de deux complexes : TORC1 et TORC2. Chez la levure TORC1, contrôle la transcription Pol I et la biogenèse des ribosomes. Lorsque l’on expose des cellules à la rapamycine ou à des stress, TORC1 est inactivé et la transcription Pol