Régulation post-transcriptionnelle de l'expression du
facteur de transcription ATF4 lors de stress induits
par des agents chimiothérapeutiques
Thèse
Pauline Adjibade
Doctorat en biologie cellulaire et moléculaire
Philosophiæ doctor (Ph. D.)
Régulation post-transcriptionnelle de l'expression
du facteur de transcription ATF4 lors de stress
induits par des agents chimiothérapeutiques
Thèse
Pauline
Adjibade
Sous
la
direction
de
:
Rachid Mazroui, directeur
de
recherche
Résumé
Lors d'un stress, les cellules eucaryotes activentdes mécanismes de défense afin de
s'adapter aux conditions extrêmes et de survivre. Ces mêmes mécanismes peuvent être
exploités par les cellules cancéreuses pour survivre au stress thérapeutique. La réponse
intégrée au stress (ISR) est une réaction cellulaire qui joue un rôle crucial dans l'adaptation des cellules face à différentsstress notammentenrégulant la transcriptionet la traduction de nombreux gènes cibles spécifiques. Durant cette réponse, les cellules stressées réduisent aussi leur métabolisme général, entre autres en inhibant la traduction des ARNm, et économisent ainsi l'énergie nécessaire pour réparer les dommages causés par le stress. En conditionsde stress, latraduction généraledesARNm est inhibée principalement au niveau de son étape d'initiation. Un des mécanismes clés induisantl'inhibition de l'initiation de la
traduction est la phosphorylation du facteur d'initiation de la traduction eIF2a (eukaryotic translation initiation factor2a). Bien que cette phosphorylation d'eIF2a, induite parle stress, entraine une inhibition globale de la synthèseprotéique, elle induit d'une part la formation
d'entités cytoplasmiques à ARN appelées granules de stress mais ellefavorise également la
traduction spécifique des ARNm spécifiques codant pour desfacteurs de réponse au stress,
dont le facteurdetranscription ATF4. Ce facteur joue un rôle clé lorsdela réponse au stress
soitenactivant les voiesde survie ouaucontraireenfavorisant celles de mort cellulaire.Ces
fonctions opposées d'ATF4 dans la réponsecellulaire au stress dépendent largementde son
niveau d'expression. Ainsi, nous avons cherché à caractériser le(s) mécanismes(s) de régulation de l'expression d'ATF4 lors de stress thérapeutiques au sein de cellules cancéreuses. Nous avons montré l'implication des granules de stress (GS) dans larégulation de l'expression de l'ARNm d'ATF4 lorsd'unstressthérapeutiqueengendré par letraitement avec l'agent chimiothérapeutique sorafenib au sein de cellules d'hépatocarcinomes. Cette
régulation implique la séquestration d'une fraction del'ARNm d'ATF4dans les GSinduites
par le traitement avec la sorafenib, prévenant sa surexpression létale ; ceci permet de
maintenir un niveau d'expression d'ATF4 basal mais nécessaire à la survie des cellules
cancéreuses. Afin de caractériser le(s) mécanisme(s) responsable(s) de l'expression basale
d'ATF4, permettantla résistance des cellules cancéreuses auxtraitements thérapeutiques, un
RNA pull-down utilisant des fragments d'ARN biotinylés d'ATF4 a l'aide d'extraits de
spectrométrie de masse des complexesprotéiquesassociés aux fragmentsd'ARN d'ATF4
biotinylés a permis l'identification d'une dizaine de protéines qui pourraient interagiravec
l'ARNm d'ATF4, dont l'ARN hélicase DDX3. Nous avons observé queDDX3 promeut au
niveautraductionnel l'expression d'AFT4. Des tests d'interaction protéine-protéine montrent
que DDX3 est un composant du complexe d'initiation de latraduction eIF4F. L'interaction
entre DDX3 etle complexe eIF4F est potentiellement médiéepar lefacteur eIF4G, qui est
également nécessaire à la traduction d'ATF4 induite en condition de stress. Enfin, la
diminutionde l'expression d'ATF4 au sein de cellules issues d'hépatocarcinomesrésistants
via la déplétion de DDX3 sensibilise les cellules cancéreuses à la mort induite par le
traitement avec la sorafenib. Cetteétude caractérise de nouveaux mécanismes de régulation de l'expression du facteur ATF4quise produit lorsde stress et quipourraient être ciblés afin
Abstract
During physiologicalorenvironmental stress, cells activate defensemechanismsto survive. These same mechanisms may be exploited by cancercells to survivetherapeutic stresses. The integrated stressresponse(ISR)is one ofthe cellresponsesthat play a crucial role in the
adaptation of cells tovarious stresses such as the transcriptional andtranslational régulation ofmany specific target genes. During this cell response, stressed cells reduce their general metabolism in part by inhibiting mRNA translation, thereby saving energy neededto repair
stress-induced damages. Under stressconditions,the regulation of translationoccursmainly
at its initiation step through the phosphorylation ofthe translation initiation factor eIF2a.
Whilethis phosphorylation, induced bystress, causesa global inhibitionof protein synthesis,
it induces onthe onehand theformation of cytoplasmic RNA entities called stress granules
(GS) but it also promotes the preferential translation of specific mRNAs coding for stress
response factors. Among those, ATF4is a master transcription factor that orchestratesgene expression during various stresses including those involved in cancer, by either activating
survival pathways or on the contrary promoting those of cell death. These opposite ATF4
functions in the cellular stress response depend largely on its expression level linked to its translational regulation by the phosphorylation of eIF2a. Thereby, we soughtto characterize
themechanisms(s) that regulate the expression of ATF4duringtherapeutic stresses in cancer
cells. We described theimplication of stress granules in the regulation of ATF4 expression
during stress induced by the treatment with sorafenib, a chemotherapeutic agent usedto treat
hepatocellular carcinoma (HCC). This novel mechanism involves the formation of stress
granules that sequester afraction of ATF4 mRNAin itsrepressed form, thus preventingits lethal overexpression. This resultedin abasal level of ATF4expression which is necessary
for the survival of cancercells. Then, in order to characterize themechanism(s)(e.g. RNA-
binding proteins and translation initiation factors such as eIF4G) responsible for the basal
translationof ATF4 mRNA allowing the resistance ofcancer cells totherapeutic treatments,
we performed a biotinylated RNA-pulldown assay with cancer cells extracts subjected to
sorafenib. Mass spectrometry analyses of the protein complexes associated with ATF4
biotinylated-RNA fragments ledto the identification of the DEAD-box RNA helicase DDX3,
aspotential interactor with ATF4 mRNA. Bycombining depletion experiments with various
ATF4 mRNA expression at the translational level. Protein-interaction assays identified
DDX3as a componentofthe translation initiation complex eIF4F. The interaction between
DDX3 and the eIF4F complex is potentially mediated by eIF4GI, which we found to be
required for sorafenib-induced ATF4 expression. Furthermore,reducing ATF4 expression in
resistant HCC cells through DDX3 depletion sensitizescancercells to Sorafenib-inducedcell
death. Thus, this study identified new regulatory pathways of ATF4 factor that could be targeted to prevent drug resistance in cancer cells and improve theconventionaltherapies.
Table des
matières
RÉSUMÉ ... II
ABSTRACT...IV TABLE DES MATIÈRES ...VI
LISTEDES FIGURES ...IX
LISTEDES TABLEAUX ... XI
LISTEDES ABRÉVIATIONS ... XII REMERCIEMENTS ...XV AVANT-PROPOS ...XVII
INTRODUCTION ... 1
1. GÉNÉRALITÉS ... 1
2. LA RÉPONSE CELLULAIRE AU STRESS ... 2
2.1. Reprogrammation de l'expressiongéniqueau niveau transcriptionnel ettraductionnel... 2
2.2. Latraduction ... 3
2.2.1. Généralités... 3
2.2.3. Mécanisme de l'initiation de la traduction ... 4
2.2.3.2. Formation du complexe de pré-initiation 48S et du monosome 80S ... 6
2.3. Régulation de l'initiation de la traduction encondition de stress... 8
2.3.1. Mécanismes de régulation de l'initiation de la traduction ... 8
2.3.2. Les kinases eIF2a (HRI, GCN2, PERK et PKR) ... 14
2.3.3. Régulation de la traduction via la phosphorylation du facteur eIF2a ... 19
2.3.4. Phosphorylation d'eIF2a et traduction sélective de certains ARNm ... 20
2.3.4.1. Réponse intégrée au stress et traduction spécifique de certains ARNm ... 20
2.3.4.2. Le facteur de transcription ATF4 ... 21
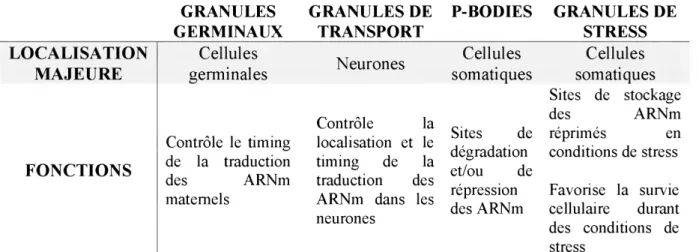
2.4. Lesgranulesà ARN ...26
2.4.1. Types de granules à ARN ... 27
2.4.2. Les granules de stress ...28
2.4.2.1. Formation des granules de stress ... 28
2.4.2.2. Composition des granules de stress ...30
2.4.2.3. Caractéristiques et dynamisme des granules de stress... 31
2.4.2.4. Rôle des granules de stress dans la survie cellulaire et la résistance des cellules cancéreuses 33 3. L'ARN HÉLICASEDDX3... 35
3.1. Structure...35
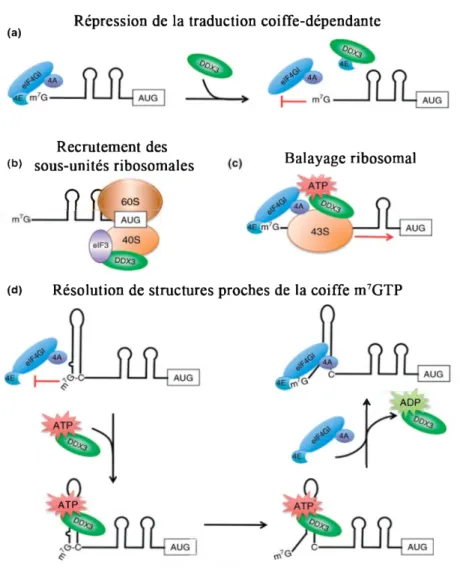
3.2. Fonctionsbiologiques de DDX3dans la traduction ...36
3.2.1. Rôle de DDX3 dans le développement du cancer ... 40
4. CONTEXTE ETOBJECTIFS ...43
CHAPITRE 1... 45
SORAFENIB, A MULTIKINASEINHIBITOR,INDUCES FORMATION OFSTRESSGRANULES IN HEPATOCARCINOMA CELLS... 45
1. AVANT-PROPOS...46
2. RÉSUMÉ ... 47
4. INTRODUCTION ...49
5. RESULTS ... 51
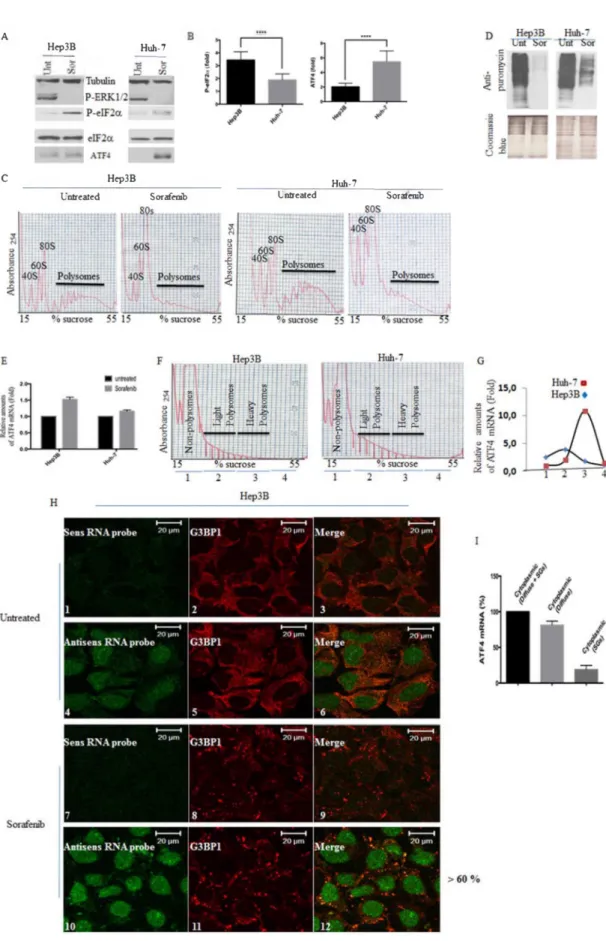
Sorafenib induces SGs formation in various cancercells including HCC ...51
Sorafenibinduces SGs in an eIF2aphosphorylation-dependentmanner...52
PERK is requiredfor eIF2aphosphorylation and associatedSGs formation in sorafenib-treatedHCC ...56
Role of PERK-SGs axis in sorafenib resistance through modulation ofATF4 expression...61
6. DISCUSSION...62
7. MATERIALS AND METHODS ... 68
Cell Lines and cultures...68
Drugs and drugstreatments...68
Antibodies...68
Small-interfering RNA (siRNA) experiments and DNAtransfection ... 68
Immunofluorescence, poly(A)+ in situhybridisationand RNAFISH ... 69
Quantitative RT-PCR...70
Polysomalprofiles andanalyses ofpolysomal-associatedRNA...71
[35S]Methionine labelingand ribopuromycylation ... 71
Annexin V-fluorescein isothiocyanate/propidiumiodide assay andclonogenicsurvival assays ... 71
8. ACKNOWLEDGMENTS ... 72 9. CONFLICTS OF INTEREST ... 72 10. GRANTSUPPORT ...72 11. ADDITIONAL INFORMATION... 72 12. REFERENCES... 73 13. SUPPLEMENTARY DATA ...78 CHAPITRE 2... 83
DDX3 REGULATES ENDOPLASMIC RETICULUMSTRESS-INDUCED ATF4 EXPRESSION 83 1. AVANT-PROPOS... 84
2. RÉSUMÉ ... 85
3. ABSTRACT ... 86
4. Introduction...87
5. RESULTS ... 89
DDX3 is involved inthe expression ofATF4 mRNA during ER stress ...89
DDX3promotesATF4expression atthe translationallevel ...93
DDX3 is a component ofthe eIF4F translationinitiationcomplexthat drives translation of the ATF4 mRNA uponER stress...97
6. Discussion...101
7. Material and Methods...104
Cellculture ... 104
Drugtreatments ... 104
Antibodies... 105
Immunofluorescence analyses ... 105
siRNAs and siRNAtransfection ...105
CapBinding assay ... 105
RNAimmunoprecipitation ... 106
Polyribosomal profiles and analyses of polyribosomal-associatedmRNA ... 106
QuantitativeReal-timePCRanalysis ...107
Clonogenic assays ...107 8. ADDITIONAL INFORMATION ... 108 9. ACKNOWLEDGMENTS ... 108 10. AUTHOR CONTRIBUTIONS ...109 11. COMPETING INTERESTS ... 109 12. REFERENCES... 109 13. SUPPLEMENTARY DATA ... 114 CHAPITRE 3... 117 DISCUSSION ET PERSPECTIVES ... 117
1. MÉCANISME ALTERNATIF DE RÉGULATION D'ATF4... 118
2. IMPLICATION DE DDX3 DANS LA TRADUCTION D'ATF4... 121
3. MÉCANISME DE RÉGULATION DE LA TRADUCTIONAVEC UORFS GÉNÉRALISÉ ? ... 125
4. LAVOIEPERK-EIF2a-ATF4 COMMECIBLE THÉRAPEUTIQUE ? ... 126
CONCLUSION ... 130
BIBLIOGRAPHIE ... 131
Liste des
figures
INTRODUCTION
Figure 1 : L'information codée par l'ADN est convertie en protéines par un processus à
plusieurs étapes ... 1
Figure 2 : Initiation de la traduction... 7
Figure 3 : Mécanismes de régulation de la traduction ... 9
Figure 4 : Les rôles des protéines de liaison à l'ARN... 13
Figure 5 : Les kinases phosphorylant la Serine 51 de la sous-unité a du facteur eIF2 ... 14
Figure 6 : Phosphorylation de eIF2a et réponse au stress...20
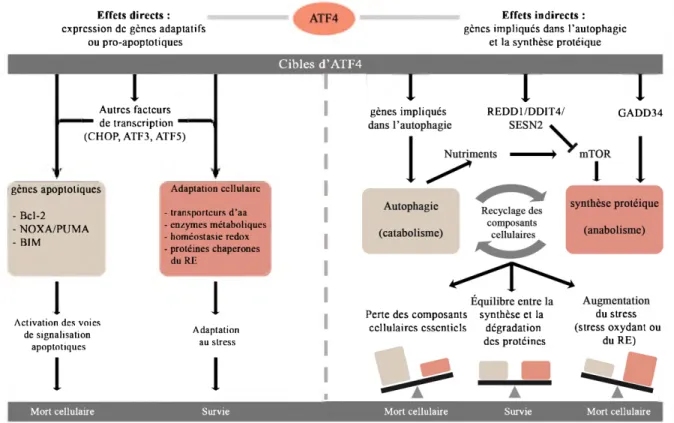
Figure 7 : Le contrôle d'ATF4 sur l'activation des voies de survie ou de mort cellulaire lors de stress...22
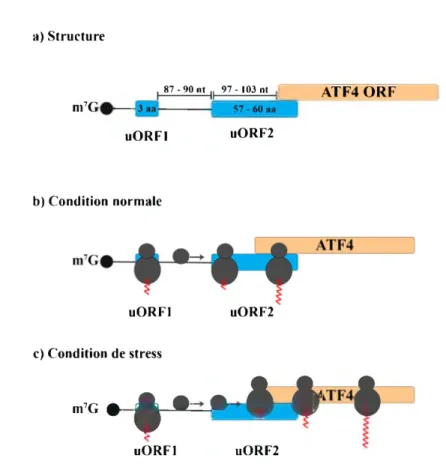
Figure 8 : Mécanisme de régulation de la traduction d'ATF4 ... 24
Figure 9 : Modèle d'assemblage des granules de stress... 32
Figure 10 : Représentation schématique de l'ARN hélicase DDX3 ... 36
Figure 11 : Différents modèles pour le rôle de DDX3 dans la traduction ... 37
Figure 12 : Quelques partenaires d'interaction de DDX3 ...38
CHAPITRE 1 Figure 13 : Sorafenib induces SGs in HCC ... 52
Figure 14 : Analysis of eIF2a phosphorylation, ATF4 mRNA expression and localization, and general translation ...54
Figure 15 : Sorafenib induces SGs in MEFs ...57
Figure 16 : PERK activation is required for sorafenib-induced SGs ...60
Figure 17 : The activation of PERK-P-eIF2a-SGs pathway correlates with HCC résistance to sorafenib... 62
Figure 18 : Model for the cross-talk between SGs and eIF2a phosphorylation in sorafenib- treated HCC ...67
Figure 19 : Sorafenib induces SGs in prostate, breast and cervix cancer cells ... 78
Figure 20 : Time course analysis of formation of SGs, phosphorylation of eIF2a, ATF4 expression and general translation in sorafenib-treated HCC... 79
Figure 21 : Sorafenib-induced SGs in HCC contain eIF4E and eIF4GI but lack the GFP- dcp1a, a classical P-bodies marker ... 80
Figure 22 : Sorafenib-induced SGs contain poly(A)+ mRNA ... 81
Figure 23 : Pharmacological inactivation of PERK reduces SGs formation in sorafenib- treated Hep3B ... 82
Figure 24 : Depletion of HRI does not affect SGs formation in sorafenib-treated Hep3B...82
CHAPITRE 2 Figure 25 : DDX3 is required for ER stress-induced ATF4 expression ...91
Figure 27 : eIF4GI is required for ATF4 expression upon stress ... 98
Figure 28 : DDX3 is a component of the eIF4F complex that is required for Sor-and Thap-induced ATF4 expression... 100
Figure 29 : Supplementary data 1 ... 114
Figure 30 : Supplementary data 2 ... 114
Figure 31 : Supplementary data 3 ... 115
Figure 32 : Supplementary data 4 ... 116
CHAPITRE 3 Figure 33 : Modèle de régulation de l'expression d'ATF4... 118
Liste des
tableaux
INTRODUCTION
Tableau 1 : Liste des facteurs impliqués dans l'initiation de la traduction... 5 Tableau 2 : Les différents types de granules à ARN ... 28
Liste des
abréviations
3'UTR 5'UTR 4E-BP ADN Ago1 Ago2 ALKBH5 ARN ARNdb ARNt ARNm ATF3 ATF4 ATF5 ATF6 ATF/CREB ATG5 ATP BiP bZIP CARE CBC Cdk CHOP CLIP-seq CPE CPEB CRM1 CUGBP1 DCP1 DCP2 DDIT4 dsRBD eEF2 eIF1Extrémité 3' non traduite
Extrémité 5' non traduite
eIF4E-bindingprotein Acide désoxyribonucléique Protein argonaute 1 Proteinargonaute 2 alkB homolog5 Acide ribonucléique ARN doublebrin
ARN detransfert
ARN messager
Activating transcription factor 3 Activating transcription factor 4 Activating transcription factor 5 Activating transcription factor 6
Activating transcription factor / cyclic AMP response element binding protein
Autophagy related5
Adénoside triphosphate
Cinding immunoglobulin protein
Casic leucine zipperprotein
C/EBP[CCAAT-enhancer-bindingprotein]- ATF [activating transcription
factor] - responsive element
Cap-bindingcomplex
Cyclin-dependent kinase
C/EBP Homology Protein
cross-linking immunoprecipitation combined with high-throughput sequencing
Cytoplasmic polyadenylation element
CPE-bindingprotein
chromosome region maintenance protein 1
CUG triplet repeats Binding Protein 1 mRNA-decappingenzyme 1
mRNA-decappingenzyme 2
DNA-damage-inducible transcript 4
Double-strandedRNA-bindingdomain
Eukaryotic elongation factor 2
eIF1A Eukaryoticinitiation factor 1A
eIF2 Eukaryoticinitiation factor 2
eIF2a Sous-unité a du facteur eIF2 eIF2B Eukaryoticinitiation factor 2B
eIF3 Eukaryoticinitiation factor 3
eIF4A Eukaryoticinitiation factor 4A
eIF4B Eukaryoticinitiation factor 4B
eIF4E Eukaryoticinitiation factor 4E
eIF4G Eukaryoticinitiation factor 4G
eIF4H Eukaryoticinitiation factor 4H
eIF5 Eukaryoticinitiation factor 5
eIF5B Eukaryoticinitiation factor 5B
ERAD ER-associated-degradation
FMRP Fragile X mental retardation protein
FRAP Fluorescence recovery after photobleaching
FXR1 Fragile X mental retardation syndrome-related protein 1
G3BP RasGTPase-activating protein-binding protein
GADD34 Growth arrest andDNA damage-inducible protein
GCN2 General Control Non-depressible-2
GEF Guanine-nucleotide exchange factor
GS Granule de stress
GDP Guanosine diphosphate
GTP Guanosine triphosphate
HCC Hepatocarcinoma cell / hépatocarcinome
HIF-1 Hypoxia-inducible factor 1
HisRS Domainehistidyl-ARNtsynthétase like
HO-1 Hemeoxygenase 1
HRI Heme-regulated inhibitor
IDR Intrinsically disordered regions
IRES Internal Ribosome Entry Site
ISR Integrated stress response / réponse intégrée au stress
ISRIB Integrated StressResponseInhibitor
JNK c-Jun N-terminal kinase
LLPS Liquid-Liquidphase separation
m6A N6-méthyladénosine
MAP1LC3B Microtubule-associated proteins 1A/1B lightchain 3B
MAPK Mitogen-activated protein kinase
Met-ARNti ARN de transfert initiateur-méthionine
miARN microARN
MTK1 aussi appeléMEK4, Mitogen-activated protein kinase kinase kinase 4
mTORC1 Mammalian/mechanistic target of rapamycin complex 1
mTORC2 Mammalian/mechanistic target of rapamycin complex2
mRNP Messenger ribonucleoprotein particle ORF Open reading frame
PABP Poly(A)-BindingProtein
PACT PKR-associatedprotein
P-Bodies Processingbodies
PERK PKR-likeERkinase
PI3K Phosphoinositide 3-kinase
PKR Protein kinasedouble-stranded RNA-dependent
PP1c Protein phosphatase 1 catalytic subunit
PTEN Phosphatase and tensin homolog
PUMA p53 upregulatedmodulatorofapoptosis
PUNCH Puromycin-associated Nascent Chain
REDD1 Regulated indevelopment andDNAdamage responses 1
Rac1 Ras-related C3 botulinum toxin substrate 1
RACK1 Receptor of activated protein C kinase 1 Raptor Regulatory-associated protein of mTOR
RE Réticulum endoplasmique
RISC RNA-induced silencing complex
RRM RNArecognitionmotif
S6K Ribosomalprotein S6 kinase
Ser51 Sérine 51
SESN2 Sestrin2
SHH Voie Sonic Hedgehog
shRNA Short hairpin RNA
SILAC Stable Isotope Labelling byAmino acids in Cell culture
siRNA Small interferenceRNA
SMN Survival of motor neuron
STAT Signal transducer and activatorof transcription
TAP Tip-associated protein
Thr Thréonine
TOP Terminal Oligopyrimidine
TRAF2 TNF receptor associated factor 2
TRAIL-R2 Tumor necrosis factor-related apoptosis-inducing ligand - receptor 2
uORF Upstream open reading frame
UPR unfolded protein response
UV Ultraviolet
TRB3 Tribbles 3
Remerciements
Je voudrais d'abordet avant tout remercier mon directeurde recherche, le Dr Rachid
Mazroui pour m'avoir accueillie en 2012 dans son laboratoire.Jelui suisreconnaissante pour
le support qu'il m'a apporté toutaucoursde mes années de doctorat et d'avoir cruen mes capacités. J'ai été honoréequ'il m'ait confiée d'excellentsprojets et je le remercie pourla
confiance qu'il m'a témoignée auseindeson laboratoire.Jetiens également à remercier mon
co-directeur, le Dr Edouard W. Khandjian pour sonaide et les conversationsscientifiques.
Jetiens ensuite à remercier laDre Barbara Papadopoulou, le Dr Imed Gallouzi etle
Dr. Samer Hussein pouravoir accepté d'évaluermathèse.
Je voudrais également remercier tous les membres présents et passésdu laboratoire
qui ontcontribués àmonbien-être tantscientifique quepersonnel. Jen'oublierais jamais nos
discutions pendant les pauses-café, qu'elles fussent scientifiques ou non-scientifiques. Un grand merci à Marie-Josée, Valérie, Bryan, Laetitia, Cristina, Aurélie, Claudia,Nassim et
Émeline. Merci Marie-Josée pour m'avoir parfaitement accueillie et formée dans le
laboratoire. Ta gentillesse, tonsoutienetton expérience ontété d'une grande aideaudébut de mon doctorat. Merci Valérie pour ton aide et nos discussions. Ce fut un réel plaisir de travailler avec toi ! Merci Laetitia pour ton aide lors de l'apprentissage de la technique
d'analyse des profilsde polysomes. Je remercieégalementlesstagiaireset notamment ceux
que j'aipu encadrer :France-Hélène,Frédérique, Sara, William etZora.
Je remercie très chaleureusementtousmes amis au Canada et en Europe qui m'ont énormément soutenue durant ces années de doctorat. Vous m'avez offert de très beaux moments,et également soutenue dans les moments plus difficiles. Je ne serai jamais allée
aussi loin sans vous! Je remercie tout d'abordles membres des différentslaboratoires qui ont croisé ma route. Merci Cécile, Halima, Karima, Antoine, Sébastien, Jonathan, Laurence,
Alexia,Surya et Justine. Merci à mes Gymmates: Clémence et Fanny.MerciClémence pout
tabonne humeur et pour nos discussions durant nos pauses-thé. Merci Fanny pour ton soutien
sansfaille. On a commencé àseconnaîtrequerécemmentmais ton soutiendurant la dernière
Europe, et plus particulièrement Anne-Laure, avec qui j'ai partagé tellement d'aventures
avant et pendant mon doctorat.Mercid'avoirété présents malgré ladistance.
Finalement, je remercie ma famille. Jesuisextrêmement reconnaissante envers mon
père et ma mère pour leurs encouragements et leur énorme supportenvers mes études. Merci
d'avoir cruen moi, merci tellement de m'encourager danstout ce que je faiset d'avoir été
toujours présents malgré la distance que nous sépare. A mon frère Aurélien,ma sœur Laure
Avant-propos
Les travaux présentés dans cette thèse sont le fruitde six années d'étudesausein du laboratoire du Dr Rachid Mazroui. Au cours de mon doctorat, j'ai contribué en tant que première auteure à laréalisationd'un article derevue de littérature, publié dansSeminarsin Cell & Developmental Biology et accessible en annexe 2, ainsi que de deux articles
scientifiques. La référence de la revue de littérature est la suivante : Adjibade Pauline,
Mazroui R. (2014) Control ofmRNA turnover : Implication of cytoplasmic RNAgranules.
Semin. Cell. Dev. Biol. fdoi: 10.1016/j.semcdb.2014.05.013.) J'ai bénéficié de l'aide de nombreux collègues au cours demes annéesde formation au doctorat qui ont rendus possible
la publication de mes travaux de recherche. Lacontribution des co-auteurs sera davantage
détaillée avant la présentationde chacun des manuscrits présentés auxchapitres 1 et 2.
Dans lechapitre 1, il sera question de mes travaux surla formation des granules de stress induite par l'agent chimiothérapeutique sorafenib au sein de cellules d'hépatocarcinomes, ainsi que de leur implication dans la régulation de l'expression du
facteur de transcription ATF4. Le manuscrit intitulé «Sorafenib, a multikinase inhibitor,
induces formation ofstress granules in hepatocarcinoma cells. » (Adjibade Pauline,Grenier
St-Sauveur Valérie,Quevillon-HuberdeauMiguel, Fournier Marie-Josée, SavardAndréanne,
Coudert Laetitia, Khandjian Édouard W., Mazroui Rachid) a été publié dans le journal
Oncotarget en novembre 2015. Une version PDF est disponible à l'annexe 3 de cette thèse.
Suite à ces résultats, nous avons investigué les mécanismes de régulation de la
traduction d'ATF4.Au cours de ces études, j'ai identifié pour la premièrefois uneprotéine
de liaison à l'ARNnommée DDX3, qui estimpliquée dans la traduction du facteurATF4.
J'ai égalementmontré l'implication des complexeseIF4E-eIF4GI danslatraduction d'ATF4.
Pour cet article, présenté dans le chapitre 2, j'ai réalisé une grande partie des résultats et
contribué àl'analyse des résultats ainsi qu'à la rédactiondu manuscrit. Lemanuscritintitulé « DDX3 regulates endoplasmic reticulum stress-induced ATF4 expression” (Adjibade Pauline, Grenier St-Sauveur Valérie, Bergeman Jonathan, Huot Marc-Etienne, Khandjian Edouard W., Mazroui Rachid)a été publié dans le journalScientificReportsenoctobre2017.
Lorsde mon arrivéedansl'équipe du Dr Mazroui, j'ai aussi contribué entant que
co-auteureà uneétudeportant sur l'étude du rôle des facteursd'initiation de la traductioneiF4E
et eiF4Gi, dans la formation des granules de stress induite par des stress modérés tels que celui induitparlebortezomib.cetravail décrit l'importancede la formation descomplexes
eiF4E-eiF4Gi dansl'assemblage des granules de stress et que cette formation estdépendante
dela voie de signalisation mToR.Danscet article, publié en 2013 dans le journalMolecular and Cellular Biology, j'ai participé à la réalisation des résultats montrant l'impact de la
drogue 4EGi,qui inhibe l'interaction entre eiF4E et eiF4G, dans la formation desgranules de stress.
J'ai également participé, en tant que co-auteure, à la rédaction d'une revue de littérature sur le rôle deHRIdans la résistance des cellules à l'apoptose, publiée dans le
journalMédecine Sciences en 2014 (Joncas FH, Adjibade P, Mazroui R. Rôle deHRI dans
la résistance des cellules à l'apoptose. Med. Sci. (Paris) 2014; 30(10):882-8; doi:
10.1051/medsci/20143010015). Pour cette revue, j'ai aidé et supervisé la rédaction de
France-Hélène Joncas, stagiaire aubaccalauréatau sein du laboratoire en2014.
En 2014,j'ai co-rédigé avec ma collègue Laetitia coudert une vidéo-publication du
protocole d'analyse de profils de polyribosomes au sein de cellules optimisé au sein du
laboratoire du Dr Mazroui. Elle a été publiée en mai 2014 dans le Journal of Visualized Experiments. Une versionPDF est disponibleà l'annexe 5 (Adjibade P, CoudertL, Mazroui
R. Analysis of translationinitiation by polysome profile analysis.J. Vis. Exp. 2014; (87); doi:
10.3791/51164). J'aiégalement réalisé, entantquepremièreauteure, un deuxième article sur
la technique des polysomes appliquée cette fois-ci à desxénogreffes et des biopsies. cette
publication, présentée en annexe 6, a été publiée dans Journal ofBiological Methods en
novembre 2016 (Adjibade P, Grenier St-Sauveur V, Droit A, Khandjian EW, Toren P,
Mazroui R. Analysis of thetranslatome in solidtumorsusing polyribosome profiling/RNA-
Seq. JBiolMethods 2016;3(4):e59; doi: 10.14440/jbm.2016.151). Par ailleurs, j'ai participé activement au développement de nouvelles techniques au sein du laboratoire, telles que la
mise en place de la technique PuNcH(Puromycinassociated Nascent chain) qui permettra
puromycine en condition de stress thérapeutiques au sein de cellules cancéreuses ou de
xénogreffeshumaines.
Tout au long de ma formation au doctorat, j'ai eu l'opportunité de participer à
plusieurs collaborations. J'ai tout d'abord participé à une étude portant sur la localisation d'ARN oxydés publiée dansJournal of Cell Science en novembre2015 (Zhan Y, Dhaliwal J, AdjibadeP, Uniacke J, Mazroui R,Zerges W. Localized control ofoxidizedRNA. J Cell Sci. 2015 Nov 15;128(22):4210-9. doi: 10.1242/jcs.175232). J'ai réalisé les
immunofluorescences permettantd'identifier un nouveau typede granules à ARN au sein de
cellules mammifèreset qui pourraient agir entantque sitesde stockageou de réparationdes ARN endommagés. Finalement, j'ai aidé et supervisé Claudia Marquez-Mota, étudiante au doctorat au «Instituto Nacional de Ciencias Médicas y Nutricion Salvador Zubiran
(INCMNSZ) » à Mexico etvenue au sein du laboratoire en 2015 afinderéaliser des analyses
de profils de polyribosomes sur des échantillons de foies de souris. Cette collaboration a
mené à la publication d'un article en 2016 dans le journal Nutrients (Marquez-Mota C,
Rodriguez-Gaytan CI, Adjibade P, Mazroui R, Galvez A, Tovar AR, Torres N. Hepatic
polyribosome profile and S6K-1 are enhanced after the recovery ofundernourished rats with a combination of soy or black bean with corn proteins. Nutrients, 2016 Sep 20;8(9). pii:
E573J. Étant donné que ces travaux divergentdu sujetprincipal demathèse, ces articles ne
Introduction
1.
Généralités
L'informationgénétiquequi détermine comment, quand etoùestproduit chaque type de
protéinesest codée pardes gènes auseindel'ADN. On compte entre25000 et 30000gènes
différents chez l'Homme. Les gènes qui portent l'information pour produireles protéines
contiennent généralement 2parties: une région codante qui spécifie la séquence en acides
aminés de la protéine et une région régulatrice (non codante) qui contrôle quand et dans quelles cellulesla protéine est produite. Afinde produire la protéine àpartir d'un cadre de lecture ouvert (ORF), les cellules utilisent 2processusmoléculaires (Figure 1).
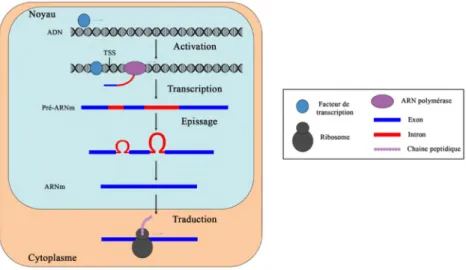
Figure 1: L'information codée par l'ADN est convertie en protéines par un processus àplusieursétapes
Dans un premier temps, les facteurs de transcription se lient aux régions régulatrices spécifiques des gènes spécifiques qu'ils contrôlent et les activent. A la suite de l'assemblage du complexe d'initiation multiprotéiques sur l'ADN, l'ARN polymérase commence la transcription du gène à partir du site d'initiation de la transcription (TSS). La polymérase se déplace le long de l'ADN produisant un transcrit simple brin (pré-ARNm) en utilisant un des brins d'ADN comme modèle. Le transcrit est par la suite épissé afin d'enlever les séquences non-codantes. L'ARNm mature obtenu est alors exporté dans le cytoplasme où il est lié par le ribosome qui va le traduire afin d'obtenir la protéine correspondante.
Dans un premiertemps, dansle noyaudela cellule, la région codanted'un gèneest copiée
afin d'obtenir un transcrit primaire (pré-ARNm) au cours d'un mécanisme appelé
transcription. L'ARN primaireest ensuitesoumis à de multiplesprocessus de maturation
(ajout de la coiffe en 5', polyadénylationen 3',épissageconstitutif et alternatif) afin d'obtenir
latraduction durant laquelle les ARNm sonttraduits en protéines. Une régulation précisede
ces mécanismes est cruciale afin d'assurerun bon fonctionnement cellulaire et permettreaux
cellules d'exprimer le bon répertoire génique nécessaire pour se diviser, se différencier et
s'adapter, notamment lors de conditions de stress cellulaire.
2.
La
réponse
cellulaire au stress
2.1. Reprogrammationdel'expression génique auniveautranscriptionnel et traductionnel
Les cellules sont continuellement sujettes à des stress tels que le stress oxydant,
l'hypoxie, lesinfections ou encore le stress induitpar des agents chimiothérapeutiques, qui
ont pour conséquence une modification de l'homéostasie cellulaire. En réponse aux
conditionsde stress,plusieurs situations sont possibles dépendammentdela nature dustress,
de son intensité ainsi que de sa durée. Soit la cellule est capable de répondre au stress en
mettant en place des mécanismes de défense lui permettant de s'adapter et de survivre au
stress, soit le stress est trop sévère et la cellule décide de se suicider (déclenchement de l'apoptose) afin de protéger les cellules environnantes et les fonctions physiologiques. La
mise en place de ces mécanismes de défense nécessite alors une reprogrammation de
l'expression génique tanttranscriptionnelle que traductionnelle.
Latraduction est un processus consommant énormément d'énergie. Lors de stress, les
cellulesréduisent la traductiongénérale, sauvantainsil'énergie nécessaire àla réparation des
dommages occasionnés par le stress (e.g. dommages de l'ADN etl'ARN, accumulation de
protéines mal repliées ou oxydation des protéines).Ainsil'inhibition dela traductionglobale
est l'une despremières réponses observables d'adaptation au stress. Toutefois laréduction
de la traduction généralefavorisela traduction prioritaire d'ARNm spécifiques codant pour des protéines impliquées dans la réponse au stress, nécessaire à l'adaptation rapide des
cellulesàleur nouvel environnement et àleur survie.
Dans le cadre de ma thèse, j'ai étudié plus particulièrement le mécanisme de
régulation de latraduction de l'ARNm d'une de ces protéinesspécifiquement traduites lors
2.2. La traduction
La plupart des ARNm contiennent une structure coiffe (m7GpppN) au niveau de leur
extrémité 5'. En générale, la traduction des ARNm s'initie par la reconnaissance de la
structure coiffe par les complexes d'initiation de latraduction. On parle alors detraduction
coiffe dépendante (cap-dependanttranslation). Ce type de traduction peut être modulé soit
par l'accessibilité des protéines de reconnaissance de la coiffe (e.g. eIF4E) ou par des modifications post-transcriptionnelles des ARNm (e.g. méthylation des adénosines), ou
encore par des ARN régulateurs qui agissent en trans comme lesmiRNAs. Certains ARNm peuvent cependant être traduits indépendamment de la coiffe. Ce type de traduction (cap-
independant translation) nécessitent la reconnaissance de séquences ARN dans la région
5'UTR, appelés IRES (de l'anglais Internal Ribosome Entry Site), ainsi que le recrutement
interne duribosome et du complexe d'initiation au niveau de cesséquences 1. Cette traduction
s'opère essentiellement en conditions destress qui inhibent la traduction coiffe-dépendante.
D'autres séquences ARN telles que les upstream ORFs (uORFs) situées au niveau de la
région 5'UTR des ARNm régulentla traduction coiffe-dépendante en condition de stress2-7 Dans le cadre de cette thèse,jem'intéresseessentiellement à latraduction coiffe-dépendante et sa régulation lors de stress.
2.2.1. Généralités
La traductionnécessite un assemblageordonné des ribosomes avec les facteurs de la
traduction et les ARN de transfert (ARNt) (Tableau 1) et peut être divisée en 3 étapes
principales : l'initiation, l'élongation et la terminaison. L'étape d'initiation correspond au
recrutement du ribosome au niveau de la région 5'UTR de l'ARNm suivi de la
reconnaissance du codon d'initiation. Durant l'élongation, le ribosome lie la séquence
codante de l'ARNm permettant la synthèse de la protéine par ajout d'acides aminés à la chaine peptidique en coursde synthèse. Enfin, laterminaison se produitlorsquele ribosome arrive au niveau d'un codon stop et se détache de l'ARNm. Cela permet alorslalibérationde la chaine protéique dans le cytoplasme de la cellule et le recyclage des sous-unités ribosomales. Laprotéineproduitepeut ensuite subir des modifications post-traductionnelles
(ex: phosphorylation, méthylation, glycosylation ou encore sumoylation)quivont réguler sa conformation, sa stabilité, sa localisation ouencore son activité.
2.2.2. Régulationde la traduction
La traduction est un évènementhautement régulé impliquant des protéines deliaison
à l'ARN ainsi que l'ARN lui-même. Les mécanismes derégulationdela traduction affectent
principalementl'étape d'initiation, qui représente l'étapelimitante dela synthèse protéique.
Toutefois, des études récentes montrentqu'il existe également des mécanismes derégulation
lors des étapes d'élongation 6,8 ou de terminaison 9. Parmi les mécanismes de régulation de
l'élongation de latraduction, on peut citer la phosphorylation du facteur eEF2 (Eukaryotic
elongation factor) durant des chocs thermiques (cold shock), qui entraine l'inhibition de la
translocation du ribosome le long de l'ARN 8,10.
Par ailleurs, la régulation de la traduction est un processus très réactif : dès l'apparition d'un signal physiologique indiquant un besoin, la protéine peut être immédiatement traduite ou inversement. Cette réactivité est particulièrement importante lorsque les cellulessont exposéesàdivers stress et doivents'adapter aux nouvelles conditions
afin de survivre. Les différents mécanismes de régulation de l'initiation de la traduction
serontdéveloppés un peuplustard.
2.2.3. Mécanisme del'initiationde la traduction
Le mécanisme d'initiation de la traduction coiffe dépendante implique la reconnaissanceet le recrutementde l'ARN par la machinerie de l'initiation de latraduction
et l'assemblage du ribosome 80S au niveau de l'ARNm. Ce processus implique l'activité d'au moinsune dizainedefacteursappelés facteurs d'initiation(Tableau 1) et peut être divisé
en plusieurs trois étapes : a) formation du complexeternaire (TC); b) association du TC avec
la sous-unité ribosomale 40S donnant formation au complexe de pré-initiation 43S; c)
recrutement du complexe 43S à l'ARNm pour former le complexe de pré-initiation 48S
positionné au niveau du codon d'initiation;d) recrutement de la sousunité ribosomale 60S au niveau du complexe 48S pourassembler le monosome 80S.
Nom Partenaires d'interaction
Fonction
Facteurs principaux
eIF1 eIF2, eIF3, eIF5, sous-unitéribosomale 40S
Assure la fidélité de la sélection du codon initiateur; coopère avec eIF1A afin de promouvoir le balayage ribosomal et la reconnaissance ducodon initiateur
eIF1A eIF5B, sous-unité ribosomale 40S
Stimule la liaison ducomplexe ternaire aveclasous-unité 40S; coopère avec eIF1 afinde promouvoir le balayage ribosomal et lareconnaissance du codoninitiateur
eIF2 eIF1, eIF2B, eIF3, eIF5, Met-ARNti, sous-unité
ribosomale 40S
Forme le complexe ternaire eIF2-GTP-Met-ARNti qui lie la
sous-unité 40S; régule le recrutement de Met-ARNti au ribosome
eIF2B eIF2 Protéine GEF (Guanine nucleotide Exchange Factor) qui catalyse l'échange de GDP/GTP sur eIF2
eIF3 eIF1, eIF1A, eIF4G,
eIF5, sous-unité
ribosomale 40S
Stimule la liaison de eIF1, eIF4G et eIF5 à la sous-unité ribosomale 40S; stimule la liaisondu complexe ternaire à la sous-unité ribosomale 40S.
eIF4A eIF4G, eIF3,ARN ARN hélicase, dépendantede l'ATP, de lafamille des protéines DEAD box, déroule les structures secondairesprésentessurles ARNm
eIF4E eIF4G, coiffe des ARNm Reconnaîtet liela structure coiffe m7GpppG à l'extrémité 5'
des ARNm
eIF4G eIF4E, eIF4A, eIF3,
PABP, ARN
Protéine d'échafaudagequi permetla liaison de eIF4E,eIF4A, eIF3, PAPB etl'ARNm ;stimule l'activité hélicase deeIF4A
eIF4F Complexe composé de eIF4E, eIF4A et eIF4G, déroule la région proximale 5' del'ARNm, régule la liaisonducomplexe 43S à l'ARNm et assiste les complexes ribosomaux lors du balayage de l'ARNm
eIF4B eIF4A, eIF3,ARN Protéine de liaison à l'ARN quistimulel'activité hélicase de eIF4A
eIF5 eIF1, eIF1A,eIF2, eIF3, sous-unité ribosomale 40S
Protéine GAP (GTPase-activating protein), lié spécifiquement à eIF2-GTP et hydrolyse le GTP de eIF2-GTP lors de la reconnaissance ducodon initiateur
eIF5B eIF1A, ribosome 80S GTPase dépendanteduribosome, impliquée dans l'association de la grande sous-unité ribosomale avec lapetite sous-unité ribosomale.
Facteurs supplémentaires
eIF6 Sous-unité ribosomale
60S
Facteur qui empêche l'assemblement de la sous-unité ribosomale 40S avecla sous-unité 60S
DDX3 eIF4E,eIF4G, ARN ARN hélicase, dela famille des protéines DEAD box, interagit aveceIF4Eet eIF4G
DHX29 eIF3, sous-unité ribosomale 40S,ARN
ARN hélicase, dela famille des protéines DEAD box, promeut le balayage ribosomal sur les ARNm possédant de longues extrémités5'très structurées
PABP eIF4G, ARN Lie la queue poly-(A) des ARNm,interagit avec
eIF4G et eRF3, favorise laliaisondu complexe eIF4F
sur la coiffe
Tableau 1:Listedes facteurs impliqués dans l'initiation delatraduction
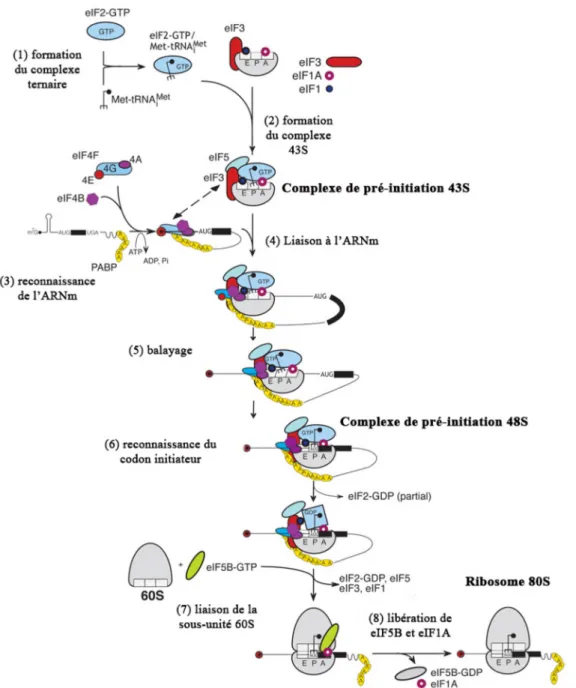
2.2.3.1. Formation du complexe ternaire etdu complexe depré-initiation43S
La première phase de l'initiation de la traduction commence par la formation du
complexeternaire(TC). Dans lacellule, le facteur d'initiation eIF2 existe sous deux formes.
Sous sa formeinactive, eIF2estassociée à unemolécule deGDPet nepeut pas lier l'ARN de transfert initiateur-méthionine (Met-ARNti). Le facteur eIF2B catalyse le remplacement
de la molécule de GDP par une molécule de GTP au niveau du facteur eIF2 permettant à celui-ci de se lier avec Met-ARNti formant ainsi le TC 11. Le TC formé s'associe avec la
sous-unité ribosomale 40S grâce aux facteurs derecrutement eIF1, eIF1A, eIF5eteIF3. Ceci
mèneàla formation ducomplexe de pré-initiation 43S (Figure 2, étapes 1 & 2).
2.2.3.2. Formation du complexe de pré-initiation 48S et du monosome80S
Le recrutement du complexe de pré-initiation 43S à l'ARNm est assuré par le
complexe eIF4F. Cecomplexe est composé des facteurs d'initiation eIF4E,eIF4GI et eIF4A.
Alorsque eIF4E se lie spécifiquement sur la structure coiffe (m7GpppN) présente en 5' de
l'ARNm 12, son partenaire de liaison eIF4GI s'associe au facteur eIF3 permettant le
recrutement du complexe de pré-initiation 43S au niveau de l'ARNm 13. eIF4GIinteragit
égalementavecla protéine PABP (Poly(A)-BindingProtein), qui selie à la queue polyA de
l'extrémité3'de l'ARNm, permettantainsi la circularisation del'ARNmnécessairepourune
traduction efficace.Le facteur eIF4GI possède également2 sites de liaison à eIF4A dans sa
partie carboxy-terminale,assurant ainsila cohésion du complexe eIF4F. Grâce à sonactivité
hélicase et avec l'aide des facteurs eIF4B et eIF4H, le facteureIF4A déroule les structures
secondaires del'ARNm se trouvant près delacoiffe dans la région 5'nontraduite facilitant
ainsi le balayage de cette région par le complexe 43S jusqu'au codon d'initiation de la
traduction AUG formant ainsi le complexe de pré-initiation 48S 14-16 (Figure 2, étapes 3 à
6). Les facteurs eIF1 et eIF1A jouent un rôle important dans la reconnaissance du codon
initiateur 17,18. La reconnaissance du codon d'initiation induit un changement conformationnel du complexe 48S l'empêchantde continuer son passage le long del'ARNm.
A ce stade, le facteur eIF5 hydrolyse le GTP d'eIF2 en GDP, ayant pour conséquence la
elF3 elF2-GTP/ J*Met-tRNA,Met elF5 (5)balayage Complexe de pré-initiation 48S elF3 4 elFIAO elF1 • (1) formation du complexe ternaire (6)reconnaissance du codon initiateur (2) formation du complexe 43S E P A y ) elF2-GDP (partial) GTr) “ } Complexe de pré-initiation 43S elF3 ' (4)Liaison à l’ARNm elF4F_.A 4EW elF4B>-'J
.JL
elF2-GTPCE} ™,r,
_
Met-tRNA^1 .i.
—*
c
Ribosome 80S elF2-GDP, elF5 ’elF3, elF1 (8) libération de eIF5B et elFlA X > -elF5B-GDP O elF1A elF5B-GTPFigure 2 : Initiation de la traduction
L'initiation de la traduction commence par la formation du complexe ternaire qui consiste en l'association du facteur eIF2- GTP avec le Met-ARNt initiation (1). Le complexe va ensuite interagir avec la sous-unité ribosomale 40S grâce aux facteurs eIF1, eIF1A, eIF5 et eIF3 afin de former le complexe de pré-initiation 43S (2). En parallèle, le complexe eIF4F, composé de eIF4E, eIF4G et eIF4A, reconnait l'ARNm (3). Le complexe 43S est ensuite recruté via le complexe eIF4F au niveau de l'extrémité 5' de l'ARNm pour former le complexe de pré-initiation 48S (4). Le complexe 48S balaye ensuite la région 5' non traduite à la recherche du codon initiateur (5). Une fois le codon initiateur reconnu, le complexe de pré-initiation 48S est dissocié et les facteurs d'initiation sont recyclés (6). La sous-unité ribosomale 60S se lie alors à la sous-unité 40S, avec l'aide du facteur eIF5B, pour former le ribosome 80S, terminant ainsi l'initiation de la traduction (7, 8). Adapté de Jackson et al., 2010, Nature Review Mol Cell Biol.
La sous-unité ribosomale 60S est alors recrutée, avecl'aidedufacteureIF5B, pourformer le monosome80S, terminantainsil'étape d'initiation de la traduction20. L'élongationpeut alors
commencer(Figure2, étapes 6à 8).
Dans le cadredemestravauxderecherche, je mesuis intéressée plusparticulièrement
à l'implication des complexes eIF4F dans la formation des granules des stress ainsi que la régulation dela traductiondecertains ARNm, dont celui du facteurATF4, lorsdeconditions de stress.
2.3. Régulation de l'initiation de la traduction en condition destress
La régulation de la traduction joue un rôle important puisqu'il s'agit du processus
majeur régulant le niveau d'expression des protéines. Les cellules régulent leur traduction
au cours de différentes conditions physiologiques (ex : cycle cellulaire, différentiation des
cellulesT)mais aussi lorsde nombreuses conditions de stress tellesque le stress oxydant, les
chocs thermiques, la privation en nutriments, les radiations ionisantes ou encore les
traitements chimiothérapeutiques 21. Dans de telles conditions, on assiste à une
reprogrammation traductionnelle majeure qui se produit essentiellement au niveau de la
phase d'initiationde la traduction.
2.3.1.Mécanismes derégulation de l'initiationde la traduction
La régulation de l‘initiation de la traduction implique l'activation de voies de signalisation traductionnelles ciblant soitdirectement les facteurs d'initiation delatraduction
soitdirectement les ARNm (via des protéines deliaisonà l'ARN ou des miARNs) 22 (Figure
Figure 3 : Mécanismesderégulationde la traduction
La régulation de la traduction des ARNm se produit à différents niveaux. Une régulation globale de la traduction se produit via des modifications des facteurs de la traduction telles que la phosphorylation du facteur eIF2a par la kinase eIF2a (PERK, HRI, GCN2 et PKR) ou la modulation des affinités des partenaires de liaison des facteurs d'initiation de la traduction comme la phosphorylation par le complexe mTORC1 du partenaire de liaison d'eIF4E, 4E-BP. La reconnaissance par des protéines de liaison à l'ARN de séquences spécifiques ou de structures secondaires dans les régions 5' et 3' non traduites mène à l'activation ou à la répression de la traduction. L'inhibition de la traduction par les miARNs peut se produire via l'inhibition de l'initiation ou de l'élongation de la traduction. La traduction de certains ARNm est régulée via la présence de courts cadres de lectures ouverts dans la région 5' non traduite appelés upstream Open Reading Frame (uORF). La traduction peut également être initiée indépendamment de la coiffe des ARNm via des séquences spécifiques appelées IRES présentes dans la région 5' non traduite. RBP : protéine de liaison à l'ARN ; miARN : microARN ; 5'UTR : région 5' non traduite ; 3'UTR : région 3' non traduite ; IRES : Internal ribosome entry site ; uORF : upstream ORF
o Contrôle dela traduction via le processus de reconnaissance delacoiffe des ARNm
Un premier mécanisme queles cellules eucaryotes utilisent afinde contrôler letaux
de traduction implique la reconnaissance de la coiffe en 5' des ARNm par eIF4E dont
l'activité est contrôlée par la voie de signalisation mTOR (mammalian Target Of
Rapamycin). mTOR est unesérine/thréonine kinase qui forme deuxcomplexes moléculaires
distincts: mTORC1 et mTORC2. C'est principalement le complexe mTORC1, dont
l'activité est stimuléepar l'insuline, les facteurs de croissance, le sérum ou encore les acides
aminés(etplusparticulièrement la leucine),qui régulelatraduction23.En effet, l'activité du
facteur eIF4Eest principalement régulée par des membres de la famille des eIF4E-binding
(4E-BP1, 4E-BP2, et 4E-BP3) partagent un site de liaison à eiF4E commun avec eiF4Gi, se
disputant ainsi la liaison avec eiF4E 24. La liaison de 4E-BP1 avec eiF4E est réguléepar la phosphorylation de 4E-BP1 via la kinase mToR25. Lorsque mToR est inactive, la protéine 4E-BP1 est hypophosphorylée. sous cette forme, 4E-BP1 possède une forte affinité pour eiF4E empêchantl'interaction de eiF4E avec eiF4G inhibant ainsi la liaison du complexe
eiF4F à la structure coiffe de l'ARNm et empêchant sa traduction. A l'inverse, l'activation
de la kinase mToR permet la phosphorylation de 4E-BP1, affaiblissant son affinité pour eiF4E et permettantainsi sa dissociation.Le facteureiF4E est ainsi libre dese lier au facteur eiF4G permettantle recrutement du complexe eiF4F auniveaudel'ARNm26.
A noter que mToR est aussi responsable, directement ou indirectement, de la
phosphorylation d'autres facteurs impliqués dans la traduction tels que eiF3, eiF4G ou la kinase S6K 25,27-29. Ces fonctions de mTOR permettent un autre niveau de régulation de la
traduction. En effet, l'activation de mToR favorise l'association entre les facteurs eiF3 et
eiF4G permettant au complexe de pré-initiation43s et au complexeeiF4F d'interagir entre
eux et de lier l'ARNm à la sous-unité 40S 27 De plus, la phosphorylation de S6K permet
d'activer l'élongation dela traduction mais entraîne également un rétrocontrôle négatifde la
voiePI3K/Akt empêchant ainsi toute sur-activation de la traduction coiffe-dépendante 29.
o Contrôle dela traduction par les miARNs
Les microARNs (miARNs) sont des ARN non codants, de petite taille (20 à 23
nucléotides), capables demoduler l'expression des gènes de façon post-transcriptionnelle. Il
est estimé que plus de 60% des gènes humains codant pour des protéines possèdent des
séquences cibles de miARNs dansleur extrémité 3'UTR 30 . La synthèse des miARNs débute
par la transcription d'un long précurseur primaire, appelé “Pri-miARN”. Le clivage de ce
transcrit primaire par laribonucléaseDrosha donne naissance au “pré-miARN”, structure en
épingle à cheveux comprenant 70 à 100 nucléotides qui est exporté vers lecytoplasme. Ce
pré-miARN est alors pris enchargepar la ribonucléase Dicer qui interagit avec les protéines
dela famille Argonaute, afin de générer un duplex de courts brins d'ARN correspondant au
miARNmature. un des brinsdu miARNs'incorpore ensuite dans le complexe RIsC
hybridationimparfaiteavec son ARNm cible, le miARN déstabilise l'ARNm et/ouinhibe sa
traduction 31,32. Unemajorité des sites de liaisondes miARNs caractérisés est située dans la
région 3'UTR des ARNm. Par exemple, uneétude de Laletal. montre que le miARN miR-
24 cible l'ARNm de p16, ARNm qui code pour un inhibiteurde kinases cycline-dépendant,
et réprime sa traduction dans des fibroblastes et des cellules de carcinomes cervicaux 33.
Cependant, plusieurs études montrent que des miARNs peuvent réguler latraduction en se
fixantdansla région 5'UTRouau niveau de la région codante deleurs ARNm cibles 34,35. Il
a été suggéré que les miARNs peuvent réprimer l'initiation de la traduction en interférant
probablementavec les fonctions de liaison à la coiffe d'eIF4E 36,37.Ilanotamment été montré
in vitro que la reconnaissance de la coiffe des ARNm estréprimée par le miARN let-7 au
seind'extraits acellulaires de souris 38. D'autresétudes décrivent également des interactions
entre la machinerie des miARNs et les facteurs d'initiation de la traduction. Chez la
Drosophile, il a été montré que Ago1-RISC induit la dissociation du facteur eIF4A de
l'ARNm cible bloquant ainsi l'assemblage du complexe eIF4F et réprimant latraduction39.
La protéine argonaute AGO2 a également le potentiel d'interagir directement avec la
structure coiffe des ARNm et réprimer ainsi l'initiation de la traduction en entrant en
compétition avec eIF4E pourla liaison àlacoiffe40.
À l'inverse, dans certaines conditions (telles que l'arrêt du cycle cellulaire), les
miARNs peuvent stimuler la traduction de certain ARNm. Dans la plupart des cas,
l'activationdelatraductiond'un ARNm par un miARN estassociée à lafixationdece dernier
dansla région 5'UTRde l'ARNm 41-43. Par exemple, Orom et al. ont montré que le miARN
miR-10a se fixe en aval d'une séquence oligopyrimidique, appelée 5'TOP , présente dans la
région 5' non traduite des ARNm codant pour des protéines ribosomales et de certains
facteursde l'appareil detraductionetaugmente leurtraduction43.
o Contrôle dela traduction par les protéines de liaison àl'ARN
Un autre mécanisme de régulation de la traduction implique une combinaison de protéines de liaison à l'ARN qui reconnaissent des motifs régulateurs ou des structures
secondaires sur les ARN. La famille de protéines de liaison à l'ARN compte plus de 1500
domaines les plusconservésau sein des protéines de liaison à l'ARN incluentnotammentles
domaines riches en glycine (RGG), les domaines de motifs de reconnaissance à l'ARN
(RRM, de l'anglais « RNA recognition motif ») et les domaines KH (pour hnRNP K
homology domain). Le domaine RGG est important pour l'agrégation réversible des protéines tandis que le domaine RRM est importantpourla régulation de laliaison à l'ARN,
bien que ce domaine puisse aussi avoir un impact surl'agrégation des protéinesde liaison à
l'ARN. Certaines protéines de liaison à l'ARN possèdent également des domaines
fonctionnels additionnels qui leur confèrentdes activités spécifiques ou contribuent à leur
spécificité de liaison à des ARNm spécifiques. Le domaine fonctionnel hélicase appelé
DExD/H estun exemple de domaine effecteur important. On leretrouvedans de nombreuses protéines impliquées notamment dans la régulation de l'initiation de la traduction, dont l'ARN hélicase DDX3 46. Les protéines de liaison à l'ARN interagissent de manière dynamique avec des ARNm ou des ARN non codants au sein des complexes
ribonucléoprotéiques (mRNP). Ces complexes mRNPs vont alors diriger le devenir des
ARNm soiten les traduisant, soiten les dégradant ou en les stockantsousformeréprimée au
sein des granules à ARN 47,48. Dans le cytoplasme, grâce à leurs interactions dynamiques
avec les ARN, les protéines de liaison à l'ARN régulent le transport et la localisation des
ARNm ainsi que la répression, la traduction ou encore la stabilité des ARNm47-50 (Figure 4). De plus, les protéines deliaisonà l'ARN participent à l'inhibition de lasynthèse protéique
globaleen séquestrant les ARNm dans lesgranules àARNtout en stimulantsimultanément
la traduction d'ARNm spécifiques impliqués dans la réponse au stress 51. Ainsi, des
interactions spécifiques protéines-ARN dans les régions 5' ou 3' non traduites peuvent
régulerlatraduction desARNm cibles. Par exemple, la liaison spécifique d'une protéinede
liaison à l'ARN en 3'UTR peut entrainerla formationd'une boucleferméequiinhibepar la suite latraduction. C'est notammentle cas des ARNm contenant des séquences CPE (pour
« cytoplasmic polyadenylation element » en anglais). Ces séquences sont notamment présentes sur des ARNm dont la traduction est contrôlée au cours du développement
embryonnaire. Avant la maturation méiotique des ovocytes de Xenopus, la protéine
Maskine/4E-T est recrutée au niveau de l'ARNm par la protéine CPEB (CPE-binding
protein). Maskine réprime alors la traduction en se liant à eIF4E, empêchant ainsi la formation decomplexeeIF4E-eIF4G52.
Épissage
Polyadénvlation
Ajout de la coiffe (m7G)à l’extrémité 5’ des ARNm
Export ARNm vers cytoplasme Localisation ( granules de stress ou P-Bodies)
/
F
\
Modificationschimiques desARNm ( mlA, m5C, m6A,*P) TraductionStabilité des ARNm ( stabilisation ou dégradation) Figure 4 : Les rôles des protéines de liaison à l'ARN
Les protéines de liaison à ARN sont notamment impliquées dans la régulation de l'épissage et l'export des ARN, dans la régulation des modifications chimiques des ARN (polyadenylation, ajout de la coiffe) ainsi que dans la localisation, la traduction et la stabilité des ARNm. m7G : 7-methylguanosine ; m1A: .V'-methvladenosine ; m5C : 5-methylcytosine ; m6A
: jV’-methyladenosine ; Y : pseudouridine
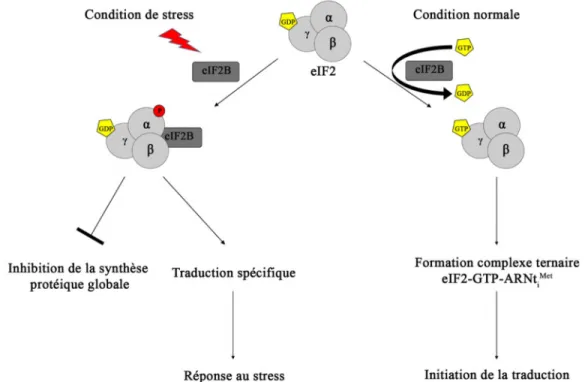
o Contrôle dela traduction via la phosphorylationd’eIF2a et des uORFs
La phosphorylation du facteur eIF2a est le mécanisme majeurinhibant l’initiation de la
traduction en condition défavorable de croissance cellulaire engendrantun stress cellulaire
53. La phosphorylation du facteur eIF2 se produit au niveau de la Serine 51 (Ser51) de sa
sous-unité a, entrainant une diminution dela formation ducomplexe ternaire compétent pour
la traduction. Bienqu'inhibantlatraduction globale, la phosphorylation de eIF2a permet la
traduction spécifique decertainsARNm contenant entreautres des upstream ORFs (uORFs).
Cette voiede régulation est développéeplus endétails dansla section « 2.3.3. Régulation de
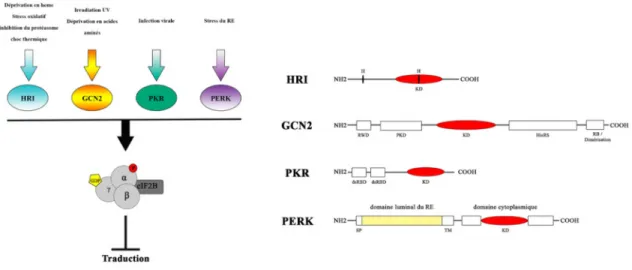
2.3.2. Les kinases eIF2a (HRI, GCN2, PERK etPKR)
Chez les eucaryotes,la phosphorylation du facteur d'initiation dela traduction eIF2a
au niveau de la Ser51 se produit via quatre sérine/thréonine kinases : HRI, GCN2, PKR et
PERK 21 (Figure 5). Ces quatre kinases partagent un domaine kinase conservé mais elles
possèdent des domaines régulateursdifférents (Figure 5) 21. L'activation de ces 4 kinases se
produit vialeur dimérisation et autophosphorylation. Cependant, chacune est activée par des
stress physiologiques ou environnementaux distincts(Figure 5) 21.
Figure 5 : Les kinases phosphorylant la Serine 51de la sous-unité a du facteur eIF2
KD : domaine kinase, H : liaison de l'hème, PKD : domaine pseudokinase, RB : domaine de liaison au ribosome, dsRBD : domaine de liaison à l'ARN double brin, SP : peptide signal, TM : domaine transmembranaire.
o La kinase HRI
La kinase HRI (Heme-regulated inhibitor) est principalement exprimée dans les
érythrocytes et estimpliquée dansla différenciation des érythrocytes pendant l'érythropoïèse
54,55. Laprotéine HRI est composée de 5 domaines dont deux sites de liaison à l'hème, un
dans la partie N-terminaleet le second est inclus dans son domaine kinase(Figure 5) 54,56,57.
Le site de liaison à l'hème présent en N-terminal est celui responsable de la régulation de
HRIparl'hème. En effet, lorsque HRI est lié par l'hème au niveau de cette région, son activité
surde multiples sitesinduisantlaphosphorylation d'eIF2a. S'en suit une inhibition globale dela traduction et notamment celle des a et B-globines qui constituent les protéines majeures
des globules rouge. Cette régulation de la synthèse des globines permet de maintenir une
homéostasie optimale entre les globines et l'hème pour la formation d'hémoglobine. En
régulant ainsi l'équilibre hème-globine, HRI joue un rôle anticytotoxique en condition de
déficience en hème. D'autres études ont également montré que la kinase HRI est activée lors
d'une réponse austress oxydant 58 ou d'inhibition du protéasome59,60, permettantaux cellules
de s'adapter aux nouvellesconditions cellulaireset de résister au stress.
o La kinaseGCN2
Lakinase GCN2 (General Control Non-depressible-2) est généralement activée lors
d'une carenceen acides aminés61.GCN2 estégalement activée par une carence en glucose
62,63 ou encore une exposition à defortes doses d'UV (minimum 50-60 J/m2)64,65. De plusil
a également été montré que GCN2 est activée et induit la phosphorylationd'eIF2a lors de
conditions hypoxiques 66,67. GCN2 est constituée de cinq domaines: un domaine kinase
typique, un domaine pseudokinase, un domaine histidyl-ARNt synthétase like (HisRS) qui
lie les ARNt non chargés, un domaine RWD de liaison au complexe GCN1/GCN20 en
N-terminaletun domainede liaison au ribosome et de dimérisation en C-terminal 21(Figure5).
Lors d'une carence en acides aminés, les ARNt non chargés s'accumulent et se lient au
domaine HisRS de GCN2 induisant un changement de confirmation de la kinase et son
activation. Chez la levure, la carence en glucose entraine l'activation GCN2 de manière
dépendante de son domaine HisRS, indiquant qu'une accumulation d'ARNt non chargés
contribue à son activation 62. L'activation de GNC2 va alors induire la phosphorylation d'eIF2a etune inhibition delasynthèse protéique globale. En parallèle, grâce àune structure
particulière de sa région 5' non traduite, la traduction de l'ARNm du facteur transcription
ATF4 est augmentée. Ce mécanisme detraduction seradiscutéplusendétails danslasection « 2.3.4.2 Le facteur de transcription ATF4». L'expression de ce facteur de transcription
induit alors la transcription de certains gènes cibles impliqués dans l'adaptation au stress
nutritionnel tels queceux impliqués danslabiosynthèse des acides aminés ou l'homéostasie
du glucose. A noterque la carence en acidesaminés ou en glucose ainsi que l'hypoxie sont
cellules cancéreuses. Cela suggère que GCN2 peut participer à la survie des cellules
tumorales. Plusieurs études ont montré l'implication de GCN2 dans la progression des
cancers. Il a été observé quela kinase GCN2 estexprimée à unniveau élevé dans certaines
tumeurs68. De plus,les travaux de l'équipe du Dr. Koumenis ont montré queGCN2 et ATF4
étaient nécessaires àla survie et lacroissancetumorale. En effet, ils ontobservéque l'activité de GCN2 au sein detumeurs humaines etmurines est augmentée alors que la déplétion de
GCN2 ralentit significativementla croissance tumoralein vivo 63. Cette activationde GCN2
est cohérente avec la nécessité pour la tumeur d'augmenter l'approvisionnementenéléments
nutritifs, tels que lesacidesaminés.
o La kinase PKR
La protéine PKR(proteinkinase double-stranded RNA-dependent) estune kinasequi
est principalement activée lors d'infection virale 69. PKR est la kinase eIF2a la mieux
caractérisée. Elle est principalement localisée dans le cytoplasme. Mais dans certaines
conditionsde stress cellulaire, PKR est transloquéeau niveau du noyau. PKR se compose de
2 domaines principaux : une région N-terminale régulatrice qui contient un domaine de
liaison aux ARN doublesbrins (dsRBD/ARNdb) composé de 2 motifs d'environ 70acides
aminés séparés par une région linker et une région C-terminale composée du domaine kinase
70 (Figure 5). Lors d'une infection virale, l'ARN double brin (ARNdb),crée parlaréplication
virale, liela régionN-terminale au niveau de son domaine dsRBD.Cette liaison de l'ARNdb
entraîneun changement de conformation permettant sa dimérisation àpartir de sonextrémité N terminale, donnantaccès à son site catalytique C-terminal. L'activation de PKR nécessite
également son autophosphorylation au niveau de ses résidusthréonine Thr446 et Thr45171,72.
A noter que PKR peut également être activée, en absence d'ARNdb, par la protéine
intracellulaire PACT (PKR-associated protein) via hétérodimérisation 73,74 L'activation de
PKR induit alors une inhibition de la traduction globale via la phosphorylation de eIF2a,
inhibant également la traduction des ARNm viraux, arrêtant ainsi la progression de
l'infection virale. Ceci représente le mécanisme de base par lequel PKR exerce sonactivité
antivirale 75. PKRa également un rôle dans des voies detransduction de signaux cellulaires
et le contrôle transcriptionnel via la voie IkB/NF-kB 76. PKR est aussi capable, via un mécanisme complexe, d'induire l'apoptose des cellules afin de prévenir, entre autres, une
propagation virale plus importante 75,77 Enfin, PKR est impliquée dans le contrôle de la croissance et la prolifération cellulaire avec des fonctions de suppresseur de tumeurs 78.
Cependant, l'implication précise de PKR dans le développement du cancer dépend du type
de tumeur, du stade de développement tumoral ainsi que du microenvironnement tumoral.
Initialement, PKR a été décrit comme un suppresseur de tumeur car sa surexpression
empêche la croissance cellulaire chez la levure. PKR interagit également avec des gènes
suppresseursde tumeur telsque p53 ou PTEN 79. Dans des hépatocarcinomes,PKR inhibe la
réplication virale agissant ainsi comme suppresseur detumeur. Cependant, d'autres études
ont montré quePKRpeutpromouvoir la croissance des cellules cancéreuses via la régulation des voies de signalisation MAPK et sTAT 79.
o La kinase PERK
Laprotéine PERK (PKR-like ER kinase) est une sérine/thréonine kinase généralement
activée lors d'un stress duréticulum endoplasmique (RE)qui correspond à une accumulation
de protéines mal repliéesou mutantes au sein du RE 80,81. PERK est située au niveau de la membrane duRE. sonextrémité C-terminale estcytoplasmique et contientle domainekinase
ainsi que les sites d'autophosphorylation 80. son extrémitéN-terminale se trouveà l'intérieur
dela lumière duREet contient les régions importantes pourladimérisation, la régulationet l'associationavec la protéinechaperonne BiP (binding immunoglobulin protein) (Figure 5).
En conditions de croissance cellulaire normales, PERK est liée à la protéine BiP dans la lumière du RE via sa partie N-terminale inactivant le domaine kinase cytoplasmique. En
situation défavorable de croissance cellulaire engendrant un stress protéotoxique due à
l'accumulation de protéines malforméesau niveau duRE, la kinase PERK est activéelorsdu
déclenchement d'une réponse physiologique appelée réponse uPR (unfolded protein
response)82,83. Lors de cetteréponse au stress, BiP se dissocie de PERK permettant à cette
dernière de s'oligomériser. Cette dimérisation permet à PERK de s'autophosphoryler au niveau de plusieurs résidus dans le domaine kinase C-terminal, dont la Thr980 située dans la boucle d'activation de la kinase. PERK, ainsi activée, peut alors phosphoryler la ser51
d'eIF2a 84. Cette phosphorylation d'eIF2a bloquelasynthèsede nouvelles protéines et réduit
donc l'entrée de nouveaux polypeptides malformés dans la lumière du RE. Cela laisse du