DEVELOPPEMENT D'OUTILS MOLECULAIRES POUR DETERMINER LES NIVEAUX D'ACTIVITE DES GROUPES BACTERIENS PRESENTS DANS LE
LISIER DE PORC
par
Caroline Roy
memoire presents au Departement de biologie en vue de l'obtention du grade de maitre es sciences (M.Sc.)
FACULTE DES SCIENCES UNIVERSITE DE SHERBROOKE
1*1
Library and Archives Canada Published Heritage Branch 395 Wellington Street Ottawa ON K1A0N4 Canada Bibliotheque et Archives Canada Direction du Patrimoine de I'edition 395, rue Wellington Ottawa ON K1A0N4 CanadaYour file Votre reference ISBN: 978-0-494-49578-0 Our file Notre reference ISBN: 978-0-494-49578-0
NOTICE:
The author has granted a non-exclusive license allowing Library and Archives Canada to reproduce, publish, archive, preserve, conserve, communicate to the public by
telecommunication or on the Internet, loan, distribute and sell theses
worldwide, for commercial or non-commercial purposes, in microform, paper, electronic and/or any other formats.
AVIS:
L'auteur a accorde une licence non exclusive permettant a la Bibliotheque et Archives Canada de reproduire, publier, archiver,
sauvegarder, conserver, transmettre au public par telecommunication ou par I'lnternet, prefer, distribuer et vendre des theses partout dans le monde, a des fins commerciales ou autres, sur support microforme, papier, electronique et/ou autres formats.
The author retains copyright ownership and moral rights in this thesis. Neither the thesis nor substantial extracts from it may be printed or otherwise reproduced without the author's permission.
L'auteur conserve la propriete du droit d'auteur et des droits moraux qui protege cette these. Ni la these ni des extraits substantiels de celle-ci ne doivent etre imprimes ou autrement reproduits sans son autorisation.
In compliance with the Canadian Privacy Act some supporting forms may have been removed from this thesis.
While these forms may be included in the document page count,
their removal does not represent any loss of content from the thesis.
• * •
Canada
Conformement a la loi canadienne sur la protection de la vie privee, quelques formulaires secondaires ont ete enleves de cette these.
Bien que ces formulaires aient inclus dans la pagination, il n'y aura aucun contenu manquant.
Le 30 novembre 2007
lejury a accepte le memoire de Mme Caroline Roy dans sa version finale.
Membres dujury
Mme Carole Beaulieu Directrice
Departement de biologie
Madame Caroline Talbot Codirectrice Departement de biologie
Mme Marie-France Palin Membre
Departement de biologie
M. SebastienRoy President-rapporteur Departement de biologie
SOMMAIRE
La production porcine represente une part importante de l'economie au Quebec. Bien que Ton note une diminution du nombre de fermes possedant des pores, celles qui demeurent se specialised et augmentent leur volume. La bonne gestion du lisier produit est par consequent imperative pour le maintien de la sante des sols et des cours d'eau en plus d'une bonne qualite de Fair. Les odeurs et autres composes nuisibles sont produits lors de la degradation anaerobie de la matiere organique par les microorganismes indigenes au systeme digestif. Les etudes portant sur les communautes microbiennes du systeme digestif et du lisier de pore sont encore peu nombreuses. La connaissance de cette population microbienne et la comprehension de leurs interactions sont fondamentales dans Pelaboration des techniques de traitement. L'une de ces methodes de traitement est la digestion anaerobie effectuee par un bioreacteur.
Pour etudier les communautes mibrobiennes, Putilisation de methodes moleculaires, telles Pheterogeneite des longueurs d'amplicon PCR (LH-PCR) et le polymorphisme des longueurs de fragment terminal de restriction (T-RFLP), permet la detection d'organismes non-cultivables en laboratoire. Le projet presente dans ce memoire vise a demontrer Pefficacite de ces deux techniques d'empreintes a ADN (profils de phylotypes) dans Petude des dynamiques de communautes microbiennes dans le lisier de pore, ainsi que la quantification de certains groupes microbiens par PCR en temps reel. II vise egalement a caracteriser les microorganismes metaboliquement actifs via Panalyse de PARN ribosomal (ARNr) 16S. De plus, ce travail veut montrer Putilite de differentes analyses statistiques permettant le traitement des nombreuses donnees produites de maniere a en degager des interpretations sur le dynamisme microbien.
Pour atteindre ces objectifs, deux approches experimentales ont ete utilisees. La premiere approche consistait a Petude de la dynamique des communautes microbiennes d'un bioreacteur a huit compartiments a ecoulement piston. La seconde visait a determiner les differences de population entre des lisiers bruts preleves a la ferme. Les resultats obtenus du traitement par bioreacteur ont montre une specialisation des compartiments dans la digestion de la matiere organique. Durant les six mois d'operation, chaque compartiment
s'est specialise et a favorise le developpement d'une population qui lui etait propre et presentait differents niveaux d'acides gras volatils (AGV) et de degagement de methane. Des phylotypes qui pourraient expliquer la difference entre les profils obtenus ont ete identifies. De plus, des profils semblables ont ete correles a la production d'acides gras volatils ou de methane. La seconde approche experimentale portait sur la caracterisation de trois lisiers bruts preleves a un mois d'intervalle et provenant des operations de finition, ainsi que d'un melange de lisiers de finition et de maternite. Les analyses des profils de phylotypes obtenus ont mene a la conclusion que la structure des communautes bacteriennes et archaebacteriennes des trois lisiers de pore provenant des operations de finition etaient semblables mais se distinguaient de celle du lisier mixte provenant des operations de finition et de maternite. De plus, ces analyses ont permis de faire la detection de changements subtils entre les echantillons.
Dans ce travail, il a ete montre que l'etude au niveau de PARNr des bacteries etait plus appropriee qu'au niveau de l'ADNr. En effet, les resultats obtenus pour ces deux cibles etaient differents. L'utilisation de l'ARNr a permis d'observer plus precisement les changements des bacteries metaboliquement actives. Par contre, la difference entre les resultats de l'ADNr et l'ARNr pour les archaebacteries etait beaucoup moins marquee. Finalement, l'utilisation d'outils statistiques tels que les indices de diversite, l'analyse en composantes principales (PCA), le positionnement multidimensionnel non-metrique (NMS), l'UPGMA (unweighted pair group method with arithmetic mean) ainsi que l'analyse des especes indicatrices (ISA) a confirme leur utilite dans l'analyse des donnees complexes et la visualisation des resultats obtenus.
REMERCIEMENTS
Je remercie ma directrice de recherche Guylaine Talbot pour m'avoir offert cette maitrise et d'avoir cm en mes capacites. Elle a su rendre l'experience agreable et enrichissante tout en m'accordant toujours sa grande disponibilite. Elle est pour moi plus qu'un guide mais une amie. De plus, je remercie ma co-directrice Carole Beaulieu pour ses conseils et ses encouragements. De merae, je remercie Claude Dery pour son incroyable disponibilite et l'interet qu'il porte a tous les etudiants. Je remercie egalement Sebastien Roy pour le temps et Penergie portes a la correction et a revaluation de ce memoire.
Je desire egalement remercier la Federation des Producteurs de Pore du Quebec pour son important apport financier. Merci d'avoir cru en ce projet. De plus, je remercie Daniel Masse pour l'opportunite offerte ainsi que toute son equipe d'experts.
Je remercie tous les membres du laboratoire 211 du centre de recherche d'AAC de Lennoxville pour leur accueil, leur amitie, leurs conseils et leur comprehension. J'ai grandement apprecie l'atmosphere de camaraderie qu'ils ont si naturellement inspiree. Un gros merci a Marie-France Palin et Daniele Beaudry qui m'ont consideree comme un membre a part entiere de leur equipe. Je souligne aussi l'important apport de Benoit Labrecque par son ecoute, ses idees et surtout son amitie.
Je tiens aussi a remercier toute ma famille qui m'a soutenue et encouragee durant toute cette aventure mais surtout ma mere qui a toujours eu notre education a cceur. Un merci special a Paul-Aime Roy pour son aide financiere et a mes neveux qui m'ont laisse faire mon tres long devoir.
TABLE DES MATIERES
SOMMAIRE i REMERCIEMENTS iii
TABLE DES MATIERES iv LISTE DES ABREVIATIONS viii LISTE DES TABLEAUX x LISTE DES FIGURES xii INTRODUCTION 1
1.1. La production porcine et sa problematique environnementale 1
1.2. La degradation anaerobie du lisier 3
1.3. Les odeurs 5 1.3.1. Les acides gras volatils 6
1.3.1.1. Catabolisme desproteines 6 1.3.1.2. Catabolisme des hydrates de carbone 9
1.3.1.3. Catabolisme des lipides 9
1.3.2. Indicateur d'odeur 9 1.3.3. Microorganismes indigenes au pore 10
1.4. Methodes de microbiologie classique et moleculaire 13
1.5. Le gene de l'ARN ribosomal 16S 14 1.5.1. Activite metabolique 15 1.5.2. L'ARNr et les genes fonctionnels 15
1.5.3. Methodes de biologie moleculaire 16
1.5.3.1. LH-PCR 18 1.5.3.2. T-RFLP 18 1.5.3.3. DGGE 19 1.5.3.4. Hybridation de fluorescence in-situ (FISH) 20
1.5.3.5. PCR en temps reel 21 1.6. Approche experimentale 23
CHAPITRE 1
DYNAMIQUE DES COMMUNITES BACTERIENNES ET ARCHAEBACTERIENNES D'UN BIOREACTEUR DE TYPE ECOULEMENT PISTON TRAITANT LE LISIER
DEPORC 26
Bacterial and archaeal community dynamics from an anaerobic plug flow type digester
treating swine manure 28
Abstract 29 Introduction 30 Results and Discussion 32
VFA and CH4 production 32 LH-PCR and LH-RT-PCR analyses 35 T-RFLP and RT-T-RFLP analyses 39 UPGMA analysis 42 NMS 44 Diversity indices 47 Conclusion 51 Environmental procedures 51 Environmental samples 51 Volatile fatty acids and methane production 52
DNA and RNA co-extraction 52 Bacterial community analysis using LH-PCR and reverse transcription LH-PCR
(LH-RT-PCR) 53 Archaeal community analysis using T-RFLP and reverse transcription T-RFLP
(RT-T-RFLP) 54 Analysis on automated sequencer 55
Data analysis 55 Acknowledgments 56
CHAPITRE 2
COMPARAISON DES STRUCTURES DES COMMUNITES BACTERIENNES ET ARCHAEBACTERIENNES DU LISIER DE PORC ET DE L'ACTIVITE TRANSCRIPTIONNELLE PAR L'ANALYSE STATISTIQUE MULTIVARIEE DES
EMPREINTES GENETIQUES DE L'ADN ET DES ARN RIBOSOMAL 63
Comparison of swine manure bacterial and archaeal community structure and transcriptional activity by multivariate statistical analysis of ribosomal DNA and RNA
fingerprints 65 Abstract 66 Introduction 67 Materials and methods 68
Environmental samples 68 Volatile fatty acids production 69 DNA and RNA co-extraction 69 Bacterial community analysis using LH-PCR and reverse transcription LH-PCR
(LH-RT-PCR) 69 Archaeal community analysis using T-RFLP and reverse transcription T-RFLP
RT-T-RFLP) 70 Analysis on automated sequencer 71
Data analysis 71 Results and discussion 72
Volatile fatty acids production 72 DNA- and RNA-based length heterogeneity polymerase chain reaction (LH-PCR)
analysis 74 DNA- and RNA-based terminal restriction fragment length polymorphism (T-RFLP)
analysis 81 Conclusions 87 Acknowledgments 87
CONCLUSION 92 ANNEXE
PCR EN TEMPS REEL 95
1.0. Introduction 95 2.0. Materiel et methodes 96
2.1. Designs des amorces oligonucleiques desbacteries cibles 96 2.2. Culture de Thermus aquaticus et decompte microbien microscopique 98
2.3. Extraction de l'ARNr 16S et synthese de l'ADNc 98 2.4. Extraction de l'ADN genomique (ADNg) des bacteries en cultures pures 99
2.5. Courbes d'etalonnage et specificite des designs 99 2.6. Essais de PCR en temps reel du lisier en traitement dans un bioreacteur de type
ecoulement piston 100 3.0 Resultats et Discussion 100 3.1. Courbes d'etalonnage 100 3.2. Verification de la specificite des designs 102
3.3. Resultats des echantillons du lisier en traitement 109
4.0. Conclusion 112
LISTE DES ABREVIATIONS 16S 23S ACP ADN ADNc ADNdb ADNg ADNr ARN ARNm ARNr AGV ATP bp °C cDNA CH4 cm
co
2 COD Ct ARn DGGE EB xg GC H2 H20 H2Spetite sous-unite ribosomale 16 Svedbergs grande sous-unite ribosomale 23 Svedbergs analyse en composantes principales acide desoxyribonucleique ADN complementaire ADN double brin ADN genomique ADN ribosomal acide ribonucleique ARN messager ARN ribosomal acide gras volatil adenosine triphosphate paire de bases degre Celsius ADN complementaire methane centimetre dioxyde de carbone demande chimique en oxygene cycle seuil delta de reaction electrophorese de gel denaturant a gradient tampon d'elution fois la gravite guanine et cytosine dihydrogene eau sulfure d'hydrogene
ISA IV L LH-PCR m/s mg min mM N20 NH3 NMS nM dNTP P pb PCA PCR PEG PVC R2 REA RT-PCR s SSCP T-RFLP T-RF U ul UNG UPGMA VFA
analyse d'especes indicatrices valeur indicatrice litre heterogeneite des longueurs d'amplicon PCR metre par seconde milligramme minute milimolaire oxyde nitreux
ammoniac positionnement multidimensionnel non-metrique
nanomolaire desoxynucleotide triphosphate
probabilite paire de bases analyse de composante principale polymerase chain reaction polyethylene glycol chlorure de polyvinyle coefficient de determination reglement sur les exploitations agricoles PCR de transcription inverse seconde polymorphisme de conformation simple brin polymorphisme des longueurs de fragment terminal de restriction fragment terminal de restriction unite microlitre uracile N-glycosylase unweighted pair group method with arithmetic mean
LISTE DES TABLEAUX Introduction
1. Acides gras volatils obtenus par desamination par les bacteries en mode
anaerobie dans le systeme gastro-intestinal et les dejections animales 8 2. Genres bacteriens indigenes au lisier de pore et leurs composes odorants 12
Chapitre 1
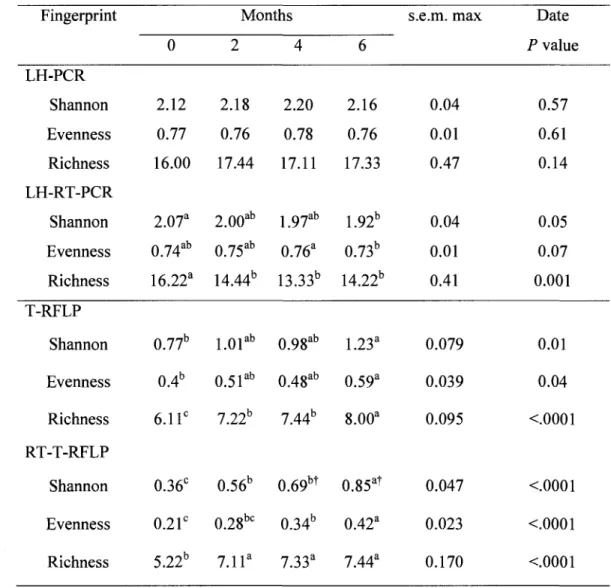
1. Effect of sampling date on diversity indices 49
2. Analysis of diversity indices in the compartments 50
Chapitre 2
1. Indicator species analysis from LH-PCR and LH-RT-PCR fingerprint data... 76
2. Diversity indices calculated from LH-PCR and LH-RT-PCR fingerprints 77
3. Diversity indices calculated from T-RFLP and RT-T-RFLP fingerprints 82
4. Indicator species analysis from T-RFLP and RT-T-RFLP fingerprint data 83
Annexe
1. Designs d'amorces utilises pour le PCR en temps reel et les courbes
d'etalonnage 97
3. Specificite du design Clostridium coccoides pour le PCR en temps reel 104
4. Specificite du design Syntrophomonas pour le PCR en temps reel 105
5. Specificite du design Peptostreptococcus pour le PCR en temps reel 106
6. Specificite du design Methanosaeta pour le PCR en temps reel 107
LISTE DES FIGURES
Introduction
1. Les trois etapes de la degradation anaerobie du lisier de pore par
methanogenese 4 2. Production et utilisation des composes odorants en condition anaerobie 6
Chapitre 1
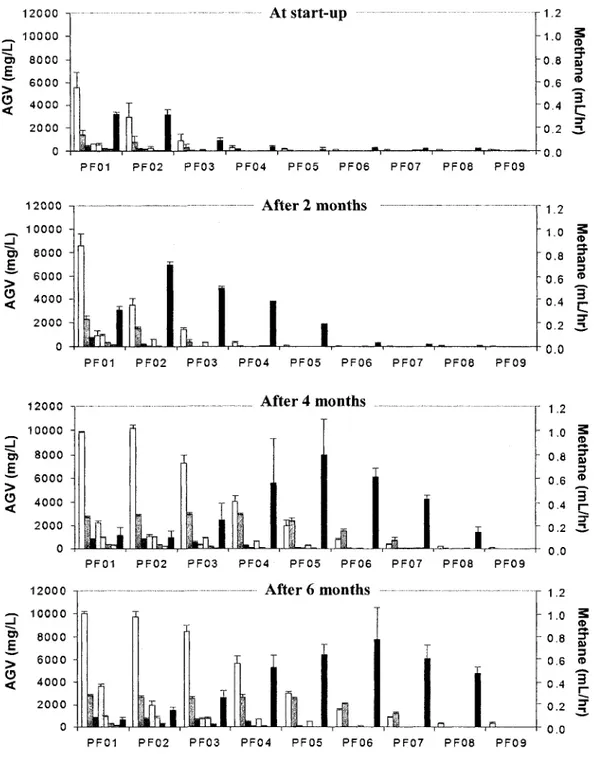
1. Volatile fatty acids concentration and methane production rate in the reactor compartments (PF01-PF08) and the effluent (PF09) during a 6-month
trial 34
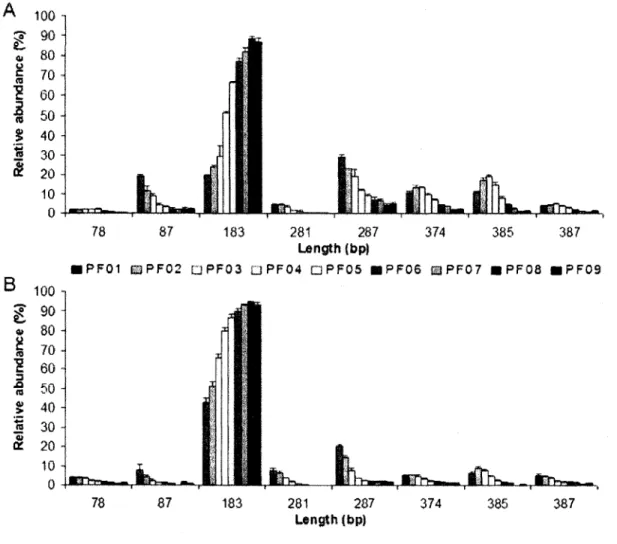
2. Standardized 16S rRNA-based profiles obtained after 6 months of operation
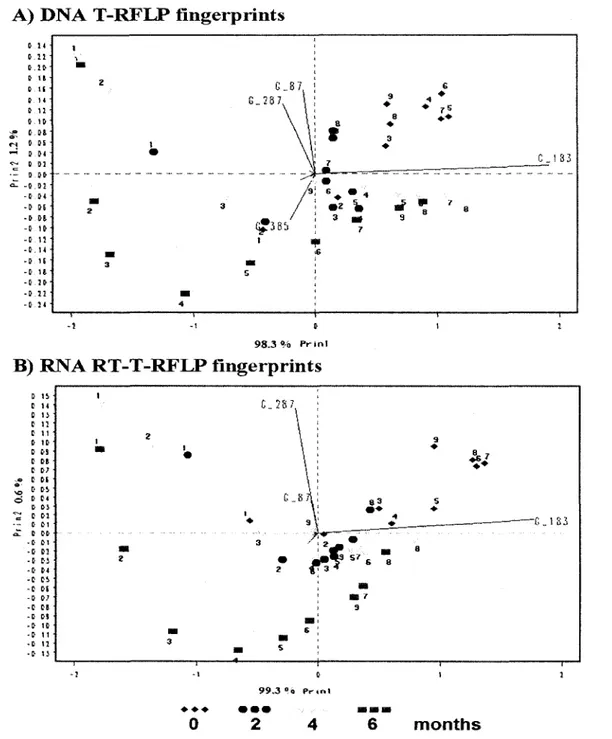
A. LH-PCR; B. LH-RT-PCR 36
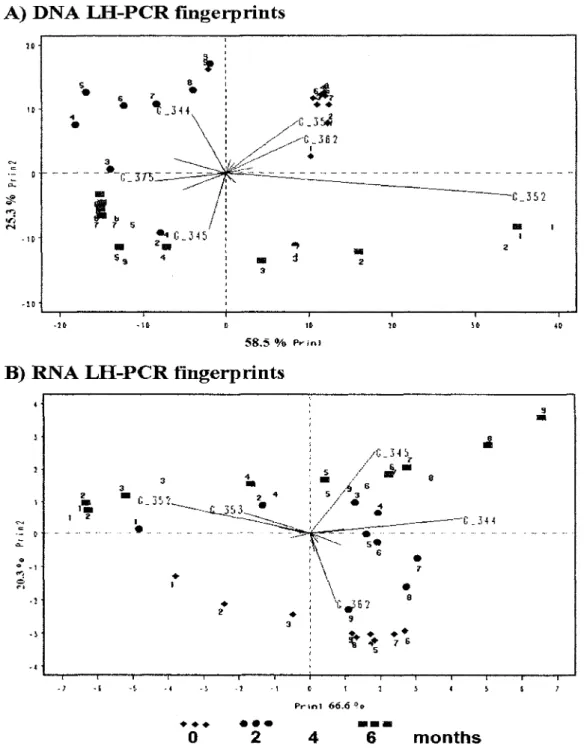
3. Principal components analysis of (A) LH-PCR and (B) LH-RT-PCR
fingerprints after 0, 2, 4 and 6 months 38
4. Standardized 16S rRNA profiles obtained after 6 months of operation.
A. T-RFLP; B. RT-T-RFLP 39
5. Principal components analysis of (A) T-RFLP and (B) RT-T-RFLP
fingerprints after 0, 2, 4 and 6 months 41
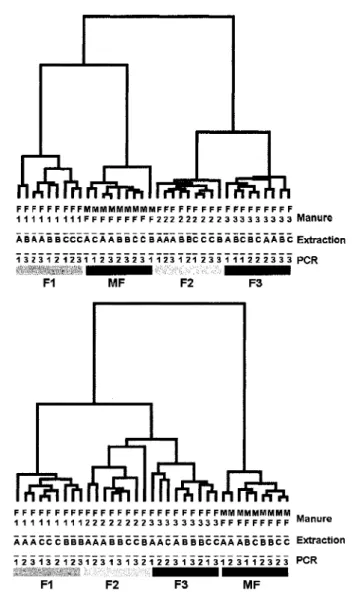
6. Comparison of fingerprints by UPGMA cluster analysis from (A) LH-PCR; (B) LH-RT-LH-PCR; (C) T-RFLP; and (D) RT-T-RFLP fingerprints
7. Overlays of (A) DNA LH-PCR and (B) RNA LH-PCR fingerprints with total volatile fatty acids level and methane production rate using nonmetric
multidimensional scaling (NMS) ordination 45
8. Overlays of (A) DNA T-RFLP and (B) RNA T-RFLP fingerprints with total volatile fatty acids level and methane production rate using
nonmetric multidimensional scaling (NMS) ordination 46
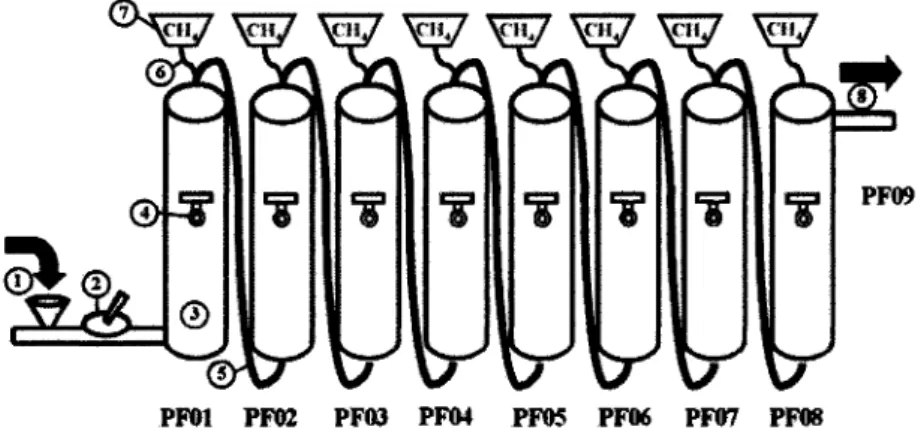
9. Schematic diagram of an 8-compartment plug-flow reactor 52
Chapitre 2
1. Volatile fatty acids concentration (A) and relative percentage (B) for the
manures Fl, F2, F3 and MF 73
2. UPGMA dendrograms of A. LH-PCR and B. LH-RT-PCR fingerprints 74
3. LH-PCR (A) and LH-RT-PCR (B) fingerprints from manures F1, F2, F3
andMF 78
4. Biplot representation of Principal Components Analysis of (A) LH-PCR and
(B) LH-RT-PCR fingerprints from manures F1, F2, F3 and MF 80
5. UPGMA dendrograms of A. T-RFLP and B. RT-T-RFLP samples 81
6. T-RFLP (A) and RT-T-RFLP (B) fingerprints from manures #1, #3, #4,
and #5 84 7. Biplot representation of Principal Components Analysis of (A) T-RFLP
Annexe
1. Courbes d'etalonnage des differents designs pour les PCR en temps reel 102
2. Nombre de copies d'ARNr 16S par mL provenant des compartiments 1, 3, 5
et 8 du bioreacteur 110
3. Resultats du rapport des PCR en temps reel pour les differents designs sur
INTRODUCTION
1.1. La production porcine et sa problematique environnementale
La production porcine est une industrie importante au Quebec. En effet, selon des donnees de 2002, elle representait 3,2 milliards de dollars en retombees economiques (Federation des Producteurs de Pore du Quebec, 2005). Lors d'un recensement au commencement de l'annee 2007, le Quebec possedait la plus grande part de cette industrie, soit 28,51% des pores du Canada (Conseil canadien du pore, 2007). Au Quebec comme dans le reste du pays, le nombre de fermes porcines ne cesse de diminuer. En trente ans, elles sont passees de 9 067 a 2 490. Par contre, elles se specialised et voient leur echelle de production augmenter passant de 178 a 1 635 betes en moyenne par ferme (Federation des Producteurs de Pore du Quebec, 2005; Conseil canadien du pore, 2007). Cette concentration des animaux en mega-porcheries fait peur au public qui craint des repercussions nocives sur leur environnement. Ces dernieres peuvent etre classees en trois categories selon qu'elles affectent le sol, l'eau ou Fair, mais toutes sont dues au lisier.
L'epandage du lisier sur les terres agricoles est depuis longtemps une facon de les enrichir. Par contre, le besoin nutritionnel de ces terres n'est pas toujours en equilibre avec l'apport du lisier a epandre. Par consequent, le sol peut accumuler une trop grande concentration d'azote, de phosphore et de mineraux comme le cuivre et le zinc (Foulkes et al, 2006; Jongbloed and Lenis, 1998). Bien que le phosphore soit un nutriment essentiel a la croissance vegetale, la concentration maximale en phosphore que peuvent soutenir les terres agricoles est de 0,01 a 0,03 mg/L. En ce qui concerne le nitrate, son seuil tolerable pour l'eau douce est de 50 mg/L, ce qui est souvent excede (Tabbara, 2003). Lorsque l'apport du lisier en phosphore et en nitrate est superieur a ces seuils, le lessivage par les eaux de ruissellement favorise l'eutrophication des cours d'eau provoquant une penurie d'oxygene. Le Ministere du Developpement Durable, de l'Environnement et des Pares (2005) a publie une etude sur la capacite de support des rivieres du Quebec pour le phosphore residuel decoulant des activites agricoles. lis ont demontre que 83% des cours d'eau en territoires agricoles avaient depasse le seuil d'eutrophication. La pollution de l'air
par le lisier de pore provient surtout de la liberation d'odeurs derangeantes et de gaz a effet de serre comme le gaz carbonique (C02), le methane (CH4) et l'oxyde nitreux (N2O).
Puisque la problematique environnementale est maintenant connue et acceptee, le gouvernement a pris des mesures legislatives afin de coordonner les pratiques de toute l'industrie agricole par le Reglement sur les exploitations agricoles (REA) (Ministere de l'Environnement du Quebec, 2002; Ministere de l'Environnement du Quebec, 2005). En effet, les producteurs de pores comme tous les autres producteurs doivent se conformer a cette reglementation. Tout d'abord, Pepandage du lisier sur les terres agricoles n'est pas permis durant la periode de gel au sol. Consequemment, le surplus de lisier doit alors etre entrepose de fa9on etanche afin d'eviter les ecoulements. Aussi, le REA a limite la quantite de depot de matieres fertilisantes sur les terres selon leur besoins et leur capacite a retenir le phosphore selon des abaques (Federation des Producteurs de Pores du Quebec, 2002). Les producteurs faisant face a des surplus de lisier doivent alors trouver des moyens pour abaisser leur niveau phosphore. Premierement, ils peuvent reduire a la source la charge de phosphore et du volume de leur lisier. Diverses possibilites s'offrent a eux comme Pincorporation de phytase a la moulee, ou Pajustement de la diete selon Page et le sexe du pore (Jongbloed et Lenis, 1998; Knowlton et al, 2004). De plus, ils peuvent faire l'achat ou la location de superficie additionnelle en culture afin d'y epandre le lisier. Bien sur, des frais en decoulent, pouvant varier selon le cout de l'achat de nouvelles terres, la distance des terres, la marge de profit des cultures et Papport en phosphore pouvant etre depose sur ces terres. Finalement, le traitement du lisier peut aussi etre considere. Par exemple, l'utilisation de bioreacteurs permet la separation du phosphore qui s'accumule dans leurs boues sedimentees (Masse et al, 2007). La purge de ces boues allege la charge en phosphore livree par le lisier lors de Pepandage.
Pour ameliorer le probleme des odeurs, plusieurs facons de faire et technologies sont disponibles aux producteurs. Au batiment, un environnement propre, un plancher partiellement latte, une diete appropriee aux besoins specifiques des pores, une ventilation adequate et la separation de Purine des feces peuvent aider a reduire la presence des odeurs. Aussi, des moyens visant la reduction des odeurs a Pentreposage du lisier peuvent etre mis en place tels que le type de toiture des bassins, Paeration du lisier, les traitements
chimiques (ex.: controle du pH), les bioreacteurs de digestion anaerobie, la biofiltration et le refroidissement du lisier. Puis, le type de rampe d'epandage, les additifs chimiques et la presence de brise-vents permettent de minimiser les odeurs a Pepandage (Federation des Producteurs de Pores du Quebec, 2002).
1.2. La degradation anaerobie du lisier
L'entreposage du lisier dans des bassins favorise la formation d'un milieu sans oxygene qualifie d'anaerobie. Certains microorganismes provenant initialement du systeme digestif du pore degradent la matiere organique pour former du biogaz constitue surtout de methane et de C02. Contrairement a la degradation aerobie ou les bacteries peuvent
transformer directement la matiere organique complexe en CO2 et en eau (H2O); en absence d'oxygene, il s'agit plutot d'une serie d'etapes effectuees par differents microorganismes rendant le processus plus complexe dans son ensemble. Les microorganismes participant au processus sont specialises a certaines etapes et par ce fait, l'energie demandee pour la regulation de leur metabolisme en est reduite (Schink, 2006).
II est generalement admis que la degradation anaerobie se fait en trois etapes impliquant effectivement une communaute microbienne propre a chacune (Zhang et at, 2004), comme le montre la figure 1. La premiere etape est l'hydrolyse des molecules complexes en plus simples ainsi que la fermentation, aussi appelee acidogenese. Tout d'abord, les bacteries hydrolytiques reduisent des composes organiques complexes en molecules simples tels des sucres, des acides amines, des acides gras et des alcools par la secretion d'enzymes extracellulaires (Masse et Droste, 2000; Schink, 1997). Par la suite, les bacteries acidogenes transforment ces produits en molecules encore plus simples comme l'ammoniac (NH3), le sulfure d'hydrogene (H2S) et les acides gras volatils (AGV), ces
derniers constituant les principaux composes formes. De plus, il y a formation de gaz carbonique, d'hydrogene (H2) ainsi que d'acetate. Lors de la seconde etape, e'est-a-dire l'acetogenese, ces composes intermediaires (NH3, H2S, AGV, etc.) sont a leur tour
Hydrolyse et Fermentation
matiere organique
(hydrates de carbone, proteines et iipides)
Acetosenese
m
I;; iiiy Sy iaidjifes; griais; jvieijiaJcijis; (A0:VtK: gjsiOioiijy III; I l^rriltri^ i S J ^ i & # & s j Iq^jj&isfes; ^oJEnatlqjiiieisiI
n,.co.
KefdttMclluinc. t (K. humus
Figure 1. Les trois etapes de la degradation anaerobie du lisier de pore par
methanogenese. Adapte de Schink (1997) et Schink (2006). 1 bacteries hydrolytiques et fermentaires ; 2 bacteries acetogenes productrices de H2 ; 3 bacteries acetogenes
consommatrices de H2 ; 4 methanogenes hydrogenotrophes ; 5 methanogenes acetotrophes.
bacteries fermentaires secondaires et de syntrophes (Schink, 2006). II existe egalement des bacteries acetogenes consommatrices de H2 qui produisent de Pacetate a partir de l'utilisation de H2. Finalement, la derniere etape de la degradation anaerobie du lisier est la methanogenese produite par deux groupes trophiques de methanogenes. Les procaryotes responsables de cette production de methane sont des archaebacteries. Les methanogenes hydrogenotrophes metabolisent le H2 et le CO2 et produisent 25% du methane alors que les methanogenes acetotrophes ou utilisent plutot l'acetate, ils produisent 75% du methane (Masse et Droste, 2000). La cooperation syntrophique entre les bacteries acetogenes productrices de H2 et les methanogenes est capitale a la formation du biogaz par un transfert d'hydrogene donnant lieu a des conditions metaboliques et energetiques favorables (Schink, 1997). Les bacteries syntrophes ont besoin d'une faible concentration de H2 afin de permettre la conversion de leurs substrats, comme les AGV inaccessibles aux
methanogenes, en CO2. Cela est possible grace a Paction des methanogenes hydrogenotrophes qui maintiennent ces bas niveaux de H2 en les convertissant en CH4 (Zhang et al, 2004).
1.3. Les odeurs
Le lisier de pore se retrouve sous forme liquide et est constitue du melange des urines, des feces ainsi que des eaux de lavage a la ferme. L'entreposage du lisier engendre une importante formation de gaz responsables des odeurs mais leur diffusion a travers le lisier se fait lentement puisque ce milieu reste au repos. Lors du brassage des fosses et de Pepandage au champ, la liberation dans Pair des gaz odorants est fortement accrue. Les composes odorants degages proviennent de la degradation des matieres organiques, principalement en absence d'oxygene, par des microorganismes. Theoriquement, cette degradation se conclue par la production du biogaz compose de methane et de gaz carbonique, et de Phumus. Ce dernier consiste de la matiere organique stabilised et d'elements mineralises. Cependant, la pratique en est tout autre. En effet, puisque chaque etape de la degradation demande Paction de groupes bacteriens differents qui necessitent des conditions de milieux et de substrats differents, le lisier de la ferme ne subit pas une degradation complete (Pigeon, 2004). Ainsi, les composes intermediaires odorants de la degradation ne seront pas tous metabolises par les bacteries et pourront etre liberes dans Penvironnement, ce qui affectera la qualite de vie des producteurs et des membres de leur voisinage.
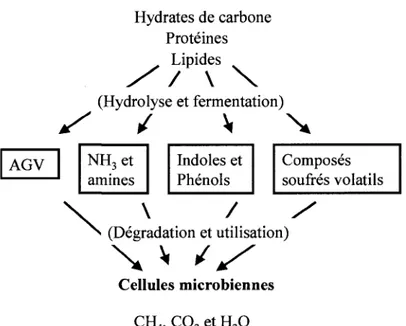
D'apres O'Neill et Phillips (1992), il existerait plus de 170 composes chimiques produits lors de la degradation anaerobie du lisier. Mackie et al. (1998) les ont classes en quatre categories chimiques (figure 2) so it: 1) les acides gras volatils, 2) les alcools, dont les indoles et les phenols (incluant indole, scatole, cresol, et 4-ethylphenol), 3) Pammoniac et les amines volatiles (incluant putrescine, cadaverine, methylamine et ethylamine) et finalement, 4) les composes volatils sulfures (sulfide, methyl- et ethyl-mercaptans).
Matieres organiques Hydrates de carbone Proteines Lipides / / \ \ (Hydrolyse et fermentation) AGV NH3et amines Indoles et Phenols Composes soufres volatils
\ / y
(Degradation et utilisation) Cellules microbiennes CH4, C02 et H20Figure 2. Production et utilisation des composes odorants en condition anaerobic D'apres Mackie et al. (1998).
1.3.1. Les acides gras volatils
Les acides gras volatils retrouves dans le lisier de pore sont generalement les acides acetique, propionique, butyrique, z'so-butyrique, valerique, wo-valerique, caproi'que et caprique. Ceux-ci proviennent de la degradation, ou catabolisme, des hydrates de carbones, des proteines et des lipides contenus dans le lisier (voir figure 2).
1.3.1.1. Catabolisme des proteines
La premiere etape de la degradation des proteines est Phydrolyse, par des proteases associees a la surface membranaire des bacteries hydrolitiques, qui brisent les liens peptidiques entre les acides amines. Ces derniers peuvent alors etre assimiles par les bacteries. Le tableau 1 montre les acides gras volatils produits lors du catabolisme des differents acides amines. La premiere etape de la degradation proteique est la desamination
oxydative qui consiste a enlever le groupement amine de l'acide amine tel qu'illustre par la formule suivante (Mackie et al, 1998):
NH2 i
R-CH-COOH + 2H20 -> R-COOH + NH3 + 2H2 + C02 (1)
Par la suite, les acides desamines peuvent subir differentes transformations. Generalement, ils sont convertis en pyruvate et en acetyl-CoA pour produire de l'energie par le cycle de Krebs. Puisqu'il y existe 20 acides amines, les voies metaboliques de decomposition par differents microbes sont done complexes. En plus des acides gras volatils, la degradation des proteines produit des acides carboxyliques a chaine aliphatique, des acides gras a chaine ramifiee, des composes sulfures, des amines, de l'ammoniac, du phenol et des indoles (Rappert et Miiller, 2005). Les travaux de Hobbs et al. (1996) ont montre que la reduction des proteines brutes dans les moulees de pores en croissance et en finition permettait de reduire la concentration d'AGV dans le lisier. Aussi, on a note une diminution de l'ammoniac et d'autres composes tels Pindole, le p-cresol et le scatole. Les acides gras volatils degages du lisier de pore proviennent principalement de la decomposition des proteines par les microorganismes comme le montrent les travaux de Miller et Varel (2003). Dans le but d'identifier les sources d'odeurs durant la fermentation de lisier, differents substrats (amidon, caseine et cellulose) ont ete ajoutes. Les composes considered comme les plus representatifs de l'odeur (AGV ramifies et les composes aromatiques) etaient toujours produits mais e'est Pajout de la caseine, utilisee comme proteine temoin, qui en a fait produire le plus. Ces auteurs ont conclu que la fermentation proteique dans le lisier de pore frais etait responsable de pres de la moitie de la perte en substrat alors qu'elle n'a pu etre detectee dans le lisier de vache frais. En combinant ces resultats avec ceux d'une etude precedente des memes auteurs sur les lisiers de vache (Miller et Varel, 2002), nous pouvons conclure que l'amidon serait le substrat principal et prefere des microorganismes fermentaires du lisier de vache. Bien que les bacteries fermentaires du lisier de pore utilisent aussi preferentiellement l'amidon, on en retrouve relativement peu par rapport aux proteines. Par consequent, toutes les proteines disponibles dans le lisier sont catabolisees et produisent alors d'avantage de composes odorants.
Tableau 1. Acides gras volatils obtenus par desamination par les bacteries en mode anaerobie dans le systeme gastro-intestinal et les dejections animales
Adapte de Muller et Rappert (2005)
Acides amines AGV produits
Alanine Asparagine Acide aspartique Cysteine Acide glutamique Phenylalanine Glycine Histidine Isoleucine Lysine Leucine Methionine Proline Glutamine Arginine Serine Threonine Valine Tryptophane Tyrosine Acetate, propionate Acetate, lactate
Acetate, lactate, propionate
Acetate, propionate, butyrate, valerate, formate Acetate, propionate, butyrate, lactate, formate Phenylpyruvate, phenyllactate, phenylacetate, phenylpropionate, benzoate
Acetate
Acetate, butylate, lactate, formate Isobutyrate, isovalerate, valerate Acetate, butyrate
Isobutyrate, isovalerate, valerate cc-cetobutyrate
Acetate, valerate, propionate Acetate, butyrate, lactate, formate Acetate, propionate, valerate
Acetate, propionate, butyrate, valerate, lactate, formate
Acetate, propionate, butyrate, valerate, formate Isobutyrate, isovalerate, valerate
Indolepyruvate, indolelactate, indoleacetate
Phenylacetate, phenylpropionate, 4-hydroxyphenyl-pyruvate,
4-hydroxyphenyllactate, 4-hydroxyphenylpropionate, hydroxyphenylacetate, 3-hydroxyphenylpropionate, 4-hydroxybezoate
1.3.1.2. Catabolisme des hydrates de carbone
La voie la plus importante pour le catabolisme microbien des hydrates de carbone (glucides) est la glycolyse ou le sucre est transforme en pyruvate et ce, en presence comme en absence d'oxygene. Dans un environnement anaerobie, le pyruvate passe par plusieurs processus de fermentation et sert aux reactions subsequentes dans lesquelles les acides gras volatils sont formes. Generalement, la fermentation des sucres produit des acides gras volatils a chaine aliphatique courte et droite comme l'acide acetique, propionique, lactique, succinique, formique et butyrique. Le catabolisme des sucres peut etre resume par 1'equation suivante :
34,5 C6H1206 -> 64 AGV + 23,75 CH4 + 34,25 C 02 + 10,5 H20 (2) (Zhu et al, 1999).
1.3.1.3. Catabolisme des lipides
Les lipides retrouves dans le lisier sont aussi degrades par les microorganismes. Tout d'abord, les lipides sont hydrolyses en acides gras a longue chaine, tels Poleate et le stearate, et en glycerol. Cette etape est catalysee par des lipases extracellulaires produites par des bacteries acidogenes. Par la suite, alors que le glycerol est fermente en differents alcools, formate et acides gras volatils, les acides gras a longue chaine sont oxydes en
acides gras a courte chaine par les bacteries acetogenes. Ultimement, P acetate et le H2
produits sont convertis en methane, C 02 et H20 (Angelidaki et al, 1999).
1.3.2. Indicateur d'odeur
L'odeur est une perception de l'odorat qui varie d'une personne a l'autre. Pour en faire F etude, il est necessaire de trouver des moyens de le quantifier. Connaitre les composes presents dans le lisier qui en sont responsables constitue la base dans le potentiel de generation malodorante. Cependant, les composes odorants sont souvent plus offensifs lorsqu'ils sont melanges ensemble et sentent differemment d'un produit pur. De plus, il n'est pas utile de s'attarder a tous les composes odorants puisqu'ils ne contribuent pas tous egalement a Pintensite de l'odeur. Chaque compose odorant a son seuil de perception et de
nuisance. Connaitre la portee de ces composes permet d'identifier les plus importants afin de determiner des indicateurs d'odeurs (Zhu et al, 1999).
L'odeur provenant des lisiers de pore peut etre evaluee par olfactometrie par panel humain qui permet d'obtenir le seuil de la concentration minimale ou un echantillon odorant peut etre distingue d'un echantillon inodore (Mackie et al, 1998; Pelletier et al, 2005; Trabue et al, 2006). Cependant, si elle est utilisee de facon routiniere, cette technique peut s'averer dispendieuse et complexe puisqu'elle demande l'implication humaine du panel (Zhang et al, 2006). Puisque la formation d'ammoniac dans le lisier est importante, il a ete initialement suppose qu'il pouvait s'agir d'un bon indicateur de l'odeur mais ce n'est pas le cas (Lunn et van De Vyver, 1977; Spoelstra, 1980; Zhu, 2000). Bien que l'ammoniac est produit lors de la degradation anaerobie des proteines, il est principalement engendre par l'hydrolyse de Puree (Rappert et Muller, 2005). Ce sont done plutot les AGV qui ont ete retenus par la communaute scientifique comme indicateur d'odeur (Williams, 1984; Zhu et al, 1999). En effet, Zahn et al (1997) ont rapporte que les acides organiques volatils de 2 a 9 carbones montraient le meilleur potentiel de l'odeur du lisier. Cependant, ce sont les AGV a longue chaine de carbone (C4 a C9) ou ramifies qui ont ete correles a une odeur plus importante (Zhu et al, 1999). Deja, plusieurs etudes ont utilise les AGV afin d'evaluer l'efficacite des techniques de traitement des lisiers de pore dans le reduction des odeurs (Castillo-Gonzalez et Bruns, 2005; Liao et al, 1997; Miller et Varel, 2003; Ndegwa et al, 2002; Zhang et al, 2000; Zhang et Zhu, 2005; Zhang et al, 2006).
1.3.3. Microorganismes indigenes au pore
Les bacteries responsables de la production d'odeurs lors de la degradation anaerobie, proviennent du systeme digestif de l'animal. En effet, e'est dans le tractus gastro-intestinal que commence la production d'acides gras volatils par sa microflore indigene. Cette derniere se retrouvera aussi dans les feces excretees et le processus se poursuivra si le milieu anaerobie est maintenu comme dans les fosses a lisier.
Depuis pres de quarante ans, quelques etudes ont ete menees afin de connaitre les genres bacteriens pouvant etre isoles des intestins et des feces chez le pore (Rail et al, 1970;
Russell, 1979; Zhu, 2000). Certaines methodes classiques de microbiologie ont isole et identifie certains genres bacteriens a partir de feces porcines. Les bacteries dominantes qui y etaient retrouvees sont les suivantes, aussi decrites au tableau 2. II y a les Streptococcus (Cotta et al, 2003), bacteries pour la plupart anaerobes facultatives. Toutes ces bacteries fermentent les hydrates de carbone et produisent principalement l'acide lactique et, en plus faibles proportions, les acides acetique, formique, propionique et butyrique (Zhu, 2000). II y a aussi les Peptostreptococcus qui sont des anaerobes chimioheterotrophes et qui catabolisent les acides amines en acides acetique, formique, iso-caprique, iso-butyrique, valerique et iso-valerique. Whitehead et Cotta (2004) ont isole des bacteries hyper productrices d'ammoniac provenant d'une fosse d'entreposage de lisier de pore. Parmi les plus efficaces, plusieurs etaient apparentees a Peptostreptococcus anaerobius selon P analyse phylogenetique de la sequence de leur gene encodant 1'ARN de la petite sous unite ribosomale 16S (ADNr 16S). Le lisier contient aussi les Eubacterium (Salanitro et
al, 1977) et les Clostridium (Russel, 1979) qui sont des bacteries anaerobes utilisant
surtout les hydrates de carbone et les acides amines, respectivement. Elles sont connues pour produire de grandes quantites d'acides gras volatils comme l'acide acetique, butyrique, formique, lactique, caproique, propionique, succinique, valerique, iso-butyrique, iso-caproi'que et iso-valerique en plus des indoles et phenols. Etant donne le grand nombre de ces produits odorants, le vaste genre Clostridium est une cible de choix dans P etude de la production des odeurs.
Les Lactobacillus (Yin et Zheng, 2005), aussi presents dans le lisier, ont une tolerance variee a Poxygene selon leur espece et produisent de l'acide lactique, du CO2, de Pethanol ou de l'acide acetique selon leur mode de fermentation. Puisque leur produit majeur est l'acide lactique, il peut etre assume que leur role dans Papport en composes odorants est negligeable (Zhu, 2000). Dans le lisier, le genre Escherichia est represente surtout par
E.coli (Salanitro et al, 1977) qui est anaerobe facultatif. II fermente le glucose et autres
hydrates de carbone en acides lactique, acetique et formique. Presque la totalite de ce genre bacterien produit de Pindole. Finalement, les Bacteroides (Cotta et al, 2003) sont des anaerobes chimioheterotrophes qui utilisent les hydrates de carbones ou la peptone. Leurs produits de fermentation sont les acides succinique, lactique, acetique, formique, propionique et butyrique.
Tableau 2. Genres bacteriens indigenes au lisier de pore et leurs composes odorants Adapte de Zhu (2000)
Genre bacterien Composes odorants potentiels Streptococcus Peptostreptococcus Eubacterium Clostridium Lactobacillus Escherichia Bacteroides
acides formique, acetique, propionique, butyrique, ammoniac et amines volatiles
acides formique, acetique, propionique, butyrique, iso-butyrique, valerique, caproi'que, iso-valerique, iso-caproi'que, ammoniac et amines volatiles
acides formique, acetique, propionique, butyrique, iso-butyrique, valerique, caproi'que, iso-valerique, iso-caproi'que, indoles et phenols
acides formique, acetique, propionique, butyrique, iso-butyrique, valerique, caproi'que, iso-valerique, iso-caproi'que, indoles et phenols acides formique, acetique, propionique et butyrique
acides formique, acetique, propionique et butyrique
acides formique, acetique, propionique, butyrique, iso-butyrique, valerique, caproi'que, iso-valerique, iso-caproi'que, ammoniac et amines volatiles
L'utilisation de techniques de biologie moleculaire dans ce domaine de recherche a permis de constater que la communaute microbienne du lisier de pore etait beaucoup plus vaste que ce qui etait anticipe au depart. Nous comprenons desormais que la communaute microbienne du lisier de pore est complexe. En effet, seulement 35 a 55% des phylotypes observes dans le lisier de pore par analyse de PADNr 16S etaient relies a un microorganisme connu (Peu et al, 2006; Snell-Castro et ah, 2005; Whitehead et Cotta, 2001). Neanmoins, la microflore du lisier de pore semble constituer un ecosysteme de digestion anaerobie qui est domine par des groupes de bacteries fermentaires telles Eubacterium, Clostridium, Lactobacillus, Streptococcus, Bacillus, Bacteroides (Cotta et ah, 2003; Leser et ah, 2002; Snell-Castro et ah, 2005; Whitehead et Cotta, 2001).
Nos connaissances sur les archaebacteries presentes dans le lisier de pore sont plus limitees. En effet, les sequences phylogenetiques disponibles dans les banques de donnees sont plutot restreintes et souvent non-identifiees. Le purin de pore s'avere lui aussi une source de nouvelles especes d'archaebacteries methanogenes. Snell-Castro et al. (2005) n'ont retrouve dans les fosses a lisier que des methanogenes hydrogenotrophes tels des Methanobacteriales et des Methanomicrobiales. Par contre, les travaux d'Angenent et al. (2002), portant sur la population microbienne d'un bioreacteur anaerobie, ont montre la presence de methanogenes acetotrophes de l'ordre Methanosarcinales. Dans celle-ci, la famille des Methanosaetaceae possede une grande affinite pour l'acetate seulement et presente une croissance relativement lente, alors que les Methanosarcinaceae ont une affinite pour l'acetate beaucoup plus faible mais croissent plus rapidement (Yu et al, 2006). Ainsi, lorsque peu d'acetate est disponible, les Methanosaetaceae sont favorises alors que dans le cas contraire, ce sont les Methanosarcinaceae qui le sont. Une etude recente d'un bioreacteur traitant le lisier de pore a permis la detection de plusieurs methanogenes dont Methanosaeta, Methanolobus, Methanobrevibacter, Methanosarcina, Thermoplasma et Methanospirillum (Padmasiri et al., 2007).
1.4. Methodes de microbiologic classique et moleculaire
L'identification bacterienne en microbiologie classique consiste, entre autres, a Putilisation de techniques de microscopie, de culture microbienne en laboratoire, de serologic et d'immunologie. De nos jours, ces techniques jouent toujours un role important dans la decouverte de microbes encore inconnus, non-caracterises et atypiques (Mackay, 2004). Par contre, ces methodes phenotypiques sont influencees par les mutations et peuvent etre sujettes aux biais d'interpretation (Petti et al, 2005). De plus, la culture microbienne en laboratoire peut etre limitee par des organismes a croissance tres lente ou non-cultivables en laboratoire. La communaute scientifique a change sa perception de la diversite microbienne dans l'environnement avec Parrivee des techniques de biologie moleculaire. Effectivement, on a decouvert que seulement un faible pourcentage des microorganismes presents dans les echantillons environnementaux pouvaient 6tre cultives selon les methodes classiques utilisees en microbiologie (Amann et al, 1995; Santos et Ochman, 2004). Au
seulement 10 a 20% pouvaient etre mises en culture sur differents milieux de culture (Peu et al, 2006). D'ailleurs, pres de 45% des phylotypes observes par l'analyse d'ADNr 16S etaient fortement apparentes a des microorganismes inconnus (Snell-Castro et al, 2005; Whitehead et Cotta, 2001).
Les methodes moleculaires ont l'avantage de cibler le materiel genetique microbien sans avoir a modifier les echantillons en isolant les individus. Bien qu'elle presente elle aussi des inconvenients, 1'identification bacterienne par genotypage a partir du gene de l'ARNr 16S est maintenant reconnue comme etant une approche plus objective, precise et fiable (Amann et al, 1995).
1.5. Le gene de l'ARN ribosomal 16S
Aujourd'hui, la cible de choix pour l'analyse des populations microbiennes d'echantillons environnementaux reste l'ARNr 16S. Cet ARN d'environ 1500 pb qui n'est pas traduit en proteine se lie aux proteines ribosomales pour former la petite sous-unite 30S du ribosome bacterien. Du a Pimportance que joue ce dernier dans la survie de la cellule, la sequence de son gene est rested tres conservee au cours de revolution. Des analyses comparatives de P architecture des ARNr ont revele des patrons dans lesquels des regions de structures primaires et secondaires alternaient avec des domaines variables qui different en poids moleculaires, en nucleotides et en structures secondaires (Gray et al, 1984). Ces regions conservees et variables font de l'ARNr 16S un marqueur devolution. Ce sont Woese et Fox (1977) qui, dans les annees 70, ont songe a utiliser PADNr 16S pour classer les organismes selon leur lien phylogenetique. En ciblant les regions tres conservees et universelles du gene, il etait alors possible de repecher des organismes non-cultivables ou inconnus. Quant aux regions plus variables, elles peuvent permettre de faire la distinction entre deux organismes apparentes.
II est a noter que le nombre d'operons des genes ribosomaux varie d'une espece a Pautre. La probability que des especes bacteriennes possedent le meme nombre de copies augmente avec leur rapprochement phylogenetique. II est done possible de surestimer certaines populations. Cette variabilite de la quantite d'operons ribosomaux est contestee
par certains auteurs. Par exemple, Lyons et al. (2000) n'ont montre qu'une petite variation des signaux PCR en temps reel entre quatre especes differentes utilisees afin de construire une courbe d'etalonnage suggerant que le nombre d'operons ribosomaux est un facteur negligeable pour la quantification.
1.5.1. Activite metabolique
L'etude moleculaire des microorganismes a partir de la sous-unite ribosomale 16S peut se faire sur deux niveaux, soit l'ADNr 16S et l'ARNr 16S. L'utilisation de l'ARN est connue pour etre plus ardue puisque l'ARN est tres sensible a la degradation par les ribonucleases presentes dans les milieux. La duree de vie de l'ADN est superieure a celle de l'ARN. Effectivement, il a ete montre que l'ADN de bacteries mortes peut persister longtemps dans un milieu sans etre degrade (Aellen et al, 2006; Keer et Birch, 2003). Bien que l'ARNr possede une demi-vie plus longue que l'ARNm, l'ARNr peut tout de meme fournir une information de viabilite cellulaire et d'activite metabolique (Gourse et al, 1996; Ruimy et al, 1994). En effet, la presence de l'ARNr implique que l'organisme est actif, reagit a son milieu et y croit. Deja en 1966, Maaloe et Kjeldgaard avaient demontre que la regulation de la transcription des ARNr etait proportionnelle a la croissance cellulaire et depuis, plusieurs etudes l'ont cible dans les etudes de communautes bacteriennes actives (Felske et al, 1997; Morgan et al, 2002).
1.5.2. L'ARNr et les genes fonctionnels
Puisque la majorite des etudes sur les microorganismes environnementaux se font a partir du gene de l'ARNr 16S, l'information disponible dans les banques de donnees telle que GenBank concerne majoritairement les sequences de ce gene. Le Ribosomal Database Project II (Cole et al, 2005) comporte aujourd'hui plus de 350 000 sequences d'ADNr 16S, ce qui peut influencer favorablement le choix d'une telle cible. Cependant, 1'identification des microorganismes peut se faire aussi par des genes plus specifiques au role que joue une bacterie dans son milieu. Par exemple, on peut cibler les bacteries capables de denitrification, permettant a 1'azote fixe de retourner dans 1'atmosphere. L'un des genes responsables de cette denitrification est le gene nirK. C'est ce qu'a utilise
l'equipe de Philippot et al. (2004) dans leurs travaux sur les bacteries denitrifiantes dans le sol. Leur PCR en temps reel etait lineaire et sensible, permettant la detection jusqu'a 102
copies du gene nirK. Contrairement a l'ARNr 16S, ce gene n'est present qu'en une seule copie dans le genome ce qui permet de correler directement le nombre de copies du gene au nombre de cellules. En clonant et sequencant les produits PCR obtenus, ils ont remarque qu'il y avait une forte similitude entre les genes nirK caracterises. Par contre, une analyse phylogenetique a montre que la majorite de ces clones n'etaient pas apparentes au gene
nirK des bacteries cultivables en laboratoire.
Dans le but de faire l'etude de methanogenes dans un site d'enfouissement des dechets, Luton et al. (2002) ont plutot cible un gene fonctionnel specifique aux methanogenes, soit le gene mcrA codant pour une enzyme, methyle coenzyme-M reductase, de la formation du methane unique aux methanogenes. Ils ont demontre que Putilisation de mcrA permettait la detection des cinq ordres connus de methanogenes chez les archaebacteries. De plus, une comparaison des resultats obtenus a partir de ce gene a ceux de l'ARNr 16S a montre que ces deux approches phylogenetiques s'equivalaient. Par contre, un probleme potentiel et non negligeable de cette detection avec le gene mcrA est le haut degre de conservation entre sa sequence et celle du gene mrtA present chez quelques especes de methanogenes. Consequemment, le risque de detection non specifique pourrait etre present. De plus, certains methanogenes possedent ces deux genes. Ainsi, les deux copies seraient detectees ce qui occasionnerait une surestimation de cette population (Friedrich, 2005). L'avantage de cibler un gene fonctionnel plutot que l'ADNr 16S est la formation de PARNm. Ainsi, en etudiant cet ARNm, on peut detecter les bacteries ayant une fonction particuliere et qui y participent activement dans le milieu etudie.
1.5.3. Methodes de biologie moleculaire
L'etude des populations presentes dans les echantillons environnementaux s'est grandement developpee depuis la venue des techniques moleculaires dans ce domaine. On peut separer ces techniques en deux grandes categories soit les profilages et les detections specifiques.
Les methodes de profilage se basant sur le gene de l'ARNr 16S permettent de faire l'etude des communautes bacteriennes des echantillons environnementaux simplement en faisant l'extraction directe du materiel genetique suivi d'une amplification PCR a partir d'amorces universelles. L'avantage principal de ces techniques est qu'une connaissance prealable de la communaute bacterienne n'est pas essentielle, ce qui s'avere tres utile. Cependant, les amplifications PCR peuvent aussi apporter des biais. Premierement, les amorces se voulant universelles ne peuvent l'etre totalement. De plus, l'activite de la polymerase peut etre modulee selon le contenu en bases GC de l'ADN (Sipos et ah, 2007). L'amplification exponentielle d'un melange de fragments d'ADN peut resulter en une distorsion des ratios entre les amplicons des differents ADNr 16S et ceux du materiel de depart. II s'agit alors de techniques semi-quantitatives. Finalement, bien que chaque « pic » ou « bande » obtenu represente theoriquement une sequence precise, il peut etre difficile de distinguer les profils biomoleculaires d'ecosystemes ayant une grande diversite (Dabert et ah, 2002).
Lorsqu'il s'agit de detection specifique, le bon choix des amorces et des sondes est primordial. Bien entendu, la detection specifique implique une connaissance minimale des microorganismes recherches. Le Ribosomal Database Project II (Cole et ah, 2005) represente un outil sur le Web tres pratique pour le design des amorces et des sondes ciblant le gene d'ARNr 16S. Elle comprend des milliers de sequences d'ADNr 16S de bacteries identifiees ou non, provenant de differents milieux environnementaux. Selon le niveau phylogenetique des groupes a etudier, les designs d'amorce peuvent etre laborieux. En effet, trouver des regions du gene specifiques au groupe a cibler tout en etant assez different du gene des autres organismes pour en eviter la detection n'est pas toujours aise. Song et ah (2004) ont voulu quantifier les Clostridia dans les feces humaines. Ce groupe de bacteries represente un bon exemple des difficultes rencontrees lors d'etudes sur les echantillons environnementaux. En effet, la detection au niveau de l'espece fut impossible due a la grande diversite de l'ecosysteme dans l'intestin humain. II est alors plus simple de faire la detection au niveau du genre bacterien. Malheureusement, le genre Clostridium est un genre entrelace, c'est-a-dire qu'il est extremement difficile de trouver des sequences assez specifiques pour tous ses membres sans inclure d'autres genres bacteriens. Done, pour detecter le maximum d'individus, ils ont du les separer par groupes a Pinterieur du genre (Song et ah, 2004). Ces auteurs ont ainsi utilise trois paires d'amorces specifiques
aux groupements des Clostridium (groupes I, XI et XlVab) avec une sonde TaqM&n commune, pour quantifier par PCR en temps reel.
1.5.3.1. LH-PCR
Le LH-PCR {length heterogeneity PCR) est une technique moleculaire de profilage basee sur Famplification PCR de FADNr 16S. Plus precisement, cette methode cible une region du gene qui possede une variabilite de longueur de sequence specifique a un organisme. Cette variabilite est due aux changements evolutifs de deletions et d'insertions de bases. On utilise alors des amorces universellement reconnues chez les bacteries qui bordent cette region variable. Ainsi, on obtient un amalgame d'amplicons, de differentes longueurs, associes a un microorganisme particulier. La detection des differentes longueurs d'amplicon se fait grace au fluorophore couple a une des deux amorces de PCR et qui est detecte par un laser lors de la migration sur sequenceur automatique. Cette technique est considered comme une methode simple et reproductible (Ritchie et al, 2000; Tiirola et al, 2003). Par contre, il n'existe pas de banque de donnees publique qui permette de faire directement F identification bacterienne des profils obtenus. En effet, pour ce faire, le sequencage demeure le moyen le plus sur. Le sequenceur automatique a capillaire est l'outil de choix puisqu'il permet une detection precise, nette et rapide des profils d'amplification. Cependant, il ne permet pas de faire un sequencage direct de chacun des fragments obtenus puisque ceux-ci ne peuvent etre physiquement isoles. Le LH-PCR a ete utilise dans plusieurs ecosystemes pour en evaluer la diversite bacterienne (Field et al, 2003; Mills et al, 2003; Ritchie et al, 2000; Suzuki et al, 1998; Tiirola et al, 2003; Yang et al, 2006). Cette technique n'a cependant pas encore ete utilisee pour etudier le lisier de pore et la digestion anaerobic
1.5.3.2. T-RFLP
Le T-RFLP {terminal restriction fragment length polymormism) est aussi une technique d'amplification PCR de FADNr 16S. Semblable au LH-PCR, il differe cependant par une ou plusieurs digestions enzymatiques des amplicons produisant des fragments de restriction detectables par la fluorescence. Puisqu'une seule des amorces (generalement
Pamorce sens 5') est liee au fluorophore, la notion de terminal provient du fait que seuls les fragments contenant l'extremite 5' fluorescent sont detectes. L'avantage de l'utilisation d'enzymes de restrictions est qu'elles permettent de differencier les amplicons de meme poids moleculaire. Le T-RFLP s'applique a tous les procaryotes et est aussi utilise dans Petude des archaebacteries (Banning et al, 2005; Collins et al, 2006; Lueders et Friedrich, 2002). Padmasiri et al. (2007) ont eu recours au T-RFLP afin de suivre la population methanogene dans un bioreacteur anaerobie traitant le lisier de pore. Les populations de Methanosarcinaceae et Methanosaetaceae obtenues etaient correles aux concentrations d'acetate produites.
Bien que l'emploi de plusieurs enzymes de restrictions permette une detection plus precise, l'analyse des profils obtenus devient plus ardue. L'analyse des profils de T-RFLP est possible par l'utilisation d'outils informatiques d'assignation de longueur de fragments.
Par exemple, TAP T-RFLP (http://rdp.cme.msu.edu) (Marsh et al, 2000), TORAST
(http://www.torast.com) and MiCA (http://mica.ibest.uidaho.edu/) (Kent et al, 2003) sont
disponibles sur internet. Ces outils permettent de simuler in silico cette technique de T-RFLP pour faire un choix eclaire de la combinaison optimale d'amorces et d'enzymes de restriction. Cette situation permet d'obtenir une meilleure resolution au niveau taxonomique desire, tel qu'effectue par Padmasiri et al. (2007). De plus, ces simulations peuvent etre utilisees pour predire les fragments de restriction et en faire 1'identification. Par contre, tout comme le LH-PCR, le clonage est necessaire pour permettre une identification convenable. Puisque l'electrophorese se fait generalement sur un sequenceur automatique, 1'excision des bandes est alors impossible. Une alternative interessante pour 1'identification rapide des profils obtenus est de creer une banque de reference de clones prealablement sequences et testes selon la technique de profilage utilisee.
1.5.3.3. DGGE
Le DGGE {Denaturing Gradient Gel Electrophoresis) ou l'electrophorese de gel denaturant a gradient, est aussi une technique permettant l'obtention de profils associes aux sequences de l'ADNr 16S. Se basant sur la difference de stabilite de l'ADN double brin inherent aux differences de leur composition en bases GC, cette technique permet la
separation des amplicons d'ADNr 16S lors de l'electrophorese sur un gel de polyacrylamide avec un gradient lineaire de denaturation de formamide/uree. La mobilite des molecules est reduite lorsque les deux brins se detachent partiellement. Une region riche en GC est ajoutee a l'extremite 5' d'une amorce ce qui previent la separation complete des brins. La popularity du DGGE s'explique par le fait que les fragments peuvent etre excises du gel, clones et sequences directement ou encore hybrides a des sondes specifiques. De plus, ce systeme est beaucoup moins couteux qu'un sequenceur automatique. Malheureusement, faire la distinction entre les bandes, est nettement plus difficile puisqu'il n'implique pas de detection au laser. De plus, la reproductibilite des gels entre eux et entre les laboratoires reste relativement faible (Dorigo et al, 2005). Dans l'etude des communautes microbiennes du lisier de pore, cette technique a ete effectuee avec succes par Topp et Leung (2001). En comparant un lisier traite dans des bioreacteurs anaerobies et aerobies, les patrons de bandes, semblables au depart, montraient une dominance des phylotypes specifiques aux aerobes lorsque celui-ci etait aere. Recemment, Cook et al. (2007) ont utilise le DGGE pour caracteriser les microorganismes responsables de la formation du scatole, compose malodorant, dans un lisier enrichi par son precurseur, le L-tryptophane. Les profils releves dans les echantillons riches en scatole etaient associes a une augmentation des bacteries gram-positives, faibles en GC, et des Bacteroides.
1.5.3.4. Hybridation de fluorescence in-situ (FISH)
L'hybridation de fluorescence in-situ (FISH), s'appuyant sur l'usage de sondes fluorescentes specifiques a l'ADNr 16S d'un seul microorganisme ou d'un groupe, a couramment ete realisee pour l'etude des populations microbiennes presentent dans les bioreacteurs (Sousa et al, 2007; Tang et al, 2004), de la microfiore intestinale et fecale (Franks et al, 1998; Lay et al, 2005; Martin-Orue et al, 2007) et des sols (Wellington et al, 2003). L'hybridation est effectuee sur des cellules entieres fixees. Ces dernieres sont alors observees par microscopie epifluorescente ou par microscopie confocale. Les resultats sont generalement exprimes par la proportion de microorganismes detectes par les sondes specifiques compares a la communaute microbienne totale detectee par des sondes universelles (Dabert et al, 2002). C'est une methode utile pour la determination de l'abondance de populations specifiques et par le fait meme, pour la quantification de ces
communautes. De plus, le FISH evite le biais potentiel du a 1'amplification par polymerase et permet de connaitre la distribution spatiale des cibles. Cependant, le choix de la sonde et du fluorophore, de meme que les conditions de l'astringence, la temperature d'hybridation, Pautofluorescence de certains organismes, le type d'ecosysteme et le stade physiologique de la cellule cible peuvent influencer significativement l'efficacite' du FISH (Dorigo et al, 2005). Aussi, la limite de detection est dans l'ordre de 105 copies d'ADN/ARN et la
sensibilite est reduite pour les milieux a haute concentration microbienne.
Peu d'etudes ont ete effectuees pour connaitre la microflore intestinale des pores. Martin-Orue et al. (2007) ont fait l'usage du FISH dans une etude visant a identifier les microorganismes presents dans tout le systeme digestif de pores traites avec des dietes fournissant differentes sources de fibres. Les resultats du FISH ont permis de constater d'importantes differences des populations selon quatre sites digestifs distincts mais non en fonction de leur diete. Alors qu'un T-RFLP sur ces memes echantillons a montre une difference de profils selon la diete, une mauvaise specificite des sondes ainsi qu'un changement des populations non ciblees peuvent expliquer ces resultats.
1.5.3.5. PCR en temps reel
Le PCR en temps reel est de plus en plus utilise en microbiologic environnementale. Cette technique tres sensible permet la quantification des especes, des populations bacteriennes autant que du total bacterien. La difference majeure entre le PCR en temps reel et le PCR traditionnel est que les donnees obtenues dans le premier correspondent au debut de
V amplification exponentielle de l'amplicon alors que dans le second, elles refletent la
quantite finale de l'amplicon qui peut n'avoir aucun lien avec la quantite de materiel presente au depart. En fait, le nombre de copies du gene cible est obtenu a partir du cycle PCR (Ct) ou la fluorescence detectee depasse une ligne de base, de meme que par l'utilisation d'une courbe d'etalonnage.
Deux technologies peuvent etre utilisees pour ce type de detection. La methode 7agMan se base sur la destruction d'une sonde placee entre deux amorces. Cette destruction emet alors une fluorescence detectable par l'appareil. La methode SYBR Green est basee sur la
presence d'un agent fluorescent qui s'intercale a l'ADN double brin. Consequemment, la detection est moins specifique puisque le fluorophore peut s'intercaler dans tout fragment amplifie. Le PCR en temps reel comporte de nombreux avantages. Tout d'abord, puisqu'il detecte chaque nouvelle amplification, la phase d'ampflification logarithmique fournit les donnees utilisees pour la quantification. Cela permet d'eviter les erreurs dues a l'efficacite de l'amplification ou a la diminution de reactifs. De plus, il ne comprend pas de manipulations apres le PCR, ce qui aide a prevenir la contamination entre les echantillons. Aussi, cette methode permet d'obtenir des resultats tres rapidement pour un grand nombre d'echantillons. Enfin, il ne faut pas oublier sa sensibilite qui est beaucoup plus grande que le PCR conventionnel. II existe aussi des desavantages a cette technique. Premierement, elle demande une mise au point des conditions reactionnelles (concentrations d'amorces et d'ADN a amplifier) et il faut s'assurer de la specificite du design d'amorces et/ou de la sonde. Par contre, une fois la mise au point effectuee, cette methode est tres rapide. Aussi, la sensibilite de la technique doit etre analysee. Cette analyse demande la presence d'un temoin interne exogene ou endogene pour normaliser les resultats (Sharkey et al, 2004), de merae que la determination de Pechelle de linearite de la courbe d'etalonnage afin de valider les resultats. De plus, pour faire du PCR en temps reel, le laboratoire doit etre equipe de l'appareil de detection specialise qui est couteux. A cela s'ajoute les couts des reactifs et enzymes necessaires a l'amplification. S'il s'agit de la methode TaqMan, la sonde fluorescente represente des couts additionnels mais la specificite est theoriquement amelioree.
Rousselon et al. (2004) ont utilise les sondes d'ADN fluorogeniques MGB {Minor Groove
Binder), derivees de la technologie TaqMm pour faire la detection de bacteries fecales
dans l'environnement. lis ont du identifier un indicateur de la contamination fecale base sur la dominance bacterienne dans les feces humaines. Par l'analyse comparative de phylotypes a l'interieur du groupe des bacteries Gram-positif a faible teneur en GC, un design d'amorces a ete cree, appele Feci. II s'est avere que l'utilisation de ce design a permis de quantifier et de tester positivement les echantillons de feces humaines et animales et negativement les echantillons de sols, d'eaux et de bioreacteurs presumes non contamines. Le SYBR Green a ete la methode choisie par Fey et al. (2004) dans la quantification de PADNr et de l'ARNr bacteriens. Grace a ce dernier, ils ont pu determiner
la presence et Pactivite metabolique d'une bacterie pathogene dans l'eau. lis arrivent a la conclusion que leur technique peut etre utilisee dans la quantification bacterienne d'6chantillons environnementaux ainsi que dans la determination de leur niveau d'activite. De plus, en choisissant des cibles appropriees, il est possible d'etudier la situation physiologique ou le potentiel pathogenique de diverses populations bacteriennes. Les archaebacteries ont aussi ete quantifies par PCR en temps reel (Sawayama et ah, 2006; Shigematsu et ah, 2003; Stewart et ah, 2006). Yu et ah (2006) ont utilise la technologie
TaqMan pour la detection et la quantification de methanogenes acetotrophes dans
differents systemes de digestion anaerobie. lis ont recouru a des amorces et des sondes ciblant distinctement PADNr 16S de l'ordre Methanosarcinales et des families
Methanosarcinaceae et Methanosaetaceae.
1.6. Approche experimentale
L'objectif du projet presente dans ce memoire consistait a developper des outils moleculaires afin de determiner les niveaux d'activite des groupes bacteriens presents dans le lisier de pore. Les odeurs provenant du lisier sont produites lors de la degradation anaerobie de la matiere organique par des microorganismes indigenes. Puisque la microbiologic classique ne permet la detection et 1'identification que d'une faible proportion des populations microbiennes environnementales, des techniques de biologie moleculaire ont ete utilisees. Differents lisiers ont ete analyses; soit du lisier brut recueilli directement a la ferme, du lisier entrepose ainsi que du lisier en traitement dans un bioreacteur anaerobie a ecoulement piston.
Differents protocoles d'extraction du materiel genetique ont ete eprouves dans le lisier de pore. Suite a quelques modifications, il a ete determine que la co-extraction de l'ADN et de l'ARN, selon la methode de Griffiths et ah (2000), permettait d'obtenir un materiel de meilleure qualite tout en etant simple, rapide et peu couteuse. Afin de faire la detection des populations microbiennes actives, l'etude a ete faite en ciblant l'ARNr 16S en plus de l'ADNr 16S.
Dans le but d'etudier les changements de la communaute bacterienne dans un bioreacteur en fonction, les techniques moleculaires de LH-PCR et de T-RFLP ont ete utilisees afin de suivre les bacteries et les archaebacteries sur une periode de six mois. Ce bioreacteur etait compose de huit compartiments relies en serie et munie d'une pompe manuelle situee a l'entree de meme que d'une sortie de Peffluent a son extremite. De la periode de demarrage a six mois d'utilisation, les compartiments ont vu leur composition microbienne se preciser et se specialiser selon les differentes etapes de la degradation anaerobie.
Pour correler les resultats a la production des odeurs, Paccumulation d'acides gras volatils a ete quantified par chromatographie en phase gazeuse puisque ces acides gras volatils sont considered comme etant des indicateurs d'odeur adequats. De plus, l'efficacite du bioreacteur a pu etre suivie par la production du biogaz, produit final de la degradation anaerobie.
Des essais de PCR en temps reel ont aussi ete effectues sur les echantillons de lisier provenant du meme bioreacteur a ecoulement piston apres les 6 mois d'operation. De nouvelles extractions d'ARNr 16S ont ete realisees en inoculant chaque echantillon d'une quantite connue d'une bacterie de reference Thermus aquaticus. La quantification du nombre de copies d'ARNr 16S a ete obtenue a partir des designs specifiques aux genres Clostridium coccoides, Syntrophomonas, Peptostreptococcus, Methanosarcina et Methanosaeta.
Lors d'un autre volet du projet, l'analyse de profits de LH-PCR et T-RFLP sur des lisiers de pores en finition, preleves de la fosse a lisier d'une ferme estrienne ainsi que d'un melange de lisiers de pores en finition et de maternite utilise pour l'alimentation de bioreacteur, a ete effectuee pour observer les differences microbiologiques qui existaient entre eux.
Puisque les techniques moleculaires utilisees produisent de nombreuses donnees, l'analyse des profils a ete facilitee par divers outils statistiques. L'analyse en composante principale (ACP) et le positionnement multidimensionnel non-metrique (NMS) ont ete utilises pour visualiser les differences de profil entre les echantillons de lisier. L'ACP a pour but