Différenciation génétique et sélection chez le cerf de
Virginie (Odocoileus virginianus) introduit à l’île
d’Anticosti.
Mémoire
Jérémie Fuller
Maîtrise en biologie
Maître ès sciences (M. Sc.)
Québec, Canada
© Jérémie Fuller, 2018
Différenciation génétique et sélection chez le cerf de
Virginie (Odocoileus virginianus) introduit à l’île
d’Anticosti.
Mémoire
Jérémie Fuller
Sous la direction de :
Steeve D. Côté, directeur de recherche
Louis Bernatchez, codirecteur de recherche
iii
Résumé
L’introduction d’une espèce en milieu isolé est souvent associée à une perte de la variation génétique, c’est l’effet fondateur. Les pressions de sélection peuvent également différer du milieu d’origine, accélérant la divergence de la population introduite. En 1896, environ 220 cerfs de Virginie (Odocoileus virginianus) provenant de la rive sud du fleuve Saint-Laurent ont été introduits sur l’île d’Anticosti (Québec, Canada) pour développer la chasse sportive. Cette population a rapidement atteint de fortes densités (>20 cerfs/km2) et présente une taille corporelle inférieure à celle de la
population source. Ce projet avait comme objectifs d’évaluer le rôle de l’effet fondateur et de la sélection sur la diversité et la structure génétique de la population de cerfs de l’île d’Anticosti. Pour ce faire, la variabilité génétique de cette population a été comparée à la population souche avec des techniques de séquençage à haut débit (GBS). Les résultats montrent une faible différenciation génétique entre la population source et celle de l’île d’Anticosti par rapport à l’ampleur de la différenciation génétique entre les rives nord et sud du Saint-Laurent. L’absence de structuration à l’intérieur de l’île d’Anticosti signifie que la dérive génétique n’a pas eu d’effet significatif lors de la colonisation de l’ensemble de l’île. Ainsi, la grande diversité génétique de la population de cerfs d’Anticosti et sa faible différenciation par rapport à celle du continent suggèrent un faible effet fondateur. Le grand nombre d’individus introduits et la rapide croissance démographique de la population ont sans aucun doute contribué à diminuer la dérive génétique et expliquent le maintien d’une grande diversité génétique. De plus, des loci potentiellement sous sélection ont été associés aux traits phénotypiques précédemment identifiés comme différents entre les deux populations. Ces loci appuient une divergence adaptative de la population insulaire en réponse aux conditions particulières de l’île d’Anticosti.
iv
Abstract
The introduction of a species is often associated with a loss of genetic variability, referred as the founder effect. Different selection pressures in the colonized environment may also accelerate the genetic divergence of two populations. In 1896, ca. 220 white-tailed deer (Odocoileus virginianus) captured on the southern shore of the St. Lawrence River were introduced on Anticosti Island (Québec, Canada) to develop sport hunting. The absence of predator allowed the population to quickly reached high densities (>20 deer/km2) that affected forest composition because of browsing intensity. Deer body size
decreased by half compared to the source population because of their poor-quality diet. Our objectives were to characterize the impacts of the founder effect and selection on the diversity and genetic structure of the deer population on Anticosti Island. We compared the genetic variability of this population to one of the strain populations (Montmagny-L’Islet region) with high throughput sequencing techniques (GBS). The results show a small, albeit significant genetic differentiation, between the source population and Anticosti Island relative to the extent of genetic differentiation between the north and south shores of the St. Lawrence. We found a lack of structuration within the island meaning that the genetic drift did not have a significant magnitude during the colonization of the whole island. The high genetic diversity of the Anticosti deer population and its low differentiation from the mainland suggests a weak founder effect. The large number of introduced individuals and the rapid population growth of the population have undoubtedly helped to reduce genetic drift and explain the maintenance of a great genetic diversity. Loci under putative selection were assigned to the differentiated phenotypic traits supporting an adaptive divergence of the island population.
v
Table des matières
Résumé ... iii
Abstract ... iv
Liste des tableaux ... vii
Liste des figures ... viii
Remerciements ... ix
Avant-propos ... xii
Introduction ... 1
Les mécanismes évolutifs ... 1
Adaptation locale : sélection et plasticité phénotypique ... 2
L’effet fondateur ... 3
Les systèmes insulaires ... 5
Le cerf de Virginie ... 7
Biologie ... 7
Structure génétique des populations ... 8
Le cas à l’étude : les cerfs introduits sur l’île d’Anticosti ... 10
Objectifs et hypothèses ... 11
Approche méthodologique... 12
Chapitre 1 Weak founder effect and evidence for adaptive divergence in a recently introduced insular population of white-tailed deer (Odocoileus virginianus) ... 14
Résumé ... 15
Abstract ... 16
Introduction ... 17
Material and Methods ... 19
Study area ... 19
Sampling and phenotypic measurements ... 21
Genomic analyses ... 21
Results ... 27
SNP calling ... 27
Clustering analysis ... 27
Genetic structure and population assignment ... 28
vi
Phenotype-genotype associations ... 31
Genetic basis of phenotypic differentiation ... 32
Discussion ... 34
Genetic differentiation and structure of Anticosti Island deer ... 34
Weak founder effect ... 36
Adaptation on the Island ... 37
Limitations ... 40
Management Implications ... 41
Acknowledgements ... 42
Conclusion générale... 43
Différenciation génétique ... 43
Absence de structuration génétique ... 44
Adaptation locale de la population ... 45
Taille de population efficace et sélection sexuelle ... 46
Implications pour la gestion ... 48
Forces et limites de l’étude ... 50
Perspective de recherche ... 51
Références ... 53
vii
Liste des tableaux
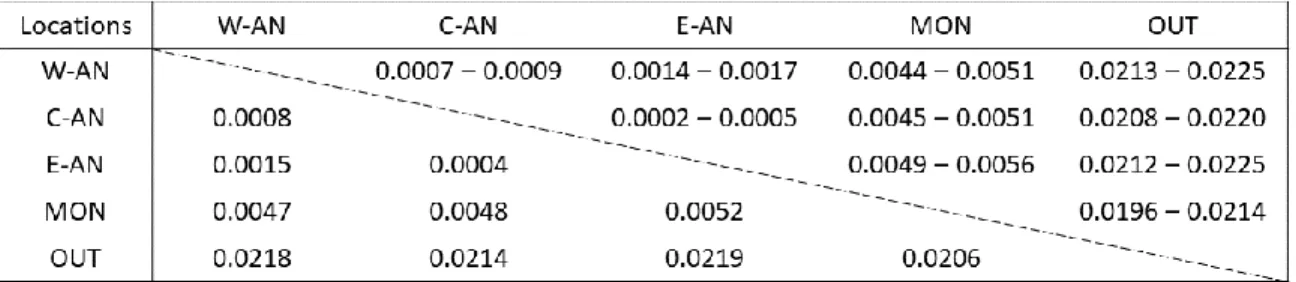
Table 1. Below main diagonal: Fixation index (FST) obtained with 8,518 SNPs between populations of white-tailed deer from western Anticosti Island (W-AN), central Anticosti Island (C-AN), eastern Anticosti Island (E-AN), Montmagny (MON) and Outaouais
(OUT); above main diagonal: 95% confidence intervals. ... 29 Table 2. Descriptive genetic statistics of white-tailed deer populations obtained with 8,518 SNPs: observed heterozygosity (Ho), expected heterozygosity (He), inbreeding coefficients (Gis and F), nucleotidic richness (π), number of polymorphic loci (N), effective population size (Ne), census population size (Nc) for Anticosti Island (ANT), Montmagny (MON) and Outaouais (OUT) populations. ... 30
viii
Liste des figures
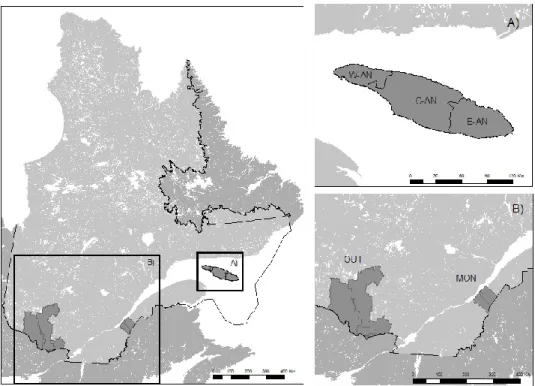
Figure 1. The three study areas (Québec, Canada). A) Anticosti Island divided into three regions: western (W-AN), central (C-AN), and eastern (E-AN). B) Montmagny-L’Islet (MON) and Outaouais (OUT)... 20 Figure 2. Discriminant analysis of principal components (DAPC) of the genetic variation of white-tailed deer without prior information on their group provenance: Western
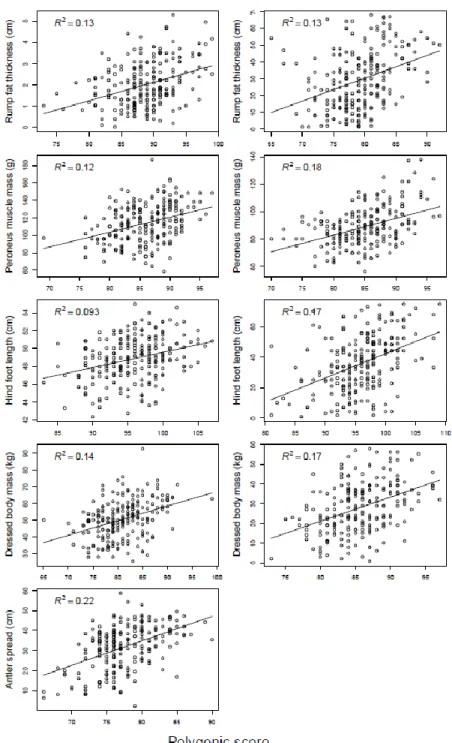
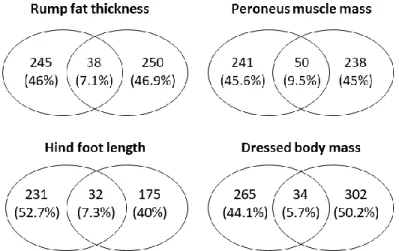
Anticosti (W-AN), Central Anticosti (C-AN), Eastern Anticosti (E-AN), Montmagny (MON) and Outaouais (OUT). ... 28 Figure 3. Venn diagrams of outliers detected by OutFLANK and pcadapt between white-tailed deer from Anticosti Island and Montmagny (ANT&MON), and between Anticosti Island and Outaouais (ANT&OUT). ... 30 Figure 4. Venn diagrams of loci, detected by LFMM, associated with rump fat thickness, peroneus muscle mass, hind foot length and dressed body mass of male (A) and female (B) white-tailed deer from Anticosti Island. ... 31 Figure 5. Correlations between the polygenic score and the corresponding phenotypic trait (rump fat thickness, peroneus muscle mass, hind foot length, dressed body mass, and antler spread) for male (Left) and female (Right) white-tailed deer from Anticosti Island. All correlations were significant (p ˂ 0.001). ... 33 Figure 6. (A) Venn diagram of loci found in common by pcadapt analyses between Anticosti Island and both mainland sites (Montmagny and Outaouais), compared to all loci found by random forest and LFMM analyses. (B) Venn diagram of loci found by all genome scan approaches (OutFLANK and pcadapt) among populations compared to all loci found by random forest and LFMM analyses. ... 34
ix
Remerciements
Lorsque j’ai entendu parler du projet sur les cerfs de l’île d’Anticosti, j’ai su qu’il était fait pour moi. Depuis toujours les îles m’ont fasciné par leur caractère unique. Les aventures d’Alfred Russel Wallace et de Charles Darwin m’ont particulièrement émerveillé durant mes études. Ce fut pour moi un réel privilège de travailler sur ce projet longuement attendu.
Je tiens tout d’abord à remercier mon codirecteur, Louis Bernatchez, d’avoir eu confiance en moi et de m’avoir donné la chance de faire partie de son laboratoire. J’ai eu aussi le plaisir de te connaître en Équateur et sur les îles Galapagos! Mythique! Nous avons eu de grandes discussions, dont je garde un merveilleux souvenir. Je te remercie pour toute l’aide que tu m’as apportée. Tu t’es également assuré que mon projet de maîtrise me convenait en m’offrant la possibilité de travailler sur les mammifères. Je remercie mon directeur, Steeve Côté, qui m’a accepté dans son laboratoire alors que je n’étais qu’un inconnu. Je n’ai pas eu la chance d’assister à l’un de tes cours durant mon baccalauréat, mais heureusement j’aurai pu bénéficier de ton enseignement à titre de directeur. J’ai grandement apprécié ta rigueur, tes conseils et tes nombreuses histoires. Merci pour tout. Je remercie également Julie Turgeon et Jean-Pierre Tremblay d’avoir accepté de réviser mon mémoire.
Ce projet n’aurait pas eu la même envergure sans les professionnels de recherche que j’ai côtoyés durant ces années. Pour le labo Côté-Tremblay, je tiens à remercier Sonia de Bellefeuille, qui m’a grandement aidé avec les bases de données, Caroline Hins pour tes précieux conseils et Julien Hénault-Richard pour la relecture de mes travaux. Pour le labo Bernatchez, un immense merci à Cécilia Hernandez, Guillaume Côté et Damien Boivin-Delisle de m’avoir aidé à (finalement) extraire de l’ADN de bonne qualité, et bien sûr merci à Éric Normandeau pour son soutien en bioinformatique et nos conversations en matière d’art martial.
Je tiens à souligner le soutien financier fortement apprécié de la Chaire de recherche industrielle CRSNG en aménagement intégré des ressources de l’île d’Anticosti et de ses partenaires. Merci également à la Fédération québécoise des chasseurs et pêcheurs (FédéCP) et à la fondation Héritage faune de m’avoir décerné la bourse Aurèle-Blais en aménagement de la faune.
Je remercie les gens de l’île d’Anticosti, sans qui mon terrain n’aurait pas été aussi mémorable. Alex/Gaétan Laprise et Danièle Morin, un gros merci pour votre aide et pour le séjour de chasse! Aimée Benoist-Chénier et Stefan Tremblay, merci beaucoup pour votre accueil et votre soutien dans mes péripéties de terrain! Je remercie tous ceux et celles qui ont, de près ou de loin, participé à la récolte des échantillons pour l’ensemble du projet, notamment Alexandre Lottinville, technicien pour la chaire de recherche à l’automne 2015; Michaël Bonin et ses assistant(e)s pour les échantillons de Montmagny; Vicky Albert et l’équipe du MFFP du laboratoire d’expertise biolégale ainsi qu’André
x
Dumont et la direction régionale de l’Outaouais du bureau de Gatineau pour les échantillons d’Outaouais.
Une maîtrise, ce n’est pas uniquement du boulot, c’est aussi des rencontres inoubliables! J’ai eu la chance de côtoyer non pas un laboratoire, mais deux! Et pas parmi les plus petits.
Au labo Côté-Tremblay : Mael, Mathieu, Julien, Andréa, Karina, Brett, Amé, Pat, Ed, Pascale, Max, Dalie, Clara, Sab, Émilie, Mic, Barb, Antoine, Myriam, Milène, Nico, Béa, Flo, Laurent, Fred, Alexis (pour les pommes de 15 h), Linda, Delphine, Ève et Sophiane. Merci gang pour votre aide et pour ses rires partagés, ces bières du jeudi, ces soirées inoubliables, et cheers aux nombreux fans de Pérusse (#Fruitdemer). Vous êtes des personnes extraordinaires et je vous souhaite bonne chance dans vos projets. Un merci spécial à Marianne, ma partenaire généticienne en ce monde d’écologistes! Que de beaux souvenirs en C.B.! Merci pour ton écoute et ta sensibilité. Je te souhaite le meilleur, mon amie.
Au labo Bernatchez : J.-S., Anne-Laure, Martin, Jérémy, Ben, Hugo, Claire, Anaïs, Justine, Noé, les nombreuses Anne, Damien, Clément, Laura, Yann, Quentin, Bérénice, Lucie, Maëlle, Alysse, Simon, Charles, Émilie, Gratien, Olivier, Amanda, Madoka, Henrik et Caro. Merci pour ces discussions enrichissantes, pour votre aide indispensable et pour ces soirées qui n’en finissent plus (#workhardplayharder). Réaliser ma maîtrise avec vous fut un privilège et une fierté. Je vous souhaite du bonheur et le succès que vous méritez.
Merci également aux collègues du Vachon : Yannick, Mathilde, Guillaume, Fred, Dom, Isa, Marc-André, Marianne et Audrey-jade ainsi qu’à mes collègues de l’IBIS : F-E (aka Francisco de Padre!), F.-O., Laurence, Jeff, Bachar, Pier-Luc vous côtoyer au quotidien fut un grand plaisir.
C’est également important d’être bien entouré à l’extérieur de l’université. Je tiens donc à remercier mes ami(e)s, dont ma gang des Zozios pour votre soutien et nos soirées mémorables. M. Brousseau! même si tu as été nommé dans la précédente section, tu sais bien que tu pourrais avoir une section juste pour toi, bro! Apprendre à te connaître, surtout à titre de coloc, fut un privilège. Merci Max d’être toujours là pour moi et de me remonter le moral quand j’en ai besoin, t’es plus qu’un ami et j’espère que l’on gardera contact malgré le temps. Ma maîtrise n’aurait certainement pas été la même sans toi. Anne, toi aussi tu mérites d’avoir une section à part. Merci chère amie pour tes conseils et ton énergie contagieuse. Je te souhaite bonne chance dans tes projets. Mélina, ma grande amie, merci pour ton soutien au fil de toutes ses années. Bien sûr, Gab, la meilleure annonce Kijiji! Thanks buddy de me changer les idées avec nos journées/soirées jeux de société. Je remercie mon club de Tai Jitsu, où j’ai découvert une seconde passion, et un merci spécial à Marie-Eve Castonguay, qui a accepté de réviser mon mémoire.
À ma famille, qui a toujours cru en moi et m’a encouragé vers les études de cycles supérieurs, je vous aime. Audrée, merci pour ton soutien et ta compréhension, mais surtout merci de faire partie de ma vie. Un merci spécial à mes grands-parents, qui
xi
m’ont transmis leur passion pour la nature, me dirigeant de ce fait vers la biologie. Mom, merci d’être toujours là pour moi.
Pour tous les chasseurs et chasseuses qui ont cru en l’existence du cerf Menier, voici une histoire qui saura, je l’espère, alimenter vos discussions!
Bonne lecture.
‘’Nothing in biology makes sense except in the light of evolution’’ - Theodosius Dobzhansky
‘’Why do we fall? So that we can learn to pick ourselves up’’ - Thomas Wayne
xii
Avant-propos
Ce projet de maîtrise porte sur la différenciation génétique du cerf de Virginie introduit sur l’île d’Anticosti (Québec, Canada), une étude réalisée dans le cadre de la Chaire de recherche industrielle CRSNG en aménagement intégré des ressources de l’île d’Anticosti. Le mémoire débute par une introduction générale sur les mécanismes évolutifs qui agissent sur la divergence des populations, particulièrement en situation insulaire. Le cœur de ce mémoire consiste en un article scientifique (manuscrit) rédigé en anglais et qui a été soumis au journal scientifique Molecular Ecology le 22 juin 2018. Le mémoire se termine par une conclusion générale qui résume la contribution de mon étude au savoir scientifique et où j’énonce quelques idées de recherche soulevées par mes résultats.
En tant que premier auteur, mes responsabilités consistaient à sélectionner les échantillons parmi la collection de la chaire de recherche, à participer à la collecte de données sur le terrain à l’automne 2015, à réaliser les manipulations de laboratoire, à effectuer les analyses et l’interprétation des résultats ainsi qu’à rédiger l’article.
Les coauteurs sont Anne-Laure Ferchaud, Martin Laporte, Jérémy Le Luyer et Theodore B. Davis, mon directeur, Steeve D. Côté, et mon codirecteur, Louis Bernatchez.
Anne-Laure Ferchaud, Martin Laporte et Jérémy Le Luyer, tous postdoctorants, ont contribué aux analyses et à l’interprétation des résultats. Ils ont également participé à la révision de l’article.
Theodore B. Davis nous a fourni le génome du cerf de Virginie avant sa publication et a contribué à l’amélioration du manuscrit.
L’interprétation des résultats et la rédaction du manuscrit ont été supervisées par Louis Bernatchez et Steeve D. Côté. Ce dernier a proposé et financé l’étude. Louis Bernatchez a également contribué à une partie du financement.
1
Introduction
Les mécanismes évolutifs
La diversité biologique actuelle découle de l’évolution durant des milliers d’années des premiers microorganismes et, plus tard, des organismes multicellulaires complexes. Cette histoire a été forgée par quatre forces évolutives : la mutation, la migration, la dérive génétique et la sélection (Allendorf & Luikart 2007). La mutation induit de la diversité génétique sur une échelle de temps beaucoup plus longue que les autres forces. En effet, les taux de mutation sont en général assez faibles, soit de l’ordre de 10-4 allant jusqu’à 10-9 mutations/locus/génération selon les régions du génome
(Brumfield et al. 2003). À l’opposé, le déplacement d’individus entre populations, la migration (dispersion au sens écologique), induit un flux génétique entre ces populations et provoque une homogénéisation des fréquences alléliques entre elles (Lowe & Allendorf 2010). Inversement, l’isolation d’une population favorise sa divergence. De son côté, la dérive augmente la différenciation entre les groupes de manière stochastique provenant de la pige aléatoire des gamètes lors des événements de reproduction (Frankham 1995, Höglund 2009). Les allèles retenus ne représentent pas parfaitement la fréquence allélique de la génération précédente, ce qui résulte ultimement en une perte de diversité allélique (Charlesworth 2003). Ce phénomène est amplifié dans les populations où la taille efficace est restreinte. Ces trois forces évolutives affectent l’ensemble du génome de manière aléatoire et sont donc décrites comme « neutres », contrairement à la sélection qui agit sur certains loci en réponse aux pressions environnementales. À noter qu’il existe différents types de sélection : directionnelle, balancée, divergente et sexuelle (voir la section Adaptation locale : sélection et plasticité phénotypique).
Ces quatre forces évolutives figurent parmi les responsables des différences génétiques, morphologiques et comportementales entre les populations d’une même espèce. Parmi les nombreuses branches de la biologie, l’une tente de comprendre l’influence de ces forces évolutives sur le vivant : la biologie évolutive. Plus active que jamais, elle a bénéficié de l’essor des nouvelles technologies pour apporter plus de précisions aux questions posées depuis plusieurs années.
2
Adaptation locale : sélection et plasticité phénotypique
À l’inverse des trois forces neutres, la sélection agit directement sur le phénotype des individus, qui est déterminé par l’interaction de certains loci avec l’environnement. Ainsi, les allèles offrant un avantage sur la reproduction ou la survie d’un individu sont favorisés et sont, en théorie, présents en plus grande proportion dans une population. La fixation d’allèles bénéfiques et la disparition d’allèles délétères contribuent à la différenciation entre deux groupes soumis à différentes pressions de sélection (Kawecki & Ebert 2004, Allendorf & Luikart 2007, Nosil et al. 2009).
Une espèce fait face à différentes conditions environnementales dans son aire de répartition, ce qui occasionne une hétérogénéité de pressions de sélection dans l’espace. Les individus de différentes populations développent alors des adaptations locales, soit des changements génétiques associés à leur environnement en réponse à la sélection divergente (Le Corre & Kremer 2012, Savolainen et al. 2013). L’apparition d’adaptations locales est facilitée en absence de flux de gènes et de dérive génétique, car ces processus peuvent compromettre les génotypes favorisés par la sélection (Kawecki & Ebert 2004, Blanquart et al. 2013). La définition classique soutient que la survie des individus localement adaptés est inférieure s’ils se retrouvent dans un nouveau milieu (Kawecki & Ebert 2004). D’autres types de sélection sont également possibles. La sélection balancée, ou stabilisatrice, entraîne une augmentation de la diversité génétique par l’action de différents mécanismes, dont l’avantage de l’hétérozygote (Hansson & Westerberg 2002, Charlesworth 2006, Llaurens et al. 2017). La sélection sexuelle dépend de la compétition survenant lors du choix des partenaires et peut agir à l’encontre de la sélection naturelle. Elle se produit lorsqu’un individu augmente son succès reproducteur grâce à un caractère sexuel secondaire, même si ce caractère peur compromettre sa survie (Clutton-Brock 2017). Les changements phénotypiques issus d’un changement génétique ne sont pas uniquement causés par la sélection : la dérive peut également être responsable d’une différenciation phénotypique (Jordan & Snell 2008, Kolbe et al. 2012, Spurgin et al. 2014). Les forces évolutives neutres ont un impact aussi significatif que la sélection sur les populations et peuvent ainsi être les précurseurs à la différenciation et ultimement à la spéciation.
3
L’expression d’un phénotype différent n’implique pas nécessairement un changement génétique, car il est également déterminé par l’environnement. La plasticité phénotypique est définie comme l’expression de traits phénotypiques différents par des organismes possédant un même génotype, mais situés dans des environnements différents (Pigliucci et al. 2006). Par exemple, une population de lézard, l’anole brun (Anolis sagrei), provenant de la Floride, fut introduite sur deux îles aux conditions abiotiques fort différentes, et les individus de ces deux nouvelles populations ont rapidement divergé en taille (Campbell & Echternacht 2003). La plasticité a un rôle à jouer dans l’établissement d’une population dans un nouveau milieu, car elle détermine la capacité d’acclimatation d’un individu(Price et al. 2003, Uller & Leimu 2011). Les espèces qui font face à des conditions climatiques très variées, par exemple les espèces migratrices ou nordiques, sont aussi avantagées par une capacité à exprimer une plasticité de leurs traits.
On parlera d’adaptation lorsque le changement phénotypique se conserve à l’échelle de la population. La plasticité phénotypique peut être un trait sélectionné, car elle peut offrir un avantage à l’individu (Schmid & Guillaume 2017). L’adaptation à un environnement peut toutefois être le résultat de la plasticité phénotypique combinée à la différenciation génétique des traits adaptatifs (Sultan & Spencer 2002, Volis et al. 2015, Schmid & Guillaume 2017). Par exemple, les printemps hâtifs causés par les changements climatiques ont entraîné en 10 ans une date de mise bas précoce de 18 jours chez une population d’écureuil roux (Tamiasciurus hudsonicus) au Yukon (Réale et al. 2003). Ce trait héritable est également connu pour varier selon les conditions environnementales, dont l’abondance de nourriture (Réale et al. 2003).
L’effet fondateur
Une introduction d’espèce naturelle ou par l’intermédiaire de l’homme peut occasionner une divergence génétique marquée par rapport à la population source (Clegg et al. 2002, Kolbe et al. 2012). Ce phénomène, appelé effet fondateur, est un cas particulier de la dérive génétique (Mayr 1954). C’est l’un des processus les plus forts, avec les goulots d’étranglement (réduction drastique de la population), qui peut rapidement engendrer une divergence génétique entre deux populations à la suite d’une perte importante de la variation génétique et d’un brusque changement des fréquences
4
alléliques (Nei et al. 1975, Allendorf 1986, Clegg et al. 2002). L’intensité de l’effet fondateur varie selon le nombre d’événements d’introduction, le nombre d’individus introduits, la diversité génétique de la population source ainsi que le temps nécessaire pour que la population augmente en nombre (Clegg et al. 2002, Blanchong et al. 2013, Murphy et al. 2015). Ainsi les espèces avec un taux de croissance intrinsèque plus faible (ex. les grands mammifères) devraient perdre davantage de variation génétique que les espèces avec des taux de croissance plus élevés (ex. les insectes et les petits mammifères), car ces dernières nécessitent généralement moins de temps pour augmenter en nombre (Allendorf & Luikart 2007). À la suite d’un effet fondateur ou d’un goulot d’étranglement, la perte de variation génétique sera plus accentuée pour la richesse allélique que l’hétérozygotie, car elle est plus sensible à la perte d’allèles rares (Nei et al. 1975, Dlugosch & Parker 2008). La richesse allélique, soit le nombre d’allèles à un locus, est plus importante pour le potentiel adaptatif à long terme d’une population, car elle témoigne de la diversité sur laquelle la sélection peut opérer (Allendorf 1986, Caballero & Garcia-Dorado 2013, Greenbaum et al. 2014).
La taille de la population, ou plus exactement le nombre réel d’individus qui contribuent à la génération suivante, est un paramètre important pour la dérive génétique. Cela fut conceptualisé par la taille de population efficace (Ne) qui est définie comme l’effectif d’une population idéale, c’est-à-dire qui est soumise aux mêmes taux de dérive génétique et de consanguinité que la population étudiée (Wright 1931). Ainsi, il s’agit d’un paramètre difficile à estimer, car plusieurs facteurs peuvent l’influencer, le ratio des sexes, le succès reproducteur des individus et la démographie de la population par exemple (Frankham 1995, Kalinowski & Waples 2002, Hedrick 2005, Palstra & Ruzzante 2008). Les populations de grande taille efficace sont moins propices à la dérive génétique, ce qui accentue le rôle de la sélection sur la fréquence allélique (Hartl & Clark 1997, Blanquart et al. 2013).
Dans les petites populations, le choix du partenaire est réduit et le risque de reproduction entre individus apparentés augmente. Ainsi, les taux de consanguinité sont appelés à augmenter, ce qui diminue la variabilité génétique, indépendamment de la dérive. Une diversité génétique faible est généralement associée à une moins bonne valeur adaptative des individus (Reed & Frankham 2003, Wood et al. 2016). Cette baisse de valeur sélective nommée dépression de consanguinité s’explique par
5
l’augmentation de l’homozygotie au détriment de l’hétérozygotie sur l’ensemble du génome (Keller & Waller 2002, Allendorf & Luikart 2007). C’est pourquoi des allèles récessifs délétères, maintenus normalement à une fréquence faible par la sélection, sont statistiquement plus sujets à se trouver en état d’homozygotie dans les populations consanguines (Keller & Waller 2002, Allendorf & Luikart 2007). Ces allèles récessifs seront alors exprimés et diminuent la valeur sélective de l’individu homozygote compte tenu de leurs effets délétères. Par exemple, une population de cerfs rouges (Cervus elaphus) isolée au nord de l’Allemagne présente aujourd’hui une forte tendance à la brachygnathie, soit une réduction de la mâchoire, à la suite d’une dépression de consanguinité (Zachos et al. 2007).
Un cas spécifique de la consanguinité peut survenir lors d’une introduction ou d’une colonisation. Comme le nombre d’individus fondateurs est limité, un allèle délétère porté par un individu aura statistiquement plus de possibilités de se trouver en forte fréquence malgré sa valeur contre-sélective (Keller & Waller 2002, Casellas et al. 2009). Cela augmente la probabilité de dépression consanguine dans la nouvelle population et peut menacer la viabilité de celle-ci. C’est pourquoi les populations de taille plus restreinte sont généralement sujettes à une attention et à des efforts de conservation plus intensifs. Le maintien de certaines populations nécessite parfois un sauvetage génétique qui consiste à ajouter des individus d’une autre population pour augmenter la variation génétique (Johnson et al. 2010, Whiteley et al. 2015).
Les systèmes insulaires
Les îles ont toujours fasciné les biologistes, particulièrement pour la présence d’organismes ayant une morphologie différente de leurs homologues continentaux. La tendance morphologique généralement observée sur les îles est une augmentation de la taille des petites espèces introduites (ex. lézards et rongeurs) et une réduction de la taille des grandes espèces (ex. éléphants et cervidés) (Meiri et al. 2008). VanValen (1973) a nommé ce phénomène « règle insulaire » et plusieurs méta-analyses ont tenté d’en déterminer les causes par la phylogénie, les interactions biotiques (compétition et prédation) ou les caractéristiques des îles (Lomolino 2005, Raia & Meiri 2006, Meiri et al. 2008, McClain et al. 2013). Tout semble indiquer que ce changement de taille est influencé par la combinaison de ces différents facteurs, dont l’importance varie
6
largement selon le contexte (Durst & Roth 2012, Lomolino et al. 2012). Bien que certains clades soient sur-représentées dans ces analyses en raison du grand nombre d’introductions, il a été établi que les artiodactyles, les carnivores et les rongeurs sont plus prédisposés évolutivement à répondre à cette règle pour des raisons encore inconnues (Meiri et al. 2008, Raia & Meiri 2011). Par rapport aux carnivores, les herbivores ont tendance à diminuer davantage en taille en absence de compétiteurs hétérospécifiques et de prédateurs (Raia & Meiri 2006).
Les îles sont reconnues pour présenter des conditions environnementales différentes des continents, en raison entre autres de la proximité de l’eau, qui exerce des pressions de sélection divergentes sur les espèces (Weigelt et al. 2013). Les espèces végétales sont les premières à répondre à ces changements environnementaux, ce qui modifie la composition végétale et offre par conséquent des ressources différentes aux consommateurs primaires (Agashe et al. 2011, Weigelt et al. 2013). Ainsi, les îles moins productives sont généralement occupées par des espèces de petite taille (McClain et al. 2013). La compétition pour les ressources y est souvent supérieure en raison de la superficie limitée, ce qui favorise la spécialisation pour des niches trophiques peu utilisées (Warren et al. 2015). La faible présence de prédateurs généralement observée dans les systèmes insulaires induit une faible pression de sélection sur les espèces, ce qui leur permet d’adopter des comportements et phénotypes qui seraient autrement contre-sélectionnés en présence d’une plus grande densité et diversité de prédateurs (Runemark et al. 2014).
En général, les espèces insulaires montrent une variation génétique faible comparativement aux espèces continentales qui leur sont apparentées, en raison de leur isolement et de la plus forte fréquence de goulots d’étranglement (Frankham 1997). La méta-analyse de Uller & Leimu (2011) a révélé que cette baisse de diversité est particulièrement importante chez les insectes et les mammifères introduits dans un nouvel environnement. On retrouve parfois quelques exceptions où la diversité génétique de certaines populations introduites ne diffère pas significativement de la population source, même en milieu isolé (voir Tableau S7, Annexe).
Ces laboratoires naturels que sont les îles suscitent l’intérêt pour la recherche en biologie évolutive de par leur relative simplicité. Selon la biologie de l’espèce, la distance
7
séparant les îles de la côte peut empêcher tout flux de gènes avec les populations continentales laissant ainsi la dérive, la sélection et selon la période temporelle, la mutation agir sur les populations (Pemberton et al. 1996, Blanquart et al. 2013). Plusieurs introductions d’espèces sur des îles ont été réalisées par l’homme, ce qui permet l’accès à des informations comme la date et le nombre d’individus introduits qui, lorsque mises en lien avec la population source ou l’ancêtre continental, offrent d’incroyables possibilités d’approfondir nos connaissances en biologie évolutive (Uller & Leimu 2011).
Le cerf de Virginie
Biologie
Le cerf de Virginie (Odocoileus virginianus, Zimmermann 1780) est l’un des cervidés les plus répandus d’Amérique Nord, son aire de répartition s’étendant du Québec au Brésil (Seabury et al. 2011). Cette espèce qui a su s’adapter à différents milieux possède une grande valeur économique en raison de la chasse sportive, ce qui explique les nombreuses recherches à son sujet. En 2000, on estimait les revenus de la chasse sportive à 78,2 M$ au Québec, dont 12 M$ en provenance d’Anticosti (Huot & Lebel 2012). D’ailleurs, la chasse a provoqué une forte exploitation des cerfs dans plusieurs régions de l’Amérique du Nord au début du 20e siècle (DeYoung et al. 2003,
Monteith et al. 2013). La chasse sportive influence la structure d’âge et la dynamique des populations (Festa-Bianchet 2003, Milner et al. 2007), ce qui en fait un outil de gestion efficace, lorsque bien contrôlé. La présence de chasseurs diminue le niveau d’activité diurne, dont la quête de nourriture, ce qui affecte négativement la masse des cerfs (Little et al. 2016, Strickland et al. 2017). Dans un contexte de surabondance, où la compétition pour les ressources est déjà forte, une pression de chasse intense pourrait affecter grandement la masse des individus d’une population de cerfs et ultimement leur survie. En ciblant des phénotypes particuliers, la chasse aux trophées peut occasionner des changements dans les fréquences alléliques de la population si la pression de chasse est très intense (Coltman et al. 2003a, Monteith et al. 2013, Heffelfinger 2018).
Le temps de génération, soit le temps nécessaire au remplacement d’une cohorte, est d’approximativement quatre ans chez le cerf de Virginie (Kekkonen &
8
Brommer 2015). Il s’agit d’un mammifère polygame : les mâles peuvent se reproduire avec plusieurs femelles pendant le rut, qui a lieu à la fin de l’automne. Durant cette période, les mâles accordent moins de temps à l’alimentation et occupent la majorité de leur temps à la recherche active de partenaires et à leur défense face à d’autres mâles (Holzenbein & Schwede 1989, Mainguy & Côté 2008). La condition corporelle des mâles après le rut en est affectée, et ce, au moment d’entrer dans la difficile période hivernale. Durant cette saison, les cerfs continentaux se rassemblent généralement en des lieux denses en conifères appelés « ravages » pour retourner à leur aire estivale au printemps. C’est au printemps que les femelles mettent bas à un ou deux petits. La condition corporelle, notamment le pourcentage de gras, des femelles est positivement reliée à la taille de portée puisque le gras est essentiel à la production d’énergie requise pour le développement des fœtus et à la thermorégulation de la mère durant l’hiver (Cothran et al. 1987, Ayotte et al. non publié). De plus, les femelles avec une hétérozygotie plus élevée produisent davantage de jumeaux (Cothran et al. 1987, Leberg et al. 1992).
La plasticité du cerf de Virginie est l’une des explications de sa grande aire de répartition. La qualité des sols, et donc du fourrage, et la date de naissance expliquent davantage la morphologie des cerfs que leur génétique (Gray et al. 2002, Strickland & Demarais 2008, Jones et al. 2010). L’influence de ces composantes environnementales par rapport aux composantes génétiques a été mise en évidence dans des expériences de jardin commun avec le cerf de Virginie (Michel et al. 2016). De manière générale, l’effet de l’environnement sur la taille corporelle est largement démontré, bien que ce trait soit héritable (Ozgul et al. 2009, Morrissey et al. 2012).
Structure génétique des populations
Plusieurs études sur la génétique du cerf de Virginie, notamment aux États-Unis, ont été réalisées dans le passé. La majorité témoigne d’une diversité génétique élevée (Smith et al. 2001), et ce, même en milieu perturbé par les activités anthropiques (diversité des ADN microsatellites : H : 0.67–0.74, DeYoung et al. 2003, Blanchong et al. 2013). Le wapiti nord-américain (Cervus elaphus), qui fut lui aussi fortement exploité, affiche une diversité génétique nettement inférieure à celle du cerf (0.26-0.54, Polziehn et al. 2000). Il en va de même pour l’orignal (Alces alces) (0.22-0.41, Broders et al.
9
1999). La capacité des cerfs à atteindre de fortes densités rapidement (DeYoung 2011) et leur structure sociale peuvent expliquer le maintien de leur grande diversité génétique (Blanchong et al. 2013). Les femelles ont généralement un comportement philopatrique et tendent à demeurer à l’intérieur d’un rayon de 50 km de leur lieu de naissance une fois l’âge adulte atteint (Purdue et al. 2000). Un comportement similaire explique en grande partie la structuration génétique chez le cerf rouge (Nussey et al. 2005) et aussi chez d’autres ongulés (Coltman et al. 2003b). À l’opposé, les mâles ont tendance à se disperser, surtout en période de rut, sur des distances aussi grandes que 200 km (Purdue et al. 2000). Ils maintiennent ainsi une variation génétique à l’intérieur des sous-populations (Purdue et al. 2000). Les femelles vont effectuer leurs plus grands mouvements presque exclusivement lors de la mise bas, cherchant à s’éloigner de leurs congénères pour éviter la compétition (Lutz et al. 2015). Seules les grandes rivières semblent constituer une barrière considérable pour l’espèce (Long et al. 2010, Locher et al. 2015). Au Québec, le fleuve Saint-Laurent s’est révélé être la principale barrière aux échanges entre les populations (Albert 2007). Ces déplacements entraînent une faible structure génétique même à grande échelle (> 1000 km), comme observé entre des sous-populations de cerfs de Virginie et de cerfs mulet (Odocoileus hemionus hemionus, Rafinesque 1817) de l’Ouest canadien avec des FST globaux de 0.006 et 0.008,
respectivement (Cullingham et al. 2011a, 2011b).
En tant que taxon privilégié pour la chasse sportive, les ongulés ont subi de nombreuses translocations. Même en présence d’effet fondateur, les cerfs de Virginie peuvent avoir une croissance démographique élevée lorsque les conditions environnementales sont adéquates. Ces fortes croissances pourraient suffire à maintenir une bonne diversité génétique malgré la perte d’allèles causée par un fort effet fondateur. C’est ce qu’a illustré une étude récente portant sur la corrélation entre la valeur sélective et l’hétérozygotie chez une population de cerfs de Virginie fondée par quatre individus (Brommer et al. 2015). Ces quatre individus ont été introduits au sud de la Finlande en 1934 et, de nos jours, les recensements témoignent de >50 000 individus et une hétérozygotie moyenne de l’ADN microsatellite de 0.68 malgré une diminution de la richesse allélique (Kekkonen et al. 2012, Brommer et al. 2015). Malgré leur isolement, ces cerfs ne démontrent pas de différence morphologique par rapport à leur population source américaine (Kekkonen et al. 2016). Inversement, les cerfs des Keys (Odocoileus virginianus clavium), appartenant à une sous-espèce du cerf de Virginie isolée sur
10
l’archipel des Keys au sud de la Floride, se sont fortement différenciés morphologiquement et génétiquement des cerfs continentaux (FST = 0.202) à la suite de
leur isolement et de leur forte exploitation dans les années 1950 (Villanova et al. 2017). En comparaison, les deux lignées évolutives d’Odocoileus hemionus, soit le cerf mulet et le cerf à queue noire, ont une différenciation génétique plus modérée (FST = 0.123,
Powell et al. 2016).
Le cas à l’étude : les cerfs introduits sur l’île d’Anticosti
C’est pour la chasse sportive que le cerf de Virginie a été introduit dans la partie ouest de l’île d’Anticosti (Québec, Canada) en 1896 et 1897 avec approximativement 220 individus (Newsome 1937). Ces derniers provenaient de la rive sud du fleuve Saint-Laurent, principalement de la région de Montmagny-L’Islet (Martin-Zédé 1938, McCormick 1982, Luc Jobin communication personnelle). À cette époque, l’île d’Anticosti avait des ours noirs (Ursus americanus) comme prédateurs des faons particulièrement, ce qui a néanmoins permis l’expansion rapide du cerf de Virginie jusqu’à la disparition des ours en moins de 70 ans (Côté 2005). Dès les années 1930, les effets négatifs sur la végétation causés par le broutement intensif des cerfs signalent une grande population de cerfs (Marie-Victorin & Rolland-Germain 1969, Côté et al. 2008). Puis, vers la fin des années 1980, à l’aide de techniques d’inventaire modernes, on a estimé leur nombre à 120 000 cerfs (Potvin et al. 1991) et, plus récemment en 2006, à >160 000 (Rochette & Gingras 2007). La grande pression de broutement imposée à la végétation a même affecté la structure forestière de l’île. En effet, la diminution des sapinières, ressources hivernales importantes pour le cerf (Lefort et al. 2007, Sauvé & Côté 2007), a entraîné l’augmentation des peuplements d’épinette blanche, qui est une espèce de moins bonne qualité nutritive (Potvin et al. 2003).
La fluctuation de taille de population n’a pas été le seul changement observé sur cette population. Des changements morphologiques ont en effet été mentionnés dans la littérature, dont la petite taille des cerfs de l’île d’Anticosti (Lesage et al. 2001). Cette même étude rapportait que les faons de l’île d’Anticosti avaient une plus grande longueur des pattes, proportionnellement au corps, ainsi que des réserves de gras à l’automne supérieures à celles des faons continentaux. Ces résultats laissaient suggérer une forte sélection sur ces traits pour la première année de vie par rapport aux
11
populations continentales soumises à des pressions de sélection moins intenses (conditions hivernales et densité de population). Les rudes conditions hivernales et la faible abondance de nourriture de bonne qualité sont les causes principales de mortalité, qui peut atteindre 38 % lors des hivers les plus rigoureux (Taillon et al. 2006). La masse corporelle des individus est déterminante pour leur survie pendant l’hiver, car l’apport alimentaire diminue alors que les besoins énergétiques requis pour la thermorégulation et les déplacements augmentent (Taillon et al. 2006). Malgré l’utilisation de ressources de faible qualité nutritive, la population s’est maintenue à forte densité grâce à la plasticité digestive des cerfs (Bonin et al. 2016), à l’adoption de diverses stratégies de sélection d’habitat (Courbin et al. 2017) et de stratégies biodémographiques. Par exemple, les cerfs de l’île d’Anticosti tendent à reporter leur reproduction d’un an afin d’investir leurs réserves énergétiques dans leur condition corporelle et leur survie (Simard et al. 2014 b, Ayotte et al. non publié).
Ainsi, considérant l’historique d’introduction et les nombreux changements phénotypiques, les cerfs de l’île d’Anticosti constituent un sujet d’étude idéal pour approfondir les connaissances théoriques en biologie évolutive. Grâce à la simplicité des systèmes insulaires, cette étude offre la possibilité d’approfondir de nouvelles connaissances sur l’évolution génomique des mammifères en contexte d’effet fondateur et de surabondance.
Objectifs et hypothèses
L’objectif principal était de quantifier la potentielle différenciation génétique des cerfs de l’île à la suite d’un effet fondateur, dans un cadre de gestion de la chasse sportive. Trois objectifs spécifiques ont été définis pour y parvenir : 1) comparer la diversité génétique entre la population de l’île d’Anticosti et des populations continentales, dont la population source, 2) vérifier si la diversité génétique à l’intérieur de l’île reflète l’historique de colonisation depuis l’ouest de l’île vers l’est et 3) évaluer la différentiation adaptative par une approche génotype-phénotype associée aux traits morphologiques divergents entre les cerfs de l’île d’Anticosti et ceux du continent.
12
Approche méthodologique
La génétique des populations étudie l’histoire évolutive et la microévolution des populations entraînées par les différents mécanismes évolutifs. Avant l’avancée technologique actuelle, seules quelques dizaines de marqueurs génétiques neutres (allozymes, AFLP, microsatellites) étaient utilisés pour représenter une petite portion du génome (Davey et al. 2011, Narum et al. 2013). De nos jours, il est possible d’augmenter la couverture du génome en analysant simultanément des milliers de loci avec les polymorphismes d’un seul nucléotide (SNPs) grâce aux technologies de séquençage de nouvelle génération (Luikart et al. 2003, Allendorf et al. 2010). Ces technologies ont permis la création de plusieurs méthodes, dont le génotypage par séquençage (GBS), qui repose sur la fragmentation du génome grâce à des enzymes de restriction pour en réduire la complexité (Davey et al. 2011, Elshire et al. 2011). La diminution des coûts reliés au séquençage ouvre la voie vers des études génomiques portant sur des organismes non modèles, comme le cerf de Virginie (Helyar et al. 2011, McMahon et al. 2014).
Pour investiguer l’ampleur d’une possible divergence adaptative d’une population à un nouvel environnement, nous avons combiné une approche génomique avec des données morphologiques, ce que peu d’études ont réalisé à ce jour chez les ongulés. Comme les SNPs ne sont pas uniquement situés dans des régions neutres, mais sont également dans des gènes (ou liés à ceux-ci), ils possèdent à cet égard un autre avantage comparé aux autres types de marqueurs génétiques (Morin et al. 2004). L’expression d’un trait phénotypique est rarement régulée par un seul locus, mais bien par l’expression de plusieurs loci à faibles effets (Pritchard et al. 2010, Yang et al. 2010, Messer & Petrov 2013). En générant des milliers de marqueurs, les techniques de séquençage de nouvelle génération permettent d’identifier davantage de loci sous sélection, ce qui améliore notre compréhension de l’adaptation (Narum et al. 2013, Savolainen et al. 2013). Pour le troisième objectif de cette étude, nous avons tenu compte de la base polygénique des traits phénotypiques en utilisant des associations génotype-phénotype qui recherchent des covariations parmi les loci (Le Corre & Kremer 2012, Brieuc et al. 2015, Gagnaire & Gaggiotti 2016, Laporte et al. 2016). Notre approche a été bonifiée par la combinaison des méthodes standard de balayage génomique pour trouver des loci potentiellement sous sélection dont certains ont été
13
précédemment associés aux traits phénotypiques divergents (de Villemereuil et al. 2014, Rellstab et al. 2015). Les loci communs entre ces deux types d’approches (balayage génomique et associations génotype-phénotype) appuient l’adaptation locale de ces traits sur l’île d’Anticosti.
Des données morphologiques sur des cerfs de l’île d’Anticosti récoltés à la chasse sont amassées depuis 2002. Ce suivi a permis de recueillir des mesures morphologiques sur les 437 cerfs utilisés dans mon étude. L’âge des individus était déterminé avec les anneaux de croissance des incisives récoltées (Hamlin et al. 2000). Seuls les individus adultes (>1,5 an) ont été conservés pour cette étude, car les juvéniles peuvent biaiser la structure génétique de la population (Comer et al. 2005). Les traits connus comme étant divergents entre la population de l’île et les populations continentales ont été utilisés pour évaluer les bases génétiques des adaptations phénotypiques des cerfs de l’île. Nous avons ainsi considéré la masse corporelle éviscérée, la longueur de la patte arrière, ainsi que l’épaisseur de gras sous-cutané du fessier et la masse du muscle péronier qui ont respectivement été utilisées pour représenter les réserves adipeuses et protéiques (Crête et al. 1993, Simard et al. 2014a). Pour les mâles, nous avons également utilisé la distance entre les extrémités des deux bois pour représenter la variation de la taille du panache.
14
Chapitre 1
Weak founder effect and evidence for adaptive divergence in a recently
introduced insular population of white-tailed deer (Odocoileus virginianus)
Jérémie Fuller1, 2, 4, Anne-Laure Ferchaud4, Martin Laporte4, Jérémy Le luyer4, Theodore
B. Davis3, Steeve D. Côté1, 2, and Louis Bernatchez1, 4
1 Département de biologie, Université Laval, Québec, Québec, Canada G1V0A6 2 Natural Sciences and Engineering Research Council of Canada (NSERC) Research
Chair in Integrated Management of the Resources of Anticosti Island and Centre d’études Nordiques (CEN)
3 New England Biolabs Inc, USA
15
Résumé
Les îles sont généralement colonisées par quelques individus, ce qui pourrait entraîner un effet fondateur et provoquer une divergence rapide due à une forte dérive génétique. De plus, des conditions insulaires particulières peuvent induire de nouvelles pressions sélectives sur les populations. Dans cet article, nous avons étudié 1) l'étendue de la différenciation génétique entre une population de cerfs de Virginie (Odocoileus virginianus) introduite sur une île à la limite nordique de la répartition de l’espèce et sa population source du continent, 2) la structure génétique de la population à l’intérieur de l'île et 3) les bases génétiques de la différenciation morphologique entre les individus de l'île et du continent. Nous avons mené notre étude sur l'île d'Anticosti (Québec, Canada) où près de 220 individus ont été introduits il y a environ 120 ans, pour atteindre une taille de population de plus de 160,000 individus. Nous avons utilisé une approche de génotypage par séquençage (GBS) pour générer 8,518 SNP filtrés de haute qualité pour comparer les modèles de diversité génétique et de différenciation entre les populations continentales et celle de l'île d'Anticosti. Les analyses de regroupement appuient la présence d’une seule population dans l'île d'Anticosti et aucun signe d'isolement par la distance. Nos résultats ont révélé une faible différenciation génétique entre la population de l'île d'Anticosti et sa population source (FST moyen = 0,005), bien que suffisante pour
réussir les tests d'assignation de population. La grande diversité génétique de la population introduite suggère un faible effet fondateur suivi d’une croissance démographique rapide. Nous avons également utilisé une approche polygénique pour évaluer les bases génétiques des traits phénotypiques divergents entre les populations insulaires et continentales. Nous avons trouvé des loci liés à la réponse immunitaire, à la fonction musculaire et au métabolisme des lipides, ce qui suggère qu'ils pourraient être impliqués dans l'adaptation locale sur l'île d'Anticosti. Nous discutons de ces résultats dans un contexte de gestion de la chasse sportive.
16
Abstract
Islands are generally colonized by few individuals which could lead to a founder effect and cause rapid divergence by strong genetic drift. In addition, particular insular conditions can induce new selective pressures on populations. Here, we investigated 1) the extent of genetic differentiation between a white-tailed deer (Odocoileus virginianus) population introduced on an island at the northern limit of the species distribution and its source population from the mainland, 2) the genetic structure of the population within the island and 3) the genetic bases of the morphological differentiation between island and mainland individuals. We conducted our study on Anticosti Island (Québec, Canada) where about 220 individuals were introduced approximately 120 years ago, resulting in a population size over 160,000 individuals. We used a genotyping-by-sequencing (GBS) approach to generate 8,518 filtered high-quality SNPs used to compare patterns of genetic diversity and differentiation between the continental and Anticosti Island populations. Clustering analyses resolved a single population within Anticosti Island and no sign of isolation by distance. Our results revealed a weak genetic differentiation between the Anticosti Island population and its source population (mean FST = 0.005),
albeit sufficient for successful population assignment tests. The higher genetic diversity of the introduced population suggests a weak founder effect following rapid population growth. We further used a polygenic approach to assess the genetic bases of the divergent phenotypical traits between insular and continental populations. We found outlier loci related to immune response, muscular function and lipid metabolism, which suggested that these could be involved in local adaptation on Anticosti Island. We discuss these results in a hunting management context.
17
Introduction
The establishment of a new population from a mainland source into an isolated environment (e.g. islands) often leads to pronounced genetic divergence due to both restricted gene flow and genetic drift. As such, islands are remarkable natural laboratories to study the action of evolutionary mechanisms and their consequences for management and conservation (Pemberton et al. 1996, Warren et al. 2015). A special case of genetic drift, the founder effect, is the strongest process leading to drastic and rapid genetic differentiations between populations (Barton 1996, Hartl & Clark 1997, Abdelkrim et al. 2005, Kolbe et al. 2012). First proposed by Mayr (1954), the concept of founder effect refers to the random sampling of alleles brought by individuals from a source population to establish a new population (Barton & Charlesworth 1984, Hartl & Clark 1997). This random selection of introduced individuals leads to a genetic diversity that is not representative of the entire source population and which usually correlates with the number of introduced individuals and/or the number of introduction events (Allendorf 1986, Clegg et al. 2002, Blanchong et al. 2013). Because new populations are often founded by few individuals, especially on islands (Warren et al. 2015), they generally have low effective population size (Ne) which enhance genetic drift and consequently the loss of genetic diversity (Nei et al. 1975, Frankham 1995, Charlesworth 2003). Therefore, insular populations tend to have lower genetic diversity than mainland populations (Frankham 1997), especially in mammals (Uller & Leimu 2011). The lower genetic diversity of a population may reduce its capacity to adapt and, therefore, its chance to persist in time (Reed & Frankham 2003, Wood et al. 2016). The rapid growth of a founder population can, however, decrease the rate of genetic diversity loss (Nei et al. 1975, Allendorf 1986, Blanchong et al. 2013).
Organisms introduced on islands face novel climatic conditions (Weigelt et al. 2013) and biological communities (Warren et al. 2015). In founder populations, initial genetic adaptations can occur rapidly from standing genetic variation (Barrett & Schluter 2008, Messer & Petrov 2013, Crisci et al. 2016). Strong genetic drift in founder populations may, however, reduce standing genetic variation (Luikart et al. 1998, Dlugosch and Parker 2008) and counteract selection, thus hampering adaptive divergence (Barton 1996, Agashe et al. 2011, Swaegers et al. 2013). Rare alleles have more chance to be lost in such events (Nei et al. 1975, Luikart et al. 1998, Clegg et al.
18
2002), thus depriving founder populations of potential beneficial alleles in novel environments.
Pronounced phenotypic changes, often in body size, are commonly occurring in insular populations (Meiri et al. 2008). VanValen (1973) first proposed the island rule which states that species with small body size tend to get larger, while species with large body size tend to get smaller. This general pattern is explained by the novel selective pressures (i.e.: amount of resources available, intra- and interspecific interactions, etc.) faced by colonizing species which drive body size toward a new optimal state (Lomolino 2005, Meiri et al. 2008, McClain et al. 2013, Runemark et al. 2014). The limited resources available on islands increases competition which tends to benefit the smallest individuals of species with large body size because of their lower energy requirements (Lomolino 2005). At the opposite, small species will be advantaged toward a larger body size due to the frequent absence of large predators and competitors in islands (Lomolino 2005, Runemark et al. 2014). These phenotypic changes can occur over a few generations, including in mammals (Millien 2006). Such rapid phenotypic changes may result from either divergent selection (Grant 2001, Price et al. 2003) or genetic drift (Barton & Charlesworth 1984, Kolbe et al. 2012, Spurgin et al. 2014), and both of these evolutionary forces are more likely to occur in insular ecosystems than on the continent (Dlugosch & Parker 2008, Funk et al. 2016, Prentice et al. 2017). Phenotypic plasticity could also explain such rapid changes (Lerp et al. 2014). Despite numerous studies conducted on island systems (Grant 2001, Clegg et al. 2002, Warren et al. 2015), the link between genome-wide diversity and potential adaptive phenotypic changes considering the polygenic basis of traits has rarely been investigated.
The white-tailed deer (Odocoileus virginianus Zimmermann 1780) is one of the most widespread large mammals in North America and has been introduced in many areas because of its popularity for sport hunting (Little et al. 2016). Approximately 220 deer were introduced for this purpose on Anticosti Island (Québec, Canada) between 1896 and 1897 (Martin-Zédé 1938, McCormick 1982, L. Jobin, personnal communication). The introduced deer were taken from the region of Montmagny on the southern shore of the St. Lawrence River, Québec, Canada (Newsome 1937). Because of the low abundance of predators on the island, the population quickly increased in number. Intense browsing soon caused major impacts on the vegetation which were reported as early as 1934 (Marie-Victorin & Rolland-Germain 1969, Potvin et al. 2003, Côté et al. 2008). Phenotypic changes such as a reduction of body size, increased fat
19
storage for fawns, increased leg length and increased antler spread were also documented (Lesage et al. 2001, Simard et al. 2014a). As reported in other studies, such rapid phenotypic divergence suggests a presence of genetic differentiation between Anticosti Island and mainland deer (Koskinen et al. 2002, Funk et al. 2016, Prentice et al. 2017) or could be attributed to phenotypic plasticity (Lerp et al. 2014). Evaluating the relative role of genetic drift and adaptive genetic divergence as drivers of phenotypic divergence in Anticosti deer requires documenting patterns of both neutral and potentially adaptive genetic variations between insular and mainland deer.
Our main goal was to evaluate the patterns of genetic differentiation between white-tailed deer from Anticosti Island and their source population using genotyping-by-sequencing (GBS). We also compared divergence between the Anticosti Island deer population and a non-related continental population (Outaouais) found hundreds of kilometers away from the Anticosti source population as a reference for likely higher genetic difference in white-tailed deer (Albert 2007). More specifically, we aimed to: 1) test the hypothesis that the founder effect led to a pronounced loss of genetic diversity of white tailed deer on Anticosti Island, as frequently reported in other studies; 2) test the hypothesis that patterns of genetic diversity on Anticosti Island reflect the colonizing process which began at the site of introduction located at the western end of the Island; and 3) test the hypothesis of a genotype-phenotype association for morphological traits that diverged between Anticosti and mainland deer, which would support an adaptive basis of differentiation. We then interpret and discuss our results in the context of hunting management.
Material and Methods
Study area
Deer were sampled from three areas, including Montmagny (MON; 46°N 70°W), the continental source population, Outaouais (OUT; 46°N 76°W), a geographically distant outgroup (Fig. 1), as well as on Anticosti Island (ANT; 49ºN 62ºW; 7,943 km2).
Anticosti Island was divided into three management sectors corresponding to the three main exploited zones: i) Western Anticosti (W-AN), ii) Central Anticosti (C-AN), and iii) Eastern Anticosti (E-AN).
20
Anticosti Island is located in the Gulf of St. Lawrence, Québec (Canada) at the northern limit of white-tailed deer distribution. Climate is harsh with long, cold and snowy winters (mean of 406 cm of precipitation as snow/year; Environment Canada). High deer density on the island (>20 deer/km2 vs <3 deer/km2 in Montmagny and Outaouais; Huot
& Lebel 2012) has been maintained over the past nine decades, causing intense long-term browsing which resulted in the decline of most deciduous browse species such as Sorbus americana Marsh, Amelanchier sp., Diervilla lonicera P. Mill., and Viburnum spp. (Côté 2005, Tremblay et al. 2005). The forest is mainly composed of balsam fir (Abies balsamea), white spruce (Picea glauca) and black spruce (Picea mariana), and is located in the balsam fir-white birch bioclimatic domain (Potvin et al. 2003, Huot & Lebel 2012). Montmagny is located in the balsam fir-yellow birch bioclimatic domain on the southern shore of the St. Lawrence River where winter can also be harsh (mean of 243 cm of snow/year; Environment Canada) and Outaouais is located in southwestern Québec on the northern shore of the St. Lawrence River which is an effective barrier for white-tailed deer (Long et al. 2010). Outaouais is mostly characterized by the sugar maple-yellow birch bioclimatic domain where milder winter occurs (mean of 200 cm of snow/year; Environment Canada)
Figure 1. The three study areas (Québec, Canada). A) Anticosti Island divided into three regions: western (W-AN), central (C-AN), and eastern (E-AN). B) Montmagny-L’Islet (MON) and Outaouais (OUT).
21
Sampling and phenotypic measurements
A total of 571 individuals were sampled through sport hunting in the fall (Anticosti: 445 individuals (W-AN: 144, C-AN: 145, E-AN: 148), Montmagny: 54 and Outaouais: 72). Ear or muscle tissues were collected and preserved in 95% ethanol or kept frozen at -20ºC for genomic analyses. We used a balanced sex ratio for Anticosti samples (W-AN [71 Females: 73 Males], C-(W-AN [73F:72M], E-(W-AN [74F:74M]) collected during two periods (2003 to 2005 and 2012 to 2014). Samples from Montmagny were mostly collected on males in 2013 [15F:39M], whereas samples from Outaouais were collected in 2006 and 2015 with a balanced sex ratio [39F:33M]. Only adult individuals (>1.5 years) were used, their age was determined using cementum layers of teeth (Hamlin et al. 2000). Morphometric measures were only recorded on Anticosti Island. We recorded 1) eviscerated body mass, 2) total body length, 3) chest girth, 4) length of the left hind leg and the mass of its peroneus muscle which is a reliable estimator of protein mass (Crête et al. 1993, Simard et al. 2014a), 5) rump fat thickness which is used as an index of body fat reserves (Cook et al. 2010), and 6) antler spread for males. More details on morphometric measurements are in Simard et al. (2014a).
Genomic analyses
DNA extraction and sequencing
We extracted genomic DNA from ear or muscle tissues with a phenol-chloroform-isoamyl protocol (Sambrook et al. 1989). We checked sample concentration and quality by using a 1% agarose gel and a NanoDrop 2000 spectrophotometer (Thermo Scientific) and normalized the genomic DNA to obtain 20 ng/ul in 10 ul (200 ng) by the Biotium AccuClear protocol performed on a SPARK 10M (TECAN) 96-well plate. Genotyping-by-sequencing (GBS) libraries were prepared by the Institut de Biologie Intégrative et des Systèmes (IBIS) sequencing platform (Laval University) and sequenced on an Ion Torrent Proton with a two-enzyme GBS protocol (Mascher et al. 2013), using restriction enzymes NsiI and MspI. Individuals were barcoded with a unique sequence of six nucleotides. Each sample was sequenced a second time to reach a sufficient coverage per individual.